ERMDA:Predicting miRNA-disease associations using an ensemble learning framework with resampling method
![]()
目录
已证实的人类miRNA与疾病的关联 (The verified human miRNA-disease associations)
疾病-疾病相似性(Disease-disease similarity)
疾病语义相似性(Disease semantic similarity)
疾病的GIP核相似度 (GIP kernel similarity for diseases)

疾病整合相似度(Integrated similarity for diseases)
miRNA-疾病特征表示与选择(MiRNA-disease feature representation and selection)
实验设置和评价指标(Experimental settings and evaluation metrics)
基于不同个体学习器的ERMDA框架性能分析(Performance analysis of ERMDA framework using different individual learners)
ERMDA框架中特征选择方法的有效性(Effectiveness of the feature selection method in the proposed ERMDA framework)
基于软投票的集成学习方法的有效性(Effectiveness of the soft voting-based ensemble learning method)
与最新方法的比较(Comparisons with the state-of-the-art methods)
摘要(Abstract)
研究动机:越来越多的证据表明,microRNA(miRNA)在各种复杂疾病的发病和进展中起着至关重要的作用。 推断与疾病相关的miRNAs对于探索人类疾病的病因、诊断和治疗具有重要意义。 由于生物学实验费时费力,开发有效的计算方法来识别miRNAs与疾病之间的联系已成为必不可少的。 结果:我们提出了一个基于重采样方法的集成学习框架用于miRNA-疾病关联(ERMDA)预测,以发现潜在的疾病miRNAs。 首先,提出了重采样策略,用于建立多个不同的平衡训练子集,以解决数据库中样本不平衡的挑战。 然后ERMDA通过整合miRNA与miRNA相似性、疾病与疾病相似性以及实验验证的miRNA与疾病关联信息,提取miRNA和疾病特征表示。 然后,采用特征选择方法减少冗余信息,增加子集之间的多样性。 最后,ERMDA在每个子集上构造一个单独的学习者以产生原始结果,并引入软投票方法根据单个学习者的预测结果做出最终决策。 一系列的实验结果表明ERMDA在平衡测试集和非平衡测试集上都优于其他现有的方法。 此外,对三种人类疾病的病例研究进一步证实了ERMDA对识别潜在疾病相关miRNAs的预测能力。 总之,这些实验结果表明,我们的方法可以作为一个有效和可靠的工具来探索miRNAs在复杂疾病中的调节作用。
关键词:miRNA与疾病关联,集成学习,重采样,特征选择
引言(Introduction)
大约22个核苷酸的微小 RNA (miRNA)是一类短的内源性非编码 RNA,通过与靶 mRNA 的3-非翻译区结合在转录后水平的基因表达调控过程中起重要作用[1-3]。最近,越来越多的研究发现,miRNA 存在于各种生物体中,并参与不同的生物活动[4]。例如,秀丽隐桿线虫中的 lin-4和 let-7是首先发现的两种 miRNA [5] ,let-7在细胞周期停滞期间诱导靶 mRNA 的翻译上调[6]。林等人 [7]观察到miR-1179的过表达可明显抑制胰腺癌细胞的迁移和侵袭。 此外,miRNAs的异常和失调可能导致多种复杂疾病,包括乳腺癌[8]、心血管疾病[9]、肺癌[10]等[11]。 因此,准确识别潜在的疾病相关miRNAs将有助于理解疾病的病理机制,促进人类复杂疾病的诊断和治疗。
在过去的十年中,由于生物实验测定的费时和高成本,通过计算方法预测 miRNA 与疾病的关联已经引起了广泛的关注。现有的计算方法大致可分为两类:基于网络的方法和基于机器学习的方法。 以前的方法主要是构建miRNA-疾病异构网络,利用相似性度量来预测疾病相关的miRNAs,其假设功能相似的miRNAs可能与表型相似的疾病相关,反之亦然[12]。 例如,陈等人[13]提出了RWRMDA方法,在miRNA函数相似度网络上实现随机游走,以推断miRNA与疾病的关联。 通过将高斯互作谱(GIP)核集成到miRNA和疾病相似性的计算中,Chen等人[14]开发了WBSMDA方法,该方法可以应用于没有任何已知相关miRNAs的疾病。 PBMDA[15]在异构网络上采用深度优先搜索算法来预测miRNA与疾病的关联。 最近,于等人[16]提出了基于矩阵补全和标签传播方法的MCLPMDA方法。陈等人[17]提出了将邻域约束与矩阵补全结合起来研究疾病相关miRNAs的NCMCMDA方法,取得了较大的改进。 虽然在识别miRNAs与疾病之间的关联方面取得了很大的效果,但这些方法的性能通常受到所构建网络质量的影响[18],而且大多数这类方法都是无监督的,没有使用任何标记信息,这也在很大程度上限制了它们的预测能力。
迄今为止,随着实验证实的miRNA与疾病关联的积累,基于机器学习的研究变得更加广泛[19]。 这些方法使用已知的关联来训练机器学习模型,并预测潜在的与疾病相关的miRNAs。 然而,数据不平衡问题是基于机器学习方法的主要挑战。 具体地说,与未经证实的miRNA与疾病的关联(称为未标记的样本)相比,只有一小部分已知的关联(称为阳性样本)得到了验证。 针对这一问题,许多方法将未标记的样本视为阴性样本,然后随机选择与阳性样本数量相同的阴性样本,如EGBMMDA[20]、MLMDA[21]、ABMDA[22]等。 ABMDA在未标记样本上引入了Kmeans聚类方法,并开发了一个自适应Boosting模型来预测miRNA与疾病的关联。 然而,上述方法在构建负样本集时存在局限性,因为从大规模数据中选择少量样本可能会导致预测偏差,并且在选择的未标记样本中可能存在真正的miRNA与疾病关联。此外,对于这些基于机器学习的方法,影响预测性能的其他两个关键因素是特征表示和预测模型。大多数方法都是从异构网络中提取特征,并利用这些特征直接训练分类器。 然而,特征中可能存在一些噪声和冗余信息。 为了在降低计算代价的同时提高预测性能,Chen等人[23]提出了一种基于滤波器的特征选择方法来降低特征空间的维数。 彭等人[24]提出了一个基于卷积神经网络的框架MDA-CNN,该框架采用自动编码器来捕获基本特征组合。 IRFMDA[25]根据特征在随机森林(RF)中的重要度得分对特征进行降序排序,选取前100个特征进行分类。 在预测模型方面,以往的一些方法主要采用基于浅层学习的模型作为分类器。 例如,徐等人[26]从miRNA靶点失调网络中提取特征,并使用支持向量机(SVM)模型来识别前列腺癌的疾病相关miRNA。 陈等人[27]发展了基于正则化最小二乘法的预测miRNA与疾病关联的方法。 然而,由于基于浅层学习的模型分类能力有限,它们不能取得令人满意的性能。 在过去的几年里,深度学习发展迅速,并被应用于生物信息学的各种应用[28,29]。 李等人[30]提出了一种新的用于miRNA与疾病关联预测的图自动编码器模型,该模型使用基于图神经网络的编码器生成miRNA和疾病特征,然后使用双线性解码器重建miRNA与疾病之间的联系。 Ji等人[31]提出了一种基于深度自动编码器的模型AEMDA来检索miRNA与疾病之间的潜在联系,AEMDA比几种最先进的方法表现得更好。 然而,基于深度学习的模型的预测性能依赖于大规模数据和繁琐的超参数调整过程[32],但目前还没有足够的实验证实的miRNAs与疾病之间的关联。 最近,Chen等人[33]提出了一种基于决策树的集成方法EDTMDA来推断潜在的与疾病相关的miRNAs。 为了预测circRNA与疾病的联系,Zeng等人[34]提出了一种基于深度森林的集成学习方法,该方法克服了深度学习模型的缺点,取得了比其他相关方法更好的性能。 此外,在我们之前的工作中,我们提出了一个具有异构特征组合的堆叠集成学习框架用于ncRNA-protein相互作用预测[35],它比基于深度学习的方法具有显著的优势。 总之,开发基于集成学习的模型将是发现潜在的miRNA与疾病关联的有效可行的途径。
在本文中,我们提出了一个使用重采样方法的集成学习框架,命名为基于重采样的集成学习框架预测miRNA与疾病的关联(ERMDA)。首先,为了解决样本不平衡的问题,减少单一小样本抽样带来的偏差,提出了建立多个不同均衡训练子集的重采样方法。 其次,结合miRNA-miRNA相似性、疾病-疾病相似性以及miRNAs与疾病之间的关联信息,构建了miRNA-疾病对的特征表示。 第三,采用特征选择策略减少冗余信息,增加子集之间的多样性。 最后,我们在每个子集上建立一个个体学习者来产生原始结果,并引入软投票方法根据个体学习者的预测结果做出最终决策。 为了评估我们提出的方法的性能,在广泛使用的HMDD数据库[36]上进行了一系列五折交叉验证实验。 结果表明,ERMDA的接收机工作特性曲线下面积(AUROC)和查准率-查全率曲线下面积(AUPR)分别达到0.9561±0.0013和0.9542±0.0020,在平衡测试集上优于其他现有的测试方法。 另外,在不平衡测试集上对ERMDA进行了评估,实验结果表明,该方法能够有效地解决样本不平衡问题。 此外,对三种重要的人类疾病进行了案例研究,预测的前30个miRNAs中的大多数都得到了相关数据库[37]和生物学文献的证实,这进一步表明ERMDA在发现潜在的miRNA-疾病关联方面具有很好的能力,可以为指导艰苦的临床生物学实验做出贡献。
材料和方法(Materials and methods)
已证实的人类miRNA与疾病的关联 (The verified human miRNA-disease associations)
在这项工作中,我们从广泛使用的HMDD数据库中获得了实验验证的miRNA与疾病关联数据,该数据库可以在http://www.cuilab.cn/hmdd获得,共包含5430对miRNA-疾病对,包含495个miRNA,383种疾病。 设![]() 为miRNAs数目,
为miRNAs数目,![]() 为疾病数目,构造
为疾病数目,构造![]() 邻接矩阵来表示miRNA-疾病关联信息。 给定一对miRNA
邻接矩阵来表示miRNA-疾病关联信息。 给定一对miRNA ![]() 和疾病
和疾病![]() ,如果它们在HMDD数据库中被确认相关,则
,如果它们在HMDD数据库中被确认相关,则![]()
疾病-疾病相似性(Disease-disease similarity)
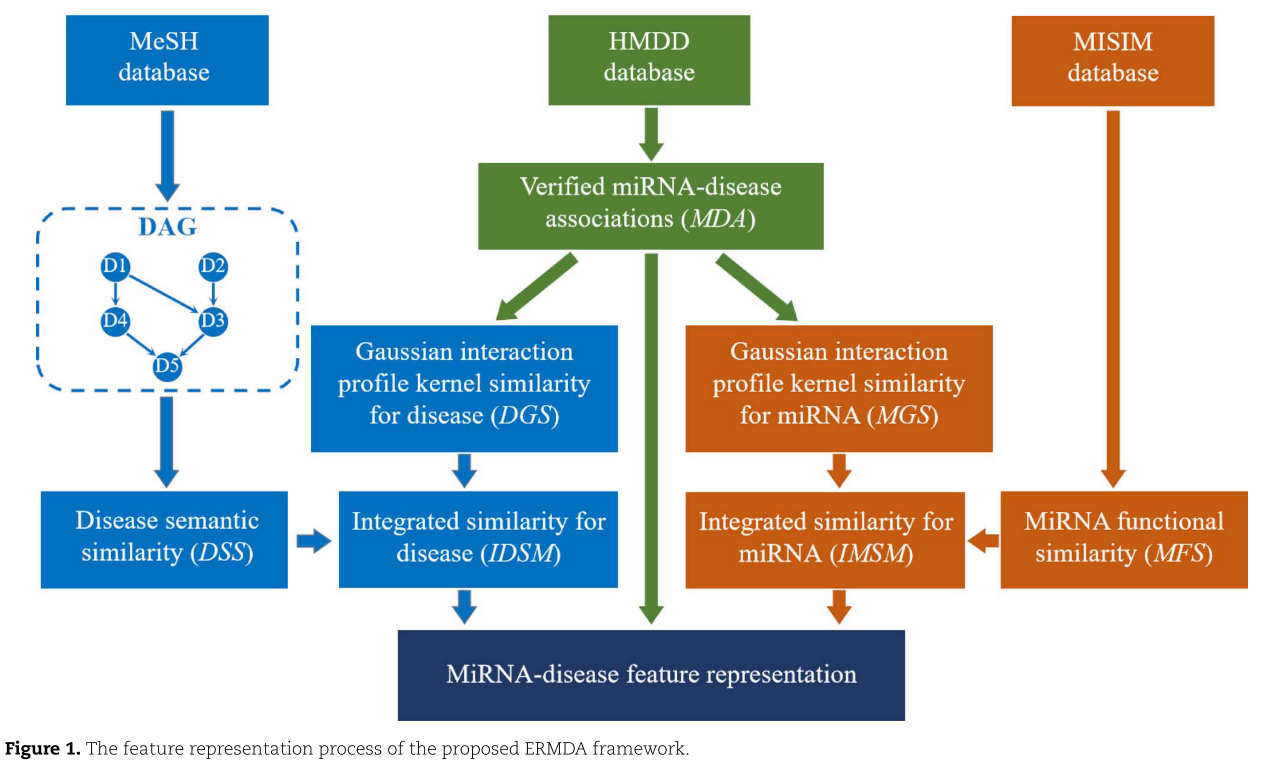
为了更全面地表示疾病之间的关系,引入了疾病语义相似度和疾病的GIP核相似度,如图1左侧所示。
疾病语义相似性(Disease semantic similarity)
受以往研究[30,38]的启发,基于从美国国家医学图书馆下载的医学主题词数据库( http://www.ncbi.nlm.nih.gov ) ,可以推断出疾病的语义相似性。在这里,我们通过Wang的测量方法计算了疾病之间的语义相似度[39]。 每个疾病都可以用一个有向无环图(DAG)来表示,该图包含了所有与疾病相关的注释项。 对于疾病![]() ,我们定义了
,我们定义了![]() ,其中
,其中![]() 表示由
表示由![]() 本身和它的祖先节点组成的节点集,
本身和它的祖先节点组成的节点集,![]() 表示包含从父节点到子节点的直接链路的相应边集。 然后,
表示包含从父节点到子节点的直接链路的相应边集。 然后,![]() 中的疾病T对疾病
中的疾病T对疾病![]() 的语义贡献
的语义贡献![]() ,可以计算如下:
,可以计算如下:

![]() 语义贡献衰减因子。
语义贡献衰减因子。
本工作将疾病![]() 对自身的贡献值等于1,并在前人研究的基础上
对自身的贡献值等于1,并在前人研究的基础上![]() 设定为0.5[39]。 因此,疾病
设定为0.5[39]。 因此,疾病![]() 的语义值
的语义值![]() ,可以计算如下:
,可以计算如下:
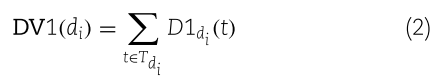
由于在DAG中共享较大部分的疾病对通常更相似,疾病![]() 和
和![]() 之间的疾病语义相似性
之间的疾病语义相似性![]() 定义如下:
定义如下:

显然,对于![]() ,存在于
,存在于![]() 同一层的不同疾病对疾病
同一层的不同疾病对疾病![]() 语义相似度值的贡献是相同的。 然而,假设
语义相似度值的贡献是相同的。 然而,假设![]() ,如果疾病
,如果疾病![]() 只出现在
只出现在![]() 中,
中,![]() 不仅出现在
不仅出现在![]() 中,而且出现在其他疾病的DAGs中,那么疾病
中,而且出现在其他疾病的DAGs中,那么疾病![]() 可能对
可能对![]() 有更高的语义贡献。 因此,我们引入了另一种受Xuan的方法[38]启发的计算疾病语义相似度的方法。 疾病
有更高的语义贡献。 因此,我们引入了另一种受Xuan的方法[38]启发的计算疾病语义相似度的方法。 疾病![]() 对
对![]() 的语义贡献值
的语义贡献值![]() ,计算如下:
,计算如下:

类似地,疾病![]() 和
和![]() 之间的疾病语义相似度
之间的疾病语义相似度![]() 定义如下:
定义如下:
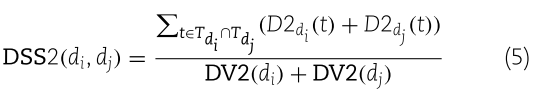
其中![]() 是疾病
是疾病![]() 的语义值。
的语义值。
无论是方程(3)还是(5),疾病语义相似度的计算都是片面的。 因此,与前人[40,41]类似,为了更全面地计算疾病之间的语义相似度,我们将两种疾病语义相似度结合起来,得到了疾病![]() 之间的最终语义相似度
之间的最终语义相似度![]() :
:

疾病的GIP核相似度 (GIP kernel similarity for diseases)
根据前人的研究[22,30],在假设具有相似表型的疾病往往更多地与相似的miRNAs相关的情况下,用疾病的GIP核相似性来描述疾病之间的关系[14]。 设![]() 表示疾病
表示疾病![]() 的二进制互作谱向量,对应于邻接矩阵MDA中的第 i 列,则疾病
的二进制互作谱向量,对应于邻接矩阵MDA中的第 i 列,则疾病![]() 与
与![]() 之间的GIP核相似度
之间的GIP核相似度![]() 可定义为:
可定义为:
![]()
其中,![]() 用于控制核带宽,
用于控制核带宽,![]() 的值按前人[14]的研究设为1。
的值按前人[14]的研究设为1。
疾病整合相似度(Integrated similarity for diseases)
利用疾病语义相似度DSS和疾病的GIP核相似度DGS,可以构造综合的疾病相似度矩阵![]() ,定义元素
,定义元素![]() 如下:
如下:

miRNA-miRNA相似性
本研究将miRNA功能相似性和GIP核相似性结合起来,来表征miRNA之间的关系,如图1右侧所示。
miRNA 功能相似性
基于具有近似功能的miRNA通常与相似疾病有关的假设[39],可以计算miRNA的功能相似性。 本文从MISIM数据库(http://www.cuilab.cn/fi les/images/cuilab/misim.zip)中获得了miRNA之间的功能相似性[22,41],构建了miRNA功能相似性矩阵![]() ,其中
,其中![]() 表示miRNA mi 与 mj 之间的功能相似性。
表示miRNA mi 与 mj 之间的功能相似性。
miRNAs的GIP核相似度
同样地,还引入了GIP内核来计算miRNAs之间的网络拓扑相似性,计算如下:
![]()
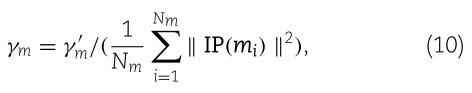
其中二值向量![]() 为邻接矩阵MDA的第 i 行,该矩阵记录了miRNA mi与每种疾病的关联信息,元素
为邻接矩阵MDA的第 i 行,该矩阵记录了miRNA mi与每种疾病的关联信息,元素![]() 表示miRNA mi 与 mj 的GIP核相似度,
表示miRNA mi 与 mj 的GIP核相似度,![]() 值也相应地设为1。
值也相应地设为1。
miRNAs的整合相似度
与疾病相似,我们将 miRNA 的功能相似性和 GIP 核相似性结合起来,构造了一个miRNA整合相似矩阵,命名为![]() 。 元素
。 元素![]() 定义如下:
定义如下:

基于重采样方法的集成学习框架用于miRNA疾病关联(Ensemble learning framework with Resampling method for miRNA-Disease Association)
为了推断潜在的miRNA与疾病的关联,在本研究中,我们提出了一个基于重采样方法的集成学习框架命名为ERMDA。 受前人研究的启发,在构建集成学习框架时,学习器的个体应该是不同的,最终的学习结果可以通过投票的方法来确定。 例如,为了确保个体学习器表现良好且彼此不同, EDTMDA[33]构建了多个不同的子集来训练基于决策树的学习器,而RPITER[42]中的个体学习器在异构特征组合上训练。 此外,为了整合个体学习器的预测结果,许多方法[43、44]引入了软投票策略来做出最终决策,它呈现出比单个学习者更好的总体结果。 因此,在本工作中,我们主要从以下两个方面来处理学习器的不同:(一)开发重采样方法来构建多个不同的训练子集;(二)应用特征选择策略来构建异构特征空间。 为了结合个体学习器的原始结果,采用软投票方法产生最终预测。
重采样策略 (Resampling strategy)
与未经证实的miRNA与疾病的关联相比,只有一小部分关联得到了实验验证。 我为了解决这种不平衡数据集引起的问题,使 ERMDA 框架中的个体学习者不同,我们提出了构建多个不同平衡训练子集的重采样方法。具体来说,随机选择一些未标记的miRNA疾病对作为阴性样本,然后将它们与阳性训练样本组合,以便在构造子集时平衡阳性和阴性样本。 如图2所示,![]()
![]() 分别表示阳性样本和未标记样本的训练集。 每个训练子集的无标号样本数与阳性标号样本数相同,即
分别表示阳性样本和未标记样本的训练集。 每个训练子集的无标号样本数与阳性标号样本数相同,即![]() ,其中n表示训练子集的个数。
,其中n表示训练子集的个数。

miRNA-疾病特征表示与选择(MiRNA-disease feature representation and selection)
为了全面学习miRNA和疾病的潜在特征表示,我们结合多个数据源计算miRNA和疾病的集成相似性(见图1)。 然后,可以得到第 i 个疾病的特征向量![]() 如下:
如下:
![]()
其中![]() 表示疾病
表示疾病![]() 之间的整合相似度。 此外,第 j 个 miRNA的特征向量
之间的整合相似度。 此外,第 j 个 miRNA的特征向量![]() 可由如下所示得到:
可由如下所示得到:
![]()
其中![]() 表示miRNA
表示miRNA![]() 之间的集成相似度。 然后,我们通过将
之间的集成相似度。 然后,我们通过将![]() 组合如下来表示每个 Disease-Mirna 对
组合如下来表示每个 Disease-Mirna 对![]() :
:

从方程(14),我们可以看到miRNA-疾病特征向量的长度取决于数据库中包含的miRNAs和疾病的数量,这可能特别大。 针对这一问题,在ERMDA框架中,我们采用特征选择方法从高维特征中捕获基本特征组合,减少冗余信息对个体学习器的干扰。 此外,值得注意的是,在构建集成学习框架时,在异构特征空间上训练的个体学习器可以获得更好的泛化能力。 因此,进行特征选择可以增加所构造的训练子集之间的多样性,从而提高集成框架的性能。通过一系列实验,将RF应用于特征选择。 我们首先通过在子集上训练一个RF模型来计算每个特征的重要性得分。 然后,将所有特征按重要度得分降序排列,并选择 top-f 特征表示每个子集内的训练样本。
基于软投票的集成学习框架
集成学习是一种基于机器学习的方法,它将多个个体学习器结合起来以提高预测性能,通常用于处理样本不平衡问题[45]。 在本文中,我们提出了一个基于软投票的多样本子集异构特征空间的集成学习框架。 如图2所示,给定重采样后的 n 个训练子集,我们首先在每个数据集上构建一个单独的学习器。 由于子集的不同以及这些子集的特征空间也是异构的,训练出来的个体学习者之间也是不同的。 在这里,我们使用极端梯度Boosting(XGBoost)[46]模型作为个体学习器,这是一种有效的基于树的方法。 将 n 个学习者的输出作为原始预测结果。 然后,将这些原始结果综合起来,引入软投票方法对给定的miRNA-疾病对进行最终决策。 它平均每个学习器的概率分数,并最终确定样本中的miRNA是否与疾病相关。 软投票法对第 i 个样本![]() 的预测输出计算如下:
的预测输出计算如下:

其中,![]() 表示第k个个体学习器对于第 i 个样本的概率得分。 在本研究中,
表示第k个个体学习器对于第 i 个样本的概率得分。 在本研究中,![]() >0.5代表样品i的miRNA与疾病有关; 否则,它们无关联。
>0.5代表样品i的miRNA与疾病有关; 否则,它们无关联。
结果(Result)
在本研究中,为了评估ERMDA的性能,在HMDD数据库上进行了一系列实验。 首先,我们研究了使用不同的机器学习模型作为个体学习器对ERMDA性能的影响。 其次,比较了不同特征选择方法下ERMDA框架的性能,验证了特征选择的有效性。 再次,研究了基于软投票的集成学习策略在ERMDA中的有效性。 最后,将该方法与基于网络的方法和基于机器学习的方法进行了比较。
实验设置和评价指标(Experimental settings and evaluation metrics)
在本研究中,我们采用了五折交叉验证来综合评估ERMDA在我们实验中的性能。 将已验证的miRNA与疾病关联随机分为5组,每组作为测试集,其余4组依次作为训练样本,然后选择与该组相同数量的未标记样本加入测试集。 在此基础上,采用了多种指标来衡量ERMDA的预测性能,包括准确率、查全率、F1评分、接收机操作特性曲线下面积(AUROC)和AUPR。
基于不同个体学习器的ERMDA框架性能分析(Performance analysis of ERMDA framework using different individual learners)
在所提出的ERMDA框架中,使用不同的机器学习模型作为个体学习器不可避免地会影响其预测性能。 在此,我们测试了几种机器学习模型,以研究它们在不使用特征选择方法的情况下对ERMDA性能的影响,如RF、极度随机化树(ERT)、adaboost(AB)和极端梯度增强(XGB)。 为了便于描述,将以RF、ERT、AB和XGB为个体学习者的框架分别表示为ERMDA-RF、ERMDA-ERT、ERMDA-AB和ERMDA-XGB。 为了客观地比较这四种方法,对学习者个体的几个重要的超参数进行了调整。 例如,RF和ERT的超参数调整范围为n_estimators从100~500,间隔为100,max_depthh取自{10,20,30,50};AB和XGB的超参数调整范围为n_estimators从{50,100,200,300,500},学习率取自{0.3,0.5,0.8}。 ERMDA的个体学习器数n在2~20之间,这是影响ERMDA性能的一个重要的超参数。 我们选择了每种方法在不同超参数下的最优性能进行比较,实验结果列于表1。 ERMDA-XGB的Precision为0.8681,Recall为0.9041,F1-score为0.8857,AUROC为0.9540,AUPR为0.9525,在五个指标上完全优于ERMDA-AB和ERMDA-ERT,在后四个指标上优于ERMDA-RF。 特别是在RE方面,ERMDA-GBDT方法远远高于其他方法,表明ERMDA-XGB可以成功地识别出较多阳性样本。 因此,XGBoost模型更适合作为MIRNA与疾病关联预测的ERMDA框架的个体学习器。
ERMDA框架中特征选择方法的有效性(Effectiveness of the feature selection method in the proposed ERMDA framework)
为了自动构造异构特征空间,减少冗余信息的干扰,在每个子集上引入了特征选择方法。 为了验证特征选择的有效性,我们采用了三种不同的特征选择模型。
(i) RF: 根据各特征在 RF 中的重要性得分,对特征进行降序排序,然后选择 top - f 特征来表示每个子集的训练样本;(ii)主成分分析(PCA):PCA是一种无监督学习算法,常用于降低输入特征的维数; 以及(iii)堆叠式自动编码器(SAE):SAE由多个自动编码器组成,可以提取具有鉴别性的高级特征。 为方便起见,它们分别表示为ERMDA-XGB-RF、ERMDA-XGB-PCA和ERMDA-XGB-SAE。 在实验中,我们分别利用30%、50%和80%的原始特征来训练个体学习者,并对三种特征选择模型的超参数进行了调整。 然后,我们在最佳AUPR值下选择每种特征选择方法的特征比率。 最后,比较了采用不同特征选择方法和不采用特征选择方法(ERMDA-XGB)的性能。 如表2所示,尽管在使用PCA和SAE进行特征选择后,这些方法的性能有所下降,但ERMDA-XGB-RF在所有五个指标上都优于ERMDA-XGB。 结果表明,基于RF的特征选择策略有利于减少冗余信息的干扰,提高预测性能。

基于软投票的集成学习方法的有效性(Effectiveness of the soft voting-based ensemble learning method)
为了验证基于软投票的集成学习框架的有效性,我们将ERMDA所有个体学习器的性能进行了比较。 比较结果如图3所示,横轴表示ERMDA框架中单个学习器的索引数,纵轴表示AUPR值。 此外,图中的红线代表ERMDA框架实现的AUPR。 如图3所示,单个学习者的AUPR在0.9381到0.9387之间。 另外,ERMDA的AUPR(0.9542)比13个单独学习者获得的最佳AUPR(0.9387)高1.65%。 实验结果表明,本文提出的基于软投票的集成学习框架能够显著提高预测模型的性能。 这也表明,发展基于集成学习的方法是发现潜在的miRNA与疾病关联的可行途径。

与最新方法的比较(Comparisons with the state-of-the-art methods)
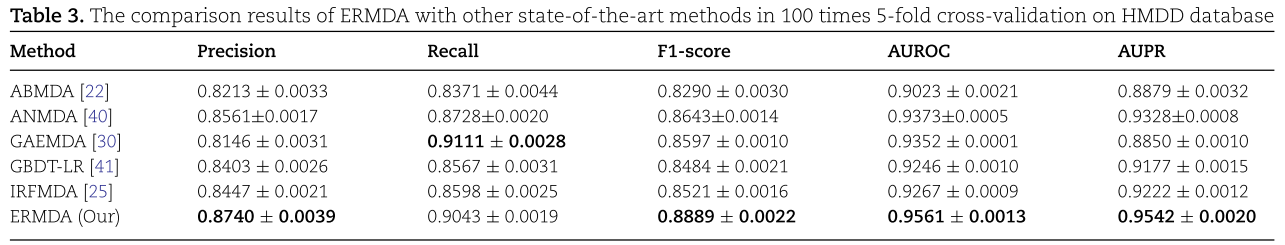
为了评价我们提出的ERMDA方法的性能,我们首先在正负样本数相等的平衡测试集上测试了它和其他现有的方法。 为了进行比较,在相同的实验条件下,在HMDD数据库上进行5次交叉验证,重复100次。 每个评价指标100次重复的平均值和SD作为方法的最终结果。 试验方法包括ABMDA[22]、ANMDA[40]、GBDT-LR[41]、IRFMDA[25]和GAEMDA[30]。 对于GAEMDA方法[30],我们下载了它的源代码,并根据推荐的超参数运行它。 对于其他方法,我们从研究[40]中获得了它们的测试结果,这些测试是在与我们相同的测试条件下进行的。 在平衡测试集上的比较结果如表3所示。 ERMDA的准确率为0.8740±0.0039,查全率为0.9043±0.0019,F1-score为0.8889±0.0022,AUROC为0.9561±0.0013,AUPR为0.9542±0.0020,在总体评价指标上优于其他方法。 ERMDA的查全率低于GAEMDA,但其准确度、F1评分、AUROC和AUPR分别比GAEMDA高0.0594、0.0292、0.0209和0.0692。 ERMDA在精确度、F1-Score、AUROC和AUPR方面分别以0.0179,0.0246,0.0188和0.0214超过第二优方法,表明ERMDA在平衡测试集上取得了优于其他比较方法的性能。
此外,为了验证 ERMDA 框架中重采样策略的有效性,我们还对不同比例的正、负样本进行了一系列不平衡测试集的实验。在上述实验中使用的测试集的基础上,我们随机选取了一些未标记的样本来构造非平衡测试集。 然后,我们比较了ERMDA与ERMDA-nores和GAEMDA[30]在正负样本比例为1:2~1:20的测试集上的性能。 这里,ERMDA-nores是不使用重采样方法的ERMDA版本。 GAEMDA也被测试了,因为它是最先进的方法之一,它的源代码可以适应不平衡的测试集。 另外,由于平衡数据集和非平衡数据集的本质区别,在评价方法时,各种度量指标的重要性也是不同的。 对于非平衡任务,AUPR更适合作为评价指标,因为它能严重惩罚假阳[47]。 上述三种方法在不同比例样品上的AUPR值如图4所示。 如图所示,随着阴性样本的增加,虽然ERMDA的性能有所下降,但在所有正负样本比例下,其性能仍优于其他比较方法。

特别是,随着不平衡度的增加,ERMDA的性能优势更加显著。 例如,当样本比例为1:5、1:10、1:15和1:20时,ERMDA获得的AUPR平均值分别为0.8373、0.7505、0.6890和0.6365,比GAEMDA分别高出14.37、18.76、20.01和20.77%,比ERMDA-nores分别高出6.74、11.60、15.41和17.98%。 因此,ERMDA框架中的重采样策略能够有效地解决样本不平衡问题。
综上所述,实验结果表明ERMDA不仅在平衡测试集上优于现有的其他方法,而且在非平衡测试集上也取得了更好的性能。 这表明我们提出的方法能够根据不平衡的数据集预测miRNA与疾病的关联。
案例研究 (Case studies)
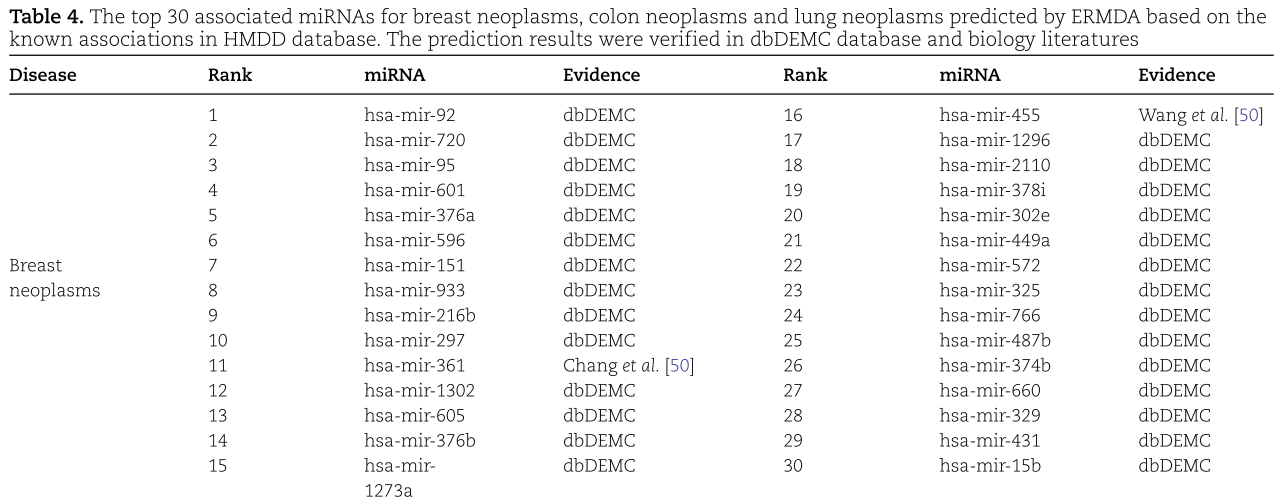
为了进一步证明ERMDA在发现潜在的与疾病相关的miRNAs方面的预测能力,我们对乳腺肿瘤、结肠肿瘤和肺肿瘤这三种重要的人类疾病进行了案例研究[48]。 首先,应用 HMDD 数据库[36]中的已知关联作为 ERMDA 的训练集,然后利用 ERMDDA 预测剩余的实验未证实的样本。将所有候选miRNAs按预测概率分数降序排列,分别选出每种被调查疾病的前30名候选miRNAs,最后用相关权威数据库dbDEMC[37]和生物学文献进行验证。
一些研究发现miRNAs在乳腺癌细胞的发生、转移、增殖和分化过程中起着至关重要的作用[49]。 如表4所示,所有前30个候选miRNAs都被证实与乳腺肿瘤相关,其中28个候选miRNAs被dbDEMC数据库验证。 而且Chang等人[50]发现miR-361的高表达是提高乳腺癌患者生存率的重要因素,Wang等[51]表明miR-455在乳腺癌组织细胞中下调。 结肠肿瘤,又称结直肠癌,是胃肠道常见的恶性肿瘤[52]。 从表4中我们可以看到26个结肠肿瘤相关的miRNAs被dbDEMC数据库确认。 此外,大量研究证实miR-629和miR-483参与结肠癌的发生发展[53,54]。 新的证据表明,肺癌是世界上最具破坏性的恶性肿瘤和癌症死亡的主要原因[55,56]。 一些miRNAs可作为肺癌的生物标志物[11]。 在此,根据dbDEMC数据库,在前30个预测的miRNAs中,有29个被证实与肺肿瘤相关。 总体而言,病例研究进一步表明ERMDA在有效预测miRNAs与疾病之间的潜在关联方面取得了很好的预测效果。

结论(Conclusion)
利用计算方法识别miRNA与疾病的关联对于理解人类复杂疾病的病理机制、诊断和治疗具有重要意义。 在本研究中,提出了一个结合重采样方法的集成学习框架来预测疾病相关的miRNAs,命名为ERMDA。 我们首先通过整合mirna-miRNA相似性、疾病-疾病相似性以及miRNA与疾病之间的关联信息,获得了miRNA与疾病的潜在特征表示。 然后,针对样本不平衡的问题,引入重采样方法和特征选择方法,构建多个异构特征空间的平衡训练子集。最后,我们在每个子集上构造了一个个体学习器来产生原始结果,并根据个体学习器的预测结果使用了软投票方法来做出最终决策。 为了评价ERMDA的性能,在HMDD数据库上进行了一系列的五折交叉验证实验。我们将它与其他相关的平衡测试集和非平衡测试集的方法进行了比较,结果表明,ERMDA 方法的性能优于目前最先进的方法,可以有效地解决样本不平衡引起的问题。此外,对三种常见复杂疾病的案例研究表明,我们提出的方法可以在miRNAs水平上指导繁重的临床生物学实验,并进一步表明基于软投票的集成学习框架和重采样方法是预测潜在miRNA-疾病关联的一种有效可行的方法。

Data availability
The source code and datasets are available at GitHub - Wang-Zhaowei/ERMDA: #ERMDA v1.0。
