前言
在4月份,我出了基于TBtools做基因家族分析,这个教程80%的分析都是基于TBtools,非常适合我们做基因家族的小白。那么,最近又在做一些相关的优化。今天在做Seqlogo图,也就查了一些教程,也做个记录吧。
– Du

Seqlogo图
在meme网址中也也给你Seqlogo图,可以直接使用,只是需要手动组图。


下载流程
- Step one
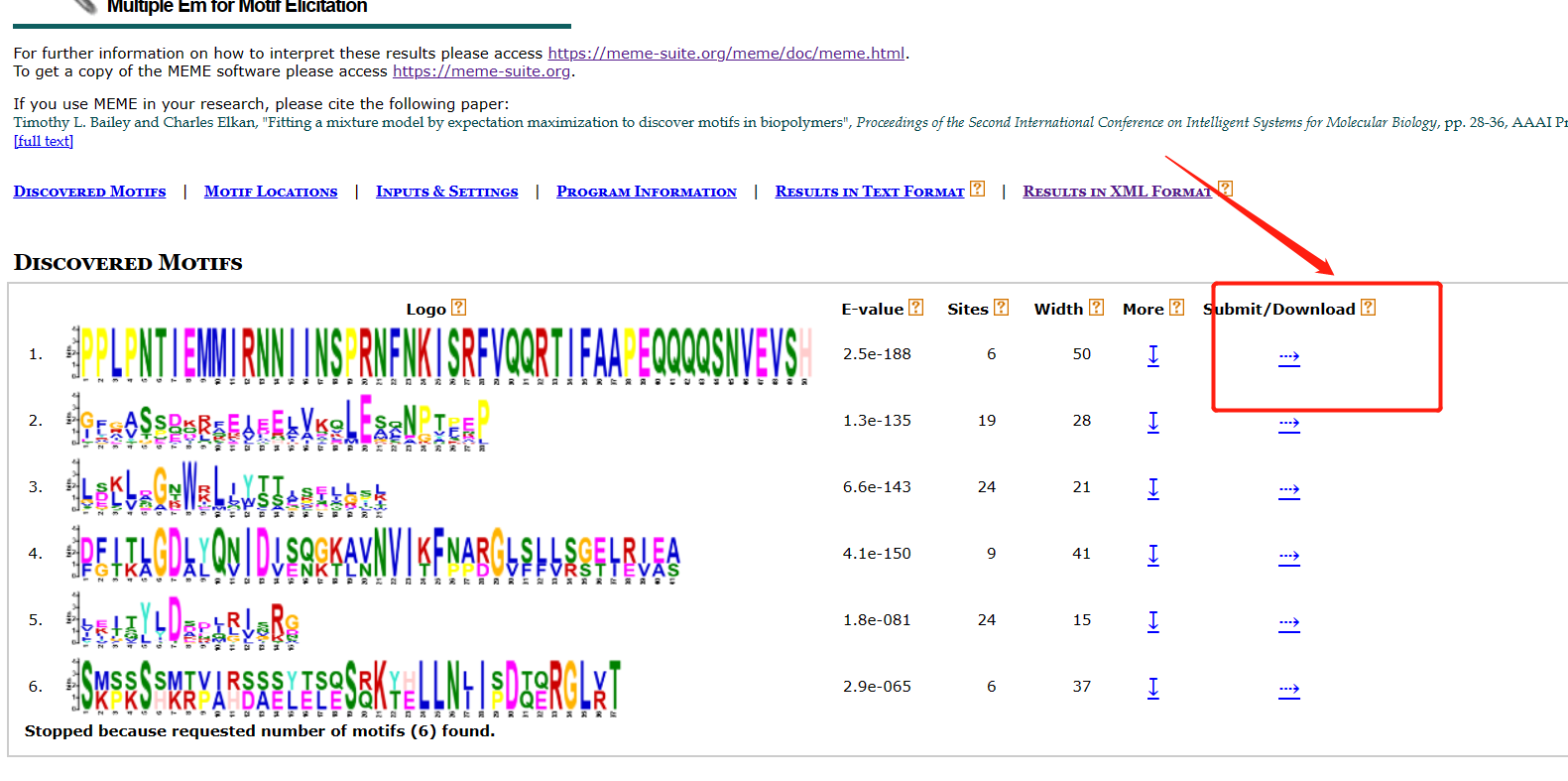
- Step Two

- ENDING !
基于ggseqlogo包绘制
ggseqlogo包发表在Bioinformatic期刊中,使用相对简洁、方便,但是感觉批量绘制时颜色很难修改(PS:应该自己参数没使用对)。

文章网址:
https://academic.oup.com/bioinformatics/article/33/22/3645/3980251?login=false
ggseqlogo包使用网址:
https://omarwagih.github.io/ggseqlogo/
在网址中有比较详细的文档说明。
安装R包
install.packages("ggseqlogo")
or
devtools::install_github("omarwagih/ggseqlogo")
加载所需包
# Load the required packages
require(ggplot2)
require(ggseqlogo)
我们这里就直接使用ggseqlogo包中序列信息吧。
如果是自己导入序列的话,可以使用read.table()函数导入。
批量导入:
·## 批量生产文件名
filelist = c(paste0('motif',1:10,'.txt'))
filelen <- length(filelist)
##批量读取
data.list <- list()
for (i in 1:10) {
data.list[[paste0('motif',i)]]=scan(filelist[i],what = '')
}
# Some sample data
data(ggseqlogo_sample)
基础图形:
ggplot() + geom_logo( seqs_dna$MA0001.1 ) + theme_logo()

添加相关参数:
氨基酸,DNA和RNA序列类型都支持ggseqlogo。默认情况下,ggseqlogo将尝试猜测您的序列类型。您可以通过seq_type选项显式地设置序列类型。
ggseqlogo( seqs_aa$AKT1, seq_type='aa' )

以数字的形式展现
# Replace DNA characters with numbers
seqs_numeric = chartr('ATGC','1234', seqs_dna$MA0001.1)
ggseqlogo(seqs_numeric, method='p', namespace=1:4)

# Replace DNA characters with Greek ones
seqs_greek = chartr('ATGC', 'δεψλ', seqs_dna$MA0001.1)
ggseqlogo(seqs_greek, namespace='δεψλ', method='p')

颜色的调整
使用col_scheme进行调整
ggseqlogo(seqs_dna$MA0001.1, col_scheme='base_pairing')

col_scheme参数有如下几种; auto, chemistry, chemistry2, hydrophobicity, nucleotide, nucleotide2, base_pairing, clustalx, taylor
指定颜色
## 设置颜色
cs1 = make_col_scheme(chars=c('A', 'T', 'C', 'G'), groups=c('gr1', 'gr1', 'gr2', 'gr2'),
cols=c('purple', 'purple', 'blue', 'blue'))
##
ggseqlogo(seqs_dna$MA0001.1, col_scheme=cs1)

颜色二
cs2 = make_col_scheme(chars=c('A', 'T', 'C', 'G'), values=1:4)
# Generate sequence logo
ggseqlogo(seqs_dna$MA0001.1, col_scheme=cs2)

批量绘制
ggseqlogo(seqs_dna, ncol=4)
## ncol:指定每行的展示个数

自定义高度
# Create a custom matrix
set.seed(123)
custom_mat = matrix( rnorm(20), nrow=4, dimnames=list(c('A', 'T', 'G', 'C')))
# Generate sequence logo
ggseqlogo(custom_mat, method='custom', seq_type='dna') + ylab('my custom height')

更详细的内容你可以看的包帮助文档!!!
往期文章:
1. 最全WGCNA教程(替换数据即可出全部结果与图形)
2. 精美图形绘制教程
3. 转录组分析教程
- 1.课程介绍
- 2.第一章 Linux基础
- 3.第一章 生信软件安装
- 4.第二章 转录组数据的下载
- 5.第三章 参考基因组序列和注释文件的下载
- 6.转录组上游分析教程 | 第四章 数据过滤
- 7.第四章 Hisat2进行数据比对
- 8.第四章 Bowtie2进行数据比对
- 9.第四章 BWA进行数据比对
- 10.第四章 Tophat2比对
- 11.第五章 无参考基因组的转录组分析
小杜的生信筆記,主要发表或收录生物信息学的教程,以及基于R的分析和可视化(包括数据分析,图形绘制等);分享感兴趣的文献和学习资料!!