Integration of pairwise neighbor topologies and miRNA family and cluster attributes for miRNA–disease association prediction

Abstract
识别疾病相关的小 RNA (miRNA)有助于理解疾病的发病机制。现有的研究方法综合了与 miRNA 和疾病相关的多种数据来推断候选疾病相关的 miRNA。然而,miRNA 节点的属性包括它们的家族和簇属性信息,还没有被深入整合。此外,对一对 miRNA 和疾病的邻域拓扑表示的学习也是一个具有挑战性的问题。我们提出了一种疾病相关的 miRNA 预测方法,通过编码和整合miRNA 和疾病节点的从生成性和对抗性的角度学习到的多种表征。我们首先构建 miRNA 和疾病节点的双层异质网络,它包含这些节点之间的多种类型的连接,这反映了 miRNA-疾病对的邻域拓扑结构,以及 miRNA 节点的属性,特别是 miRNA 相关的家族和簇。为了学习增强的成对邻域拓扑结构,我们提出了一个基于卷积自编码器的生成对抗模型来编码 miRNA-disease 对的低维拓扑表征,以及基于多层卷积神经网络的识别器来区分真假邻域拓扑嵌入。此外,我们还设计了一个新的特征类别水平的注意力机制来学习不同特征在最终自适应融合和预测中的各种重要性。与5种 miRNA-disease 关联方法的比较结果表明,我们的模型在 ROC曲线下的面积和精确召回曲线下的面积具有优越的性能和技术贡献。召回率的结果证实,我们的模型可以找到更多的实际 miRNA 疾病的关联排名最高的候选者。对三种癌症的病例研究进一步证明了检测潜在候选 miRNA 的能力。
关键词: miRNA-疾病关联预测; 生成对抗网络; 成对邻居拓扑; 具有节点属性的双层异质网络;特征类别水平的注意力
目录
2.3.2.特征类别水平的注意机制(Attention mechanism at the feature category level)
2.3.3.基于卷积神经网络的预测得分评价(Predictive score evaluation based on convolution neural network)
1.引言
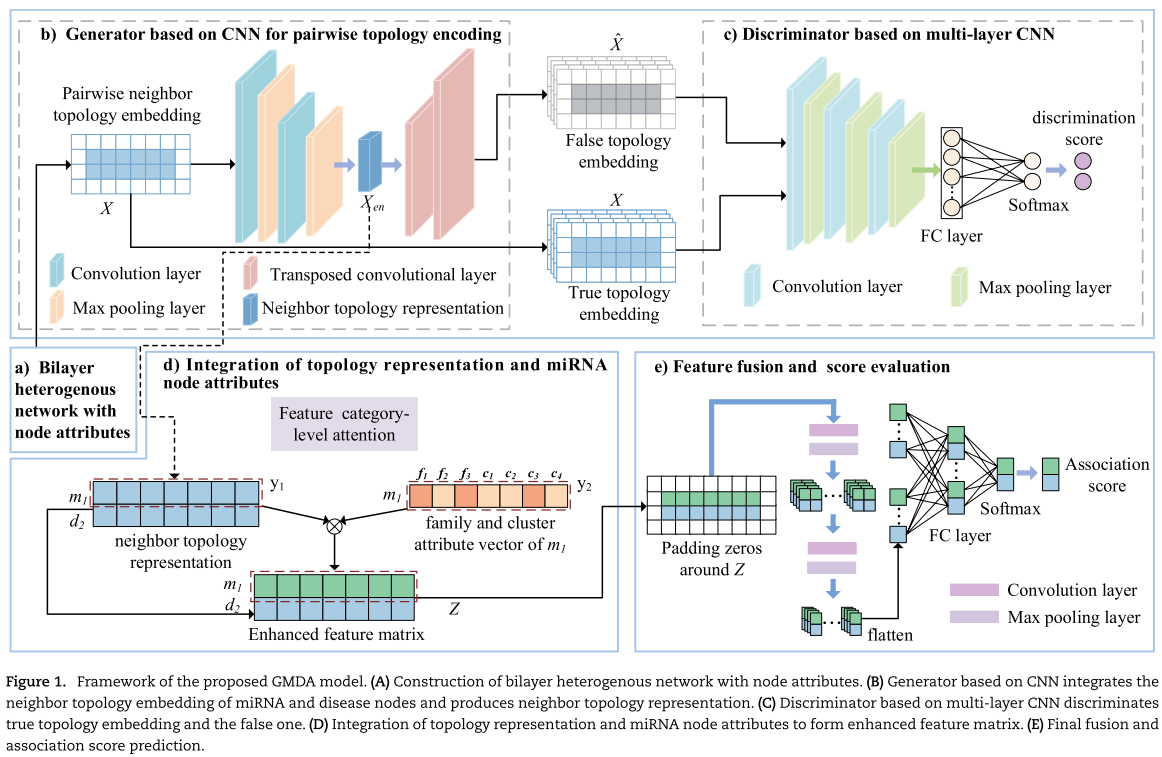
在这项工作中,我们提出了一种预测方法,生成性 miRNA 疾病关联(GMDA) ,以充分整合 miRNA 和疾病相关数据以便于疾病相关候选人 miRNA 筛选。为了整合多种节点类型和节点之间不同的连接,我们首先构建了一个具有 miRNA 疾病节点属性的双层异质网络。然后提出了一种基于生成对抗网络(GAN)的预测模型来学习和编码增强的成对邻域拓扑结构、家族和簇属性信息。输出关联得分表明一对 miRNA 疾病相关的可能性。分数越高,它们之间的联系就越紧密。我们模型的独特贡献如下:
①首先,我们构建了一个具有节点属性的双层异质网络,以便于学习 miRNA 疾病节点的邻域拓扑表示。该网络由多种类型的连接组成,以嵌入 miRNA 与疾病、 miRNA 相关家族和簇属性之间的相似性和关联。我们还设计了一种嵌入机制来从网络中提取成对邻域拓扑结构。
②其次,我们利用生成和对抗的思想来学习一对 miRNA 和疾病节点的增强表示。该生成器由自动编码器和解码器组成,用于生成嵌入节点对的假的邻域拓扑特征。该编码器基于多层卷积神经网络编码节点对的邻域拓扑表示。然后基于多层转置卷积译码器重构节点对的邻域拓扑嵌入。
③该鉴别器基于多层 CNN 神经网络来区分由生成器生成的伪邻域拓扑嵌入和原始真拓扑嵌入。该判别策略有利于生成器生成尽可能接近真实嵌入的邻域拓扑,并获得 miRNA-disease 节点对的最终邻域拓扑表示。
④由于 miRNAs 的邻域拓扑结构和属性特征对预测 miRNA 与疾病的关联具有不同的贡献,我们提出了一个注意力特征类别水平模块来区分这两类特征对自适应性融合的贡献。对公共数据集的全面评估和比较表明,我们的技术创新的性能和贡献有所提高。
2.材料和方法
2.1.数据集
在这项研究中,miRNA 疾病关联是从公共数据库[39]中提取的,其中包含7908个 miRNA 疾病关联,涵盖793个 miRNA 和341个已经通过实验验证的疾病。一种疾病通常由几个相关术语组成。该疾病的术语信息摘自美国医学图书馆[40]。疾病之间的语义相似性计算使用术语信息来构造有向无环图(DAGs)[7]。我们从 miRBase [33]中提取 miRNA 家族信息,并通过设置两个不超过20kb 的 miRNA 之间的距离来获得聚类信息。
2.2.具有节点特征的 miRNA-疾病双层异质网路
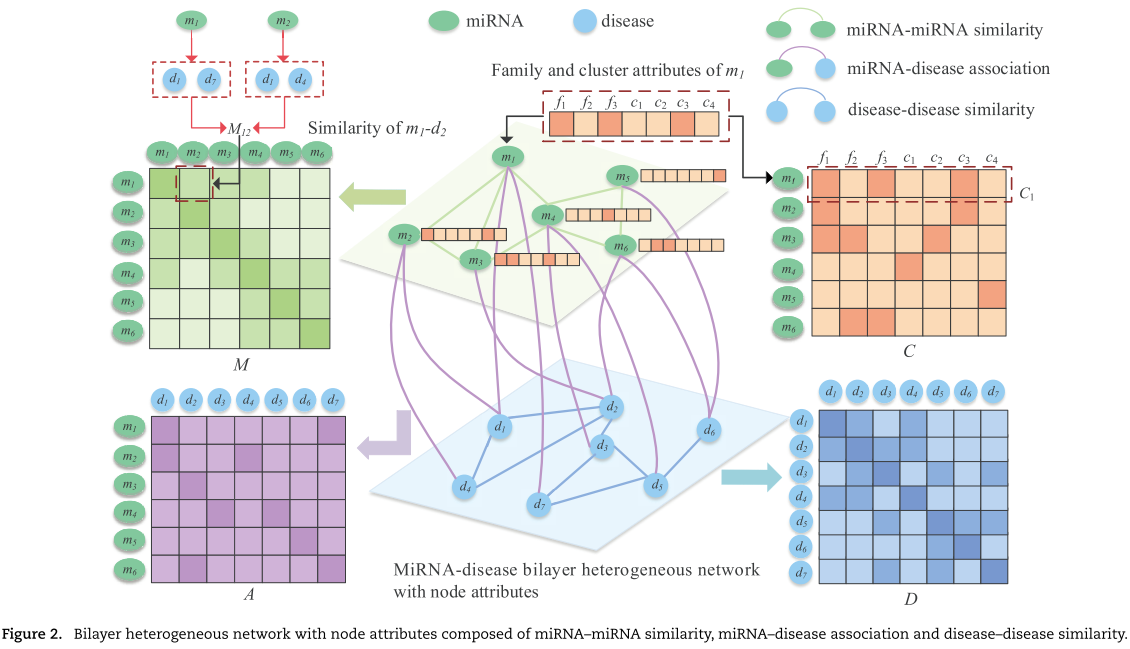
由于不同的连接从不同的角度反映了 miRNA 与疾病的关联,而 miRNA 所属的家族和聚类属性也是重要的辅助信息,我们构建了具有节点属性的 miRNA 和疾病双层异质网络,如图1所示。设 G = (V,E)表示双层 miRNA-疾病网络节点集 ![]() 由一系列 miRNA 节点
由一系列 miRNA 节点 ![]() 和疾病节点
和疾病节点 ![]() 组成。节点对
组成。节点对 ![]() 通过边
通过边 ![]() 连通。该网络包括 miRNA 与疾病之间的各种联系,包括疾病-疾病相似性、 miRNA-miRNA 相似性以及 miRNA 与疾病的关联。此外,miRNA 节点还包含其独特的生物学特性,即它所属的家族和聚类信息。
连通。该网络包括 miRNA 与疾病之间的各种联系,包括疾病-疾病相似性、 miRNA-miRNA 相似性以及 miRNA 与疾病的关联。此外,miRNA 节点还包含其独特的生物学特性,即它所属的家族和聚类信息。
2.2.1.MiRNA 相似性网络的构建
一般来说,如果两种 miRNA 具有相似的功能,它们可能与相似的疾病有关。因此,两组 miRNA 相关疾病被用来计算 miRNA 之间的相似性[7]。例如,假设 miRNA m1与疾病 d1,d3和 d6相关,并且 miRNA m2与疾病 d3,d5,d6和 d7相关。将疾病集 d1,d3,d6与 d3,d5,d6,d7的相似性作为 m1与 m2的相似性,表示为 M12。利用 miRNA 与疾病之间的关联,我们使用 Wang 的方法[7]来计算 miRNA 的相似性。为了构建 miRNA 的相似性网络,我们计算了所有 miRNA 节点对之间的相似性值,并将它们连接起来,如果它们的相似性大于0。此外,相似度值被作为两个节点边缘的权重(图2)。该网络由一个相似矩阵 ![]() 表示,其中 miRNA mi 和 miRNA mj 之间的相似性用
表示,其中 miRNA mi 和 miRNA mj 之间的相似性用 ![]() 表示,其值在0 ~ 1之间。
表示,其值在0 ~ 1之间。
2.2.2.疾病相似性网络的构建
为了构建疾病相似性网络,需要计算疾病之间的相似性。我们利用 Wang 的 DAG 方法[7]来计算疾病之间的相似性。简而言之,一种疾病是由包含与该疾病相关的所有疾病术语的 DAG 表示的。这两种疾病的 DAGs 含有的疾病术语越多,它们就越相似。
将相似度大于0的所有疾病对连接起来,将相似度作为加权边值,在此基础上构造疾病相似度网络。它可以用矩阵 ![]() 表示,其中 Dij 表示疾病 di 与疾病 dj 之间的相似性,相似性值在0和1之间。
表示,其中 Dij 表示疾病 di 与疾病 dj 之间的相似性,相似性值在0和1之间。
2.2.3.MiRNA-疾病关联网络的构建
鉴于 miRNA 相似性网络和疾病相似性网络,我们进一步构建了一个 miRNA-疾病关联网络。如果已知某个 miRNA 与某种疾病相关,我们将 miRNA 相似性网络中的 miRNA 节点连接到疾病相似性网络中的疾病节点。在此基础上,构建了 Nm miRNA 与 Nd 疾病之间的关联矩阵 ![]() 。每一行代表 miRNA 和所有疾病之间的关联,每一列代表疾病的类型。如果 Aij = 1,则表示 miRNA mi 和疾病 dj 被认为是相互关联的,并且当 Aij = 0时,在 miRNA mi 和疾病 dj 之间没有观察到关联。
。每一行代表 miRNA 和所有疾病之间的关联,每一列代表疾病的类型。如果 Aij = 1,则表示 miRNA mi 和疾病 dj 被认为是相互关联的,并且当 Aij = 0时,在 miRNA mi 和疾病 dj 之间没有观察到关联。
2.2.4.MiRNA 节点属性
如果 miRNA mi 和 miRNA mj 属于公共的家族或簇,它们可能与相同的疾病有关[41]。因此,miRNA 的家族和聚类信息在预测 miRNA 与疾病的关联中起着重要作用。使用矩阵 ![]() 表示 miRNA 家族和簇的信息(图2)。
表示 miRNA 家族和簇的信息(图2)。![]() 是矩阵C 的第 i 行,表明第 i 个 miRNA 属于 Nf 家族和 Nc 簇。Cij = 1表示属于一个家族(或簇)的第 i 个 miRNA; 否则,Cij = 0。
是矩阵C 的第 i 行,表明第 i 个 miRNA 属于 Nf 家族和 Nc 簇。Cij = 1表示属于一个家族(或簇)的第 i 个 miRNA; 否则,Cij = 0。
2.3.小RNA-疾病关联预测模型
我们提出了一种新的基于 GAN 的预测方法,用于推断潜在的与疾病相关的候选 miRNA (图1)。对于一对 miRNA 和疾病,同一家族或簇中的 miRNA 或具有较高功能相似性的 miRNA 更可能与相似的疾病相关[11]。因此,我们综合 miRNA 相似性、疾病相似性、 miRNA-疾病关联性、 miRNA 家族和聚类信息构建 miRNA-疾病对关联预测模型。
如图1所示,该模型由生成器、鉴别器和卷积模块组成。发生器和鉴别器之间的对抗学习产生 miRNA mi 和疾病 dj 的邻域拓扑表示,具有注意力机制的卷积神经网络模块集成了 mi 和 dj 的邻域拓扑表示、 mi 的节点属性表示,通过完全连通层和 softmax 层获得的 mi-dj 的关联预测得分。
2.3.1.miRNA - 疾病节点对的 GANs
基于 GAN 的网络结构主要由生成器和鉴别器组成。给定一对 miRNA 和疾病,发生器的主要目标是尽可能产生对应于节点对的假样本对。鉴别器试图区分虚假样本对和真实样本对。识别器训练得越好,生成器产生的鲁棒假样本对就越有帮助,这是迭代的。在这个迭代过程中,发生器和鉴别器的性能得到了显著的提高。最终的鉴别器能够准确地区分真假样本对,生成器的编码部分能够产生更加鲁棒的 miRNA-疾病对的低维邻居拓扑表示。
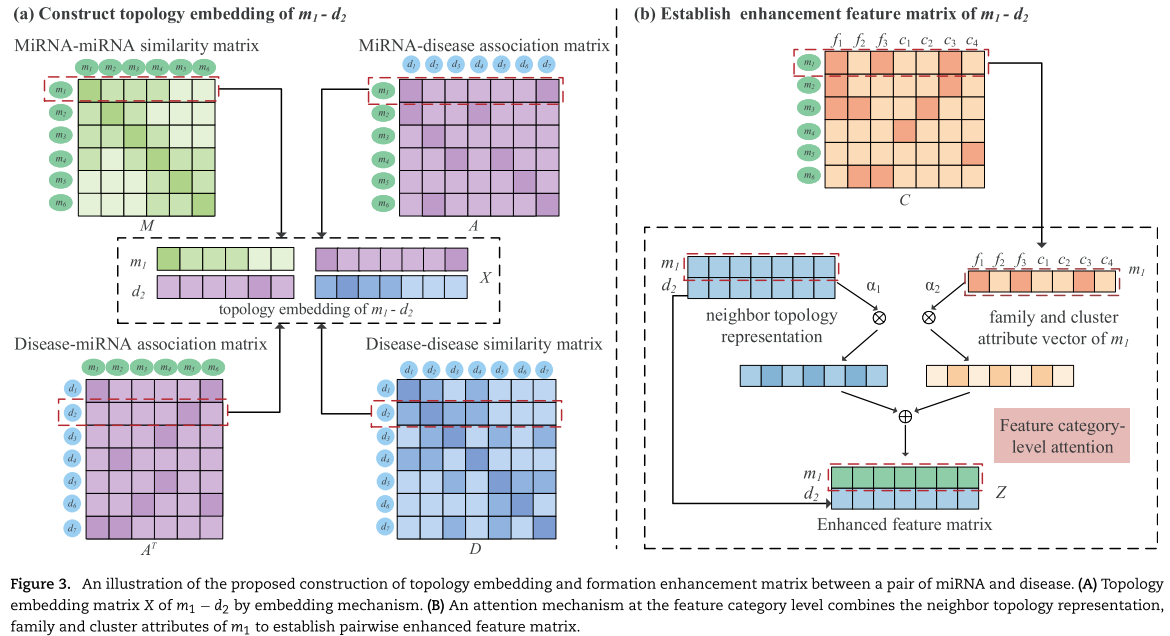
邻域拓扑嵌入矩阵的构造。构建 miRNA m1和疾病 d2节点对的嵌入矩阵的过程如图3A 所示。M1是 miRNA 相似性矩阵 M 的第一行,它代表了m1和所有 miRNA 连接边的信息,边的权重是 m1和每个 miRNA 的相似性。如图3A 所示,m1的边与 m2、 m3和 m4相连,表明它们具有相似的功能。MiRNA-疾病关联矩阵 ![]() 的第一行代表
的第一行代表 ![]() 和所有疾病之间的联系。一个连接表示有一个关联,没有连接表示它不存在或者没有关联。我们将前面和后面的连接记录为邻居拓扑特征向量
和所有疾病之间的联系。一个连接表示有一个关联,没有连接表示它不存在或者没有关联。我们将前面和后面的连接记录为邻居拓扑特征向量 ![]() 。类似地,
。类似地,![]() 和
和 ![]() 记录
记录 ![]() 与每种 miRNA 和疾病的基于关联的和相似的连接边。
与每种 miRNA 和疾病的基于关联的和相似的连接边。![]() 邻域拓扑特征向量由它们的前后拼接而成。最后,将
邻域拓扑特征向量由它们的前后拼接而成。最后,将 ![]() 和
和 ![]() 上下叠加形成节点 m1-d2对的邻域拓扑特征矩阵
上下叠加形成节点 m1-d2对的邻域拓扑特征矩阵 ![]() 。这个表示被认为是一个真实的例子。
。这个表示被认为是一个真实的例子。
基于卷积自动编码器的生成器。该生成器生成 miRNA-disease 节点对的重构邻域拓扑矩阵 ![]() ,该邻域拓扑矩阵
,该邻域拓扑矩阵 ![]() 被视为假样本。生成器的主要目标是使生成的
被视为假样本。生成器的主要目标是使生成的 ![]() 尽可能接近 X。给定一个节点对
尽可能接近 X。给定一个节点对 ![]() ,邻居拓扑嵌入为 X; 生成器
,邻居拓扑嵌入为 X; 生成器 ![]() 对它进行编码,如图1所示。
对它进行编码,如图1所示。
编码器。生成器编码器的学习框架由两个卷积层和最大池化层组成。为了学习边际信息,执行零填充并作为编码器中的第一个隐藏层输入。得到了编码器第1层隐层的输出 ![]() 。每个隐藏层表示如下:
。每个隐藏层表示如下:

其中’*’表示卷积运算。![]() 分别是
分别是 ![]() 卷积运算的权重矩阵和偏置向量。
卷积运算的权重矩阵和偏置向量。![]() 表示编码器层数。
表示编码器层数。![]() 表示非线性激活函数 Relu [42]和 max (.)表示最大池化操作,即在每个特征图卷积后进行下采样,以保留更重要的特征。
表示非线性激活函数 Relu [42]和 max (.)表示最大池化操作,即在每个特征图卷积后进行下采样,以保留更重要的特征。
解码器。采用基于双层转置CNN神经网络转置的框架重构 mi-dj 的邻域拓扑特征矩阵 ![]() 。为了得到一个更好的
。为了得到一个更好的![]() ,我们将其投影回原特征空间,计算
,我们将其投影回原特征空间,计算 ![]() 与原邻域拓扑特征矩阵 X 之间的误差。每个隐藏层的重建特征图表示如下:
与原邻域拓扑特征矩阵 X 之间的误差。每个隐藏层的重建特征图表示如下:

其中![]() 表示转置卷积运算,
表示转置卷积运算,![]() 是一个非线性激活函数 LeakyRelu 和
是一个非线性激活函数 LeakyRelu 和 ![]() 是解码器层数。得到了译码部分
是解码器层数。得到了译码部分![]() 隐层的权矩阵
隐层的权矩阵 ![]() 和偏置矢量
和偏置矢量 ![]() 。
。
生成器损失。在训练生成器时,我们希望产生尽可能接近原始 X 的重构邻域拓扑特征矩阵 ![]() ,以欺骗鉴别器。这样的鉴别者应该给出更高的分数。生成器的损失计算如下:
,以欺骗鉴别器。这样的鉴别者应该给出更高的分数。生成器的损失计算如下:
![]()
其中 ![]() 代表的是 miRNA 和疾病节点在 miRNA-疾病关联网络中的概率分布。利用
代表的是 miRNA 和疾病节点在 miRNA-疾病关联网络中的概率分布。利用 ![]() 从分布中选取样本对 mi-dj,构造其邻域拓扑特征矩阵 X。同样地,
从分布中选取样本对 mi-dj,构造其邻域拓扑特征矩阵 X。同样地,![]() 表明由 mi-dj 重构的邻域拓扑特征矩阵
表明由 mi-dj 重构的邻域拓扑特征矩阵 ![]() 是由生成器生成的。我们使用 Adam 优化算法使
是由生成器生成的。我们使用 Adam 优化算法使 ![]() 最小化,然后更新生成器
最小化,然后更新生成器 ![]() 的所有参数集。为了避免生成器过拟合,在参数集
的所有参数集。为了避免生成器过拟合,在参数集 ![]() 中加入了 L2 范数约束。
中加入了 L2 范数约束。![]() 是一个平衡生成器训练损失和正则项的参数。
是一个平衡生成器训练损失和正则项的参数。
基于多层卷积神经网络的鉴别器。给定任意节点对 mi-dj,其邻域拓扑特征矩阵 X 被视为真样本,其中基于 CNN 的自动编码器生成的![]() 被视为假样本。鉴别器试图区分真样本 X 和假样本
被视为假样本。鉴别器试图区分真样本 X 和假样本 ![]() ,从而促进自动编码器获得一个更好的 mi 和 dj 的邻域拓扑表示。鉴别器本质上是评估 X 和
,从而促进自动编码器获得一个更好的 mi 和 dj 的邻域拓扑表示。鉴别器本质上是评估 X 和 ![]() 是真或假样本的可能性。具体地说,对于真实样本 X,鉴别器应该给出一个较高的分数,而对于生成器生成的虚假样本 X,鉴别器输出一个较低的分数。设
是真或假样本的可能性。具体地说,对于真实样本 X,鉴别器应该给出一个较高的分数,而对于生成器生成的虚假样本 X,鉴别器输出一个较低的分数。设 ![]() 是鉴别器中
是鉴别器中![]() 卷积和池化后隐层的输出。
卷积和池化后隐层的输出。

其中 ![]() 分别是
分别是![]() 层的权重矩阵和偏置向量。
层的权重矩阵和偏置向量。![]() 是鉴别器中隐藏层的数目。
是鉴别器中隐藏层的数目。![]() 被激活函数 LeakyreLu 激活,然后输入全连通层,以获得真实样本和假样本的分数分布
被激活函数 LeakyreLu 激活,然后输入全连通层,以获得真实样本和假样本的分数分布 ![]() 。
。
![]()
其中 ![]() 表示鉴别器模型的一组参数。
表示鉴别器模型的一组参数。![]() 分别是鉴别器全连通层的权重矩阵和偏置向量。
分别是鉴别器全连通层的权重矩阵和偏置向量。
为了区分一对真样本和假样本的可能性,有以下两种可能的输入:
例1:miRNA - 疾病关联网络中的邻域拓扑特征矩阵 X。从 miRNA - 疾病关联网络中选取节点对 ![]() ,邻域拓扑特征矩阵定义为 X。我们期望在输入 X 之后,鉴别器输出一个高分,将损失定义为
,邻域拓扑特征矩阵定义为 X。我们期望在输入 X 之后,鉴别器输出一个高分,将损失定义为![]() 。
。
![]()
例2:由生成器生成的重构邻域拓扑特征矩阵![]() 。将 mi-dj 节点对的邻域拓扑矩阵 X 输入生成器,得到重构的邻域拓扑矩阵
。将 mi-dj 节点对的邻域拓扑矩阵 X 输入生成器,得到重构的邻域拓扑矩阵 ![]() 。模拟 X 邻居的拓扑信息,因此
。模拟 X 邻居的拓扑信息,因此 ![]() 非常接近 X。当判别器输入
非常接近 X。当判别器输入 ![]() 时,我们的目标是得到一个较低的分数,因此,我们将这部分的损失定义为
时,我们的目标是得到一个较低的分数,因此,我们将这部分的损失定义为![]() 。
。
![]()
假样本的邻域拓扑矩阵![]() 由编码器通过卷积生成,其维数与 X 相一致。该鉴别器的优化目标函数为
由编码器通过卷积生成,其维数与 X 相一致。该鉴别器的优化目标函数为 ![]() 。
。
![]()
第三项是减少模型的过拟合所建立的最优项,![]() 是一个权衡参数,以避免判别器的过拟合。我们利用Adam优化算法对鉴别器的参数集
是一个权衡参数,以避免判别器的过拟合。我们利用Adam优化算法对鉴别器的参数集![]() 进行更新。
进行更新。
在优化过程结束时,![]() 分别是矩阵的第1行和第2行,它们被表示为结点 mi 和 dj 的邻域拓扑。
分别是矩阵的第1行和第2行,它们被表示为结点 mi 和 dj 的邻域拓扑。
2.3.2.特征类别水平的注意机制(Attention mechanism at the feature category level)
MiRNA mi 和 dj 所属的家族和簇越一致,它们就越有可能与类似的疾病相关。因此,有必要对 mi 的邻域拓扑表示及其相关的族和簇信息进行集成。MiRNA-disease 的邻域拓扑结构特征及其家族聚类特征对 miRNA-disease 的关联预测有不同的贡献。因此,我们构建了一个特征类别水平注意力模块(图3B)。![]() 被认为是第一类特征,表示为
被认为是第一类特征,表示为 ![]() 。
。![]() 描述了
描述了![]() 所属的族和群,它被认为是第二类特征向量,表示为
所属的族和群,它被认为是第二类特征向量,表示为 ![]() 。第 i 类特征向量的注意得分为
。第 i 类特征向量的注意得分为 ![]() 。
。

其中![]() 是权重矩阵,
是权重矩阵,![]() 是偏置向量,
是偏置向量,![]() 是权重向量,
是权重向量,![]() 是一个指数函数,
是一个指数函数,![]() 是
是 ![]() 的标准化表示。最后,我们得到了两类 mi 加权求和向量 :
的标准化表示。最后,我们得到了两类 mi 加权求和向量 :

2.3.3.基于卷积神经网络的预测得分评价(Predictive score evaluation based on convolution neural network)
![]() 的邻居拓扑表示为
的邻居拓扑表示为![]() ,命名为
,命名为![]() 。
。![]() 和
和 ![]() 上下叠加形成 mi-dj 的增强的低维特征矩阵 Z。为了学习 Z 的边缘信息,进行零填充,然后输入卷积神经网络模块。对于卷积层,如果滤波器的长度为
上下叠加形成 mi-dj 的增强的低维特征矩阵 Z。为了学习 Z 的边缘信息,进行零填充,然后输入卷积神经网络模块。对于卷积层,如果滤波器的长度为 ![]() ,宽度为
,宽度为 ![]() ,数为
,数为 ![]() ,则滤波器
,则滤波器 ![]() 可应用于低维特征。Z 通过两个卷积和池化层后,可以得到特征映射
可应用于低维特征。Z 通过两个卷积和池化层后,可以得到特征映射 ![]() 。将平坦向量 h0输入到全连通层和 softmax 层,以获得最终的二进制评估结果得分:
。将平坦向量 h0输入到全连通层和 softmax 层,以获得最终的二进制评估结果得分:
![]()
其中 ![]() 分别表示全连通层的权重矩阵和偏置向量。
分别表示全连通层的权重矩阵和偏置向量。![]() 表示 mi 和 dj 的预测得分,
表示 mi 和 dj 的预测得分,![]() 表示它们无相关关系的预测得分。
表示它们无相关关系的预测得分。
