SGAEMDA: Predicting miRNA-Disease Associations Based on Stacked Graph Autoencoder


Abstract
小 RNA (miRNA)-疾病关联(MDA)预测对疾病的预防、诊断和治疗至关重要。另一方面,传统的 MDA 湿试验效率低下,成本高。因此,我们提出了一个多层次的协作无监督训练基础模型,称为 SGAEMDA (基于堆叠图自动编码器预测潜在的 miRNA 疾病关联)。首先,从原始的 miRNA 和疾病数据,我们定义了两种类型的初始特征: 相似性特征和关联特征。其次,然后使用堆叠的图自动编码器来学习有意义的高阶相似性特征的无监督低维表示,并且我们将关联特征与所学习的低维表示连接以获得最终的 miRNA-disease 对特征。最后,我们使用一个多层感知机(MLP)来预测未知 miRNA-疾病关联的得分。在5折交叉验证和10折交叉验证下,SGAEMDA 在 ROC 曲线下的平均面积分别为0.9585和0.9516,明显高于其他基线方法的交叉验证。此外,病例研究表明,SGAEMDA 可以准确预测脑、乳腺、结肠和肾肿瘤的候选 miRNA。
关键词: miRNA; 疾病; 关联预测; 堆叠图自动编码器; 高阶特征
目录
2.1.Datasets for MDA Prediction
2.2.MiRNA and Disease Informaton
2.2.1.MiRNA Function Similarity
2.2.2.Disease Semantic Similarity
2.2.3.Gaussian Interaction Profile Kernel Similarity of miRNAs and Diseases
2.2.4.Integration of miRNAs and Diseases Similarity
3.3.Prediction of miRNA–Disease Association Based on SGAEMDA
3.4.Effect of Similarity Feature Dimensions
3.5.Effect of Stacked Graph Autoencoder Pre-Training
3.6.Comparison of Different Classifier Models
3.7.Comparisons with Existing SOTA Methods
1.引言(Introduction)
尽管上述模型很好地预测了 MDA,但它们确实有一定的局限性。近年来,自动编码器已被广泛应用于各个领域[33,34] ,以有效地学习 miRNA 和疾病的特征表示,而不丢失图形结构拓扑信息,我们提出了一种基于堆叠图自动编码器的 miRNA 疾病关联预测算法(SGAEMDA) ,如图1所示。然后将所有的 miRNA 特征与疾病特征连接起来作为 miRNA-疾病对特征。我们采用5折和10折的交叉验证来评估我们方法的预测性能。SGAEMDA 的5折和10折交叉验证的 AUC 分别为0.9585和0.9616,远远高于其他基线方法。此外,为了证明 SGAEMDA 的表现,我们对脑肿瘤、乳腺肿瘤、结肠肿瘤和肾肿瘤进行了个案研究。根据这些发现,我们预测的大部分可能的 miRNA-疾病关联被 dbDEMC 和 miRCancer 数据库验证。本文的重要贡献归纳如下。
(1)整合关联信息和相似性信息构建初始特征,可以更好地了解 miRNA-疾病对的潜在信息。
(2)提出了一种堆叠图自动编码器预测框架。与以往采用分层训练的堆叠自动编码器不同,堆叠图自动编码器采用多层协同无监督训练。它能够有效地从相似性网络中提取潜在的、深入的和未知的特征信息,以弥补以前模型预测结果的缺陷,这些预测结果偏向于具有已知关联的 miRNA 和疾病。
(3)我们使用一个多层感知机(MLP)来预测最终的结果,这个模型具有很高的容错性,能够快速有效地从 miRNA-disease 对中学习特征信息,从而提高模型的预测性能。
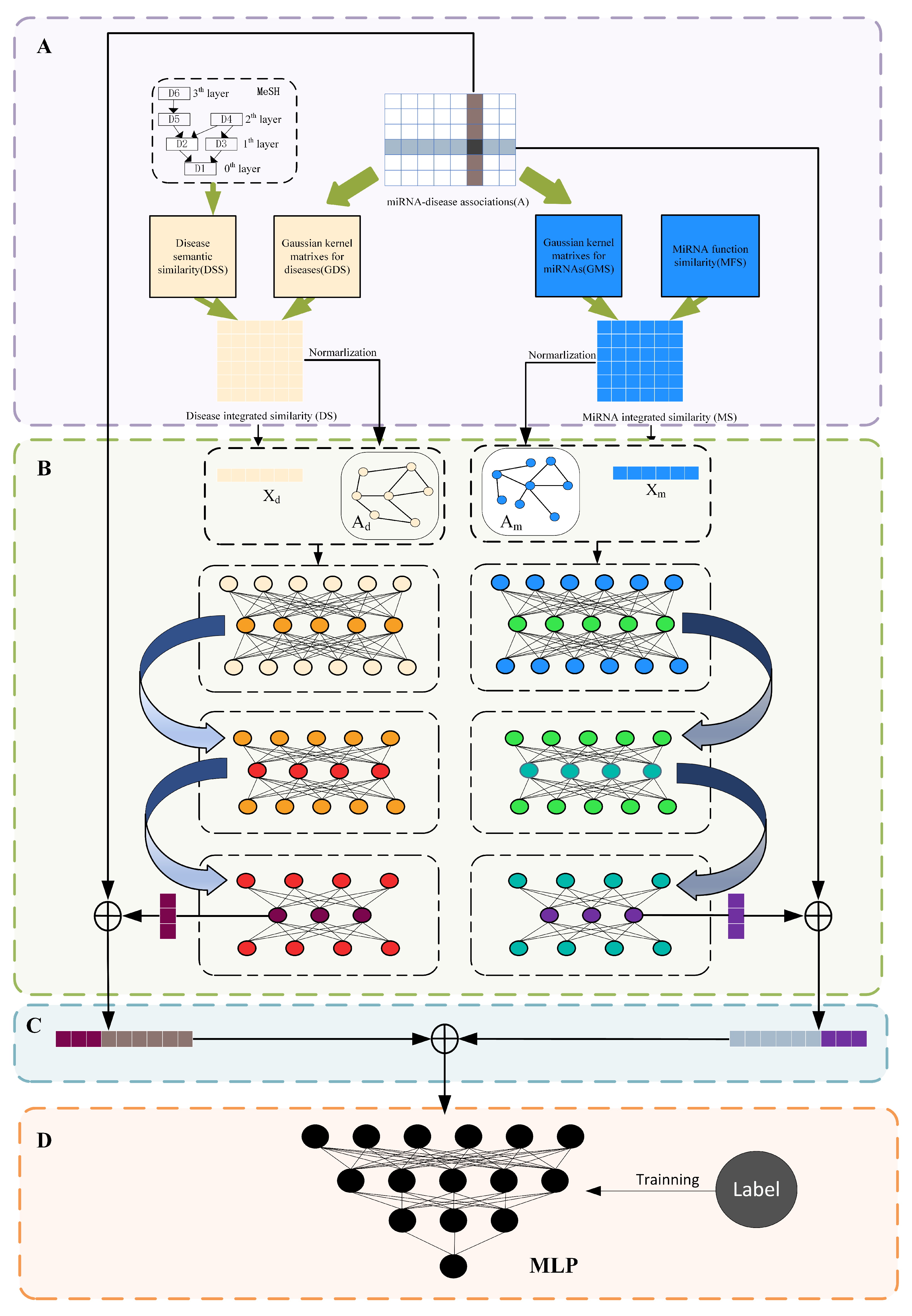
图1。SGAEMDA 流程图。(A)初始特征的建造和数据处理。(B)提取 miRNA 和疾病的低维相似性特征的预训练。(C)融合所学的 miRNA 和疾病特征以产生 miRNA-疾病对特征向量。(D) MLP 关联预测得分。
2. Materials and Methods
2.1.Datasets for MDA Prediction
HMDD v2.0
2.2.MiRNA and Disease Informaton
2.2.1.MiRNA Function Similarity
2.2.2.Disease Semantic Similarity
2.2.3.Gaussian Interaction Profile Kernel Similarity of miRNAs and Diseases
2.2.4.Integration of miRNAs and Diseases Similarity
2.3.SGAEMDA
为了预测 miRNA 与疾病的潜在关联,本研究提出了叠加图形自动编码器 miRNA-疾病关联预测模型(SGAEMDA)。为了成功提取相似网络中的潜在信息并预测 miRNA 与疾病的关联,该模型集成了一个基于图卷积网络的自动编码器和一个多层感知机。SGAEMDA 通常由以下步骤组成: (1)构造初始特征。(2)预训练堆叠图形自动编码器提取 miRNA 和疾病相似性潜在特征。(连接潜在特征和关联特征。(4)预测 miRNA 疾病。
(1)构建初始特征
我们从关联信息和相似性信息两个不同的角度构建了 miRNA 与疾病的初始特征。首先,对于 miRNA-疾病关联矩阵 A,每行可以看作是 miRNA 的关联特征,每列可以看作是疾病的关联特征。对于 miRNA 整合相似矩阵 Sm 和疾病整合相似矩阵 Sd,每行 Sm 可以看作是 miRNA 的相似特征,每行 Sd 可以看作是疾病的相似特征。具体而言,miRNA 和疾病的两个初始特征载体如下所示:
![]()
其中![]() ,当
,当 ![]() 时,
时,![]() 表示 miRNA 或疾病的关联特征,当
表示 miRNA 或疾病的关联特征,当 ![]() 时,
时,![]() 表示 miRNA 的功能相似性特征或疾病的语义相似性特征。Φ ∈{ m,d } ,φ = m 表示 miRNA 特征,φ = d 表示疾病特征,nm1,nd1,nm2,nd2分别表示 A 的列和行数,Sm 的列数和 Sd 的列数,即383,495,495和383。
表示 miRNA 的功能相似性特征或疾病的语义相似性特征。Φ ∈{ m,d } ,φ = m 表示 miRNA 特征,φ = d 表示疾病特征,nm1,nd1,nm2,nd2分别表示 A 的列和行数,Sm 的列数和 Sd 的列数,即383,495,495和383。
(2)预训练堆叠图自动编码器
参考以前的研究[39] ,图形自动编码器可以学习图形节点的低维特征表示来寻找合适的嵌入。由于 miRNA 与疾病的相似性信息是高维的,这可能会影响预测模型的预测精度。提出了一种基于堆叠图自动编码器的低维相似潜在特征提取方法,该方法比传统的图自动编码器具有更强的特征提取能力。图自动编码器由于其无监督的训练方法,特别适用于未标记数据量大、标记数据量小的数据集。具体地说,自动编码器的每一层的编码器和解码器定义如下:

其中 A、 Y、 W 表示节点的邻接矩阵、特征矩阵和可学习的参数矩阵。因此,可以通过上述编解码器结构来学习 miRNA,![]() 的特征表示:
的特征表示:
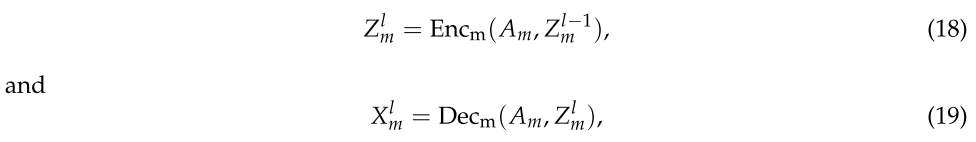
其中 l 表示图形自动编码器的层数,![]() ,
,![]() 表示由图形自动编码器的 lth 层学习的低维特征表示,当 l = 1,
表示由图形自动编码器的 lth 层学习的低维特征表示,当 l = 1,![]() 即
即![]() 表示由自动编码器的 lth 层重建的 miRNA 特征表示,
表示由自动编码器的 lth 层重建的 miRNA 特征表示,![]() 表示拉普拉斯正则化的 miRNA 邻接矩阵。公式如下:
表示拉普拉斯正则化的 miRNA 邻接矩阵。公式如下:
![]()
其中 ![]() 是 miRNA 整合相似矩阵
是 miRNA 整合相似矩阵 ![]() 的度矩阵。
的度矩阵。
类似地,我们通过相同结构的堆叠图自动编码器学习疾病的低维特征表示 ![]() ,如下所示:
,如下所示:
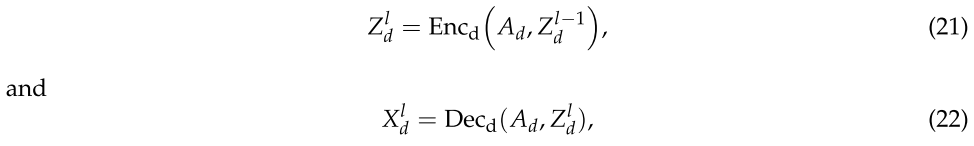
其中 ![]() 表示由 lth 层图自动编码器学习的低维特征表示,当 l = 1,
表示由 lth 层图自动编码器学习的低维特征表示,当 l = 1,![]() ,
,![]() 表示由 lth 自动编码器重建的疾病特征表示,
表示由 lth 自动编码器重建的疾病特征表示,![]() 表示疾病的拉普拉斯正则化化邻接矩阵。公式如下:
表示疾病的拉普拉斯正则化化邻接矩阵。公式如下:
![]()
在本研究中,SGAE 是由三个图自动编码器叠加而成,即 L = 3。具体地说,将所述第一图自动编码器生成的特征表示作为输入到所述第二图自动编码器中,所述第二图形自动编码器生成另一维度较低的特征表示,以此类推,直到构造出所述 L 图形自动编码器为止。基于重构损失函数协同训练多个图形自动编码器,生成 miRNA 和疾病 ![]() 的最终低维相似特征表示,其方程如下:
的最终低维相似特征表示,其方程如下:
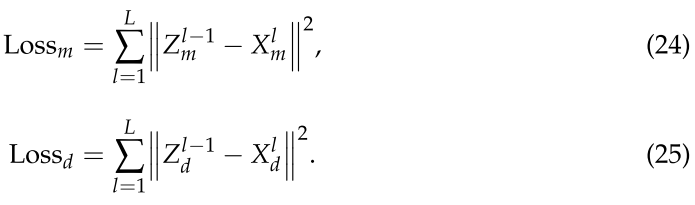
(3)连接潜在特征和关联特征
我们在预训练中将最终的嵌入维数设置为64,并且训练获得了所有 miRNA 和疾病的低维相似性表示,分别表示为 ![]() ,
,![]() 。为了在 miRNA 和疾病的特征表示中包括更多的潜在信息,我们将
。为了在 miRNA 和疾病的特征表示中包括更多的潜在信息,我们将 ![]() 和
和 ![]() 分别与 miRNA 的关联特征
分别与 miRNA 的关联特征 ![]() 和疾病的关联特征
和疾病的关联特征 ![]() 连接,最终获得447维 miRNA 嵌入和559维疾病嵌入,如下:
连接,最终获得447维 miRNA 嵌入和559维疾病嵌入,如下:
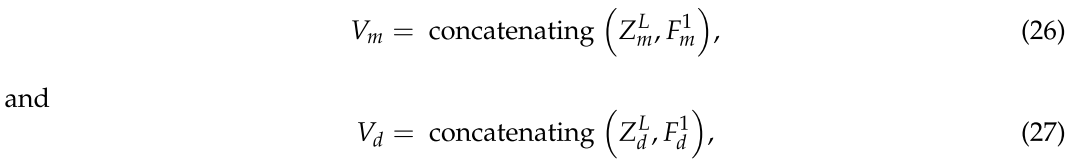
其中 ![]() 表示 miRNA 的最终嵌入,
表示 miRNA 的最终嵌入,![]() 表示疾病的最终嵌入。
表示疾病的最终嵌入。
(4)利用多层感知机预测 miRNA 与疾病的关联
在获得 miRNA 和疾病的嵌入之后,我们将每种 miRNA 的嵌入 ![]() 和每种疾病的
和每种疾病的 ![]() 连接起来形成完整的数据集 X,其中
连接起来形成完整的数据集 X,其中 ![]() ,如下:
,如下:
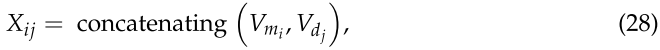
其中 ![]() 表示 miRNA
表示 miRNA ![]() 和疾病
和疾病 ![]() 的 miRNA-疾病对的特征。然后,我们使用多层感知机(MLP)对最终的 miRNA-疾病关联进行评分,以便进行预测,如下所示:
的 miRNA-疾病对的特征。然后,我们使用多层感知机(MLP)对最终的 miRNA-疾病关联进行评分,以便进行预测,如下所示:
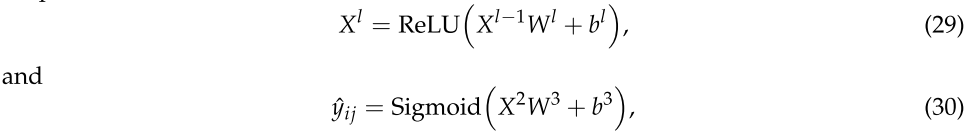
其中![]() 表示隐层的层数,
表示隐层的层数,![]() 表示 lth 隐层的输出,
表示 lth 隐层的输出,![]() 分别是 lth 隐层的可学习参数矩阵和偏差。
分别是 lth 隐层的可学习参数矩阵和偏差。![]() 是最终 miRNA-疾病对的预测得分。最后,通过最小化二元交叉熵损失函数的误差对模型进行训练:
是最终 miRNA-疾病对的预测得分。最后,通过最小化二元交叉熵损失函数的误差对模型进行训练:
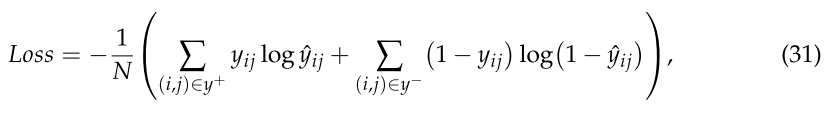
其中(i,j)表示 miRNA ![]() 和疾病
和疾病 ![]() 对。
对。![]() 子表表示正样本集和负样本集。N 表示阳性和阴性样本集中所有 miRNA 疾病对的数目。
子表表示正样本集和负样本集。N 表示阳性和阴性样本集中所有 miRNA 疾病对的数目。
3.Result
3.1.Experiment Details
在本实验中,我们基于 pytorch 框架和 scikit-learning 框架实现了 SGAEMDA 模型。在预训练和 MLP 训练过程中,采用 Adam 优化算法使损失函数最小。由于 HMDDv2.0数据库中阳性和阴性样本的显着不平衡,已知的 miRNA 疾病关联的数量为5430(阳性样本) ,其余的184,155对是未知的关联(阴性样品) ,阴性样本的数量约为阳性样品的34倍。为了使模型具有较好的鲁棒性,我们随机选取与正样本相等的负样本进行 MLP 训练,并在随后的实验中随机选取10次,以保证实验的可靠性。我们的 HSSG 源代码可在网上查阅: https://github.com/lynn0424/sgaemda (于2022年12月5日获取)。
3.2.Evaluation Metrics
3.3.Prediction of miRNA–Disease Association Based on SGAEMDA
3.4.Effect of Similarity Feature Dimensions
3.5.Effect of Stacked Graph Autoencoder Pre-Training
3.6.Comparison of Different Classifier Models
3.7.Comparisons with Existing SOTA Methods
3.8.Case Studies
4.Discussion
在过去,许多研究表明,异常的 miRNA 表达往往与许多生物学过程以及复杂的疾病的发生有关,在人类中具有相当大的影响。因此,预测潜在的 miRNA-疾病关联可以帮助医学专业人员提供分子视角洞察各种复杂疾病的发病机制,从而开发相关的新药。本文提出了一种基于堆叠图自动编码器的 SGAEMDA 模型。与以往的堆叠式自动编码器不同,SGAE 不是一层一层地训练,而是与每一层协作,这弥补了以往堆叠式自动编码器贪婪训练导致编码能力较弱的缺点。它可以从更深层次的 miRNA 相似性网络和疾病相似性网络中提取潜在的特征表示。将提取的特征与相应的关联特征连接,利用 MLP 方法预测 miRNA 与疾病的关联。实验结果显示,SGAEMDA 的 AUC 值最高,在5折和10折的交叉验证下达到0.9585。比其他基线方法要高得多。案例研究分析实验证实,我们的模型可以有效地预测潜在的 miRNA 疾病的关联。然而,我们的工作仍有一些地方需要改进:
(1)该模型没有经过端到端训练,鲁棒性较差。
(2)实验中使用的数据较少,无法从更多的角度提取更多关于 miRNA 和疾病的信息。
在未来的研究中,我们将融合更多的 miRNA 和疾病相似性信息,以进一步提高我们的预测模型的性能。此外,我们将利用类似于 EGES 模型[47]的方案来允许嵌入覆盖更多的 miRAN 和疾病,从而解决遗传疾病关联预测中的冷启动问题。