Deep-belief network for predicting potential miRNA-disease associationsDeep-belief network for predicting potential miRNA-disease associations | Briefings in Bioinformatics | Oxford AcademicAbstract. MicroRNA (miRNA) plays an important role in the occurrence, development, diagnosis and treatment of diseases. More and more researchers begin to pay a![]() https://academic.oup.com/bib/article/22/3/bbaa186/5898648?login=true
https://academic.oup.com/bib/article/22/3/bbaa186/5898648?login=true


Abstract
小 RNA (miRNA)在疾病的发生、发展、诊断和治疗中起着重要作用。越来越多的研究者开始关注 miRNA 与疾病之间的关系。与传统的生物学实验相比,利用计算方法整合异质生物学数据来预测潜在的相关性,可以有效地节省时间和成本。考虑到以往计算模型的局限性,我们建立了 miRNA-疾病关联预测的深度信念网络模型(DBNMDA)。我们构建了特征向量来预先训练所有 miRNA 疾病对的受限 Boltzmann 机,并应用阳性样本和相同数量的选定阴性样本来微调 DBN 以获得最终预测分数。与以往的监督模型相比,DBNMDA 只使用具有已知标签的对进行训练,在预训练过程中创新地利用了所有 miRNA 疾病对的信息。这一步骤可在一定程度上减少已知关联过少对预测准确性的影响。基于全局留一交叉验证(LOOCV) ,DBNMDA 达到0.9104的 AUC,基于局部 LOOCV 的 AUC 为0.8232,基于5倍交叉验证的平均 AUC 为0.9048 ± 0.0026。这些 AUC 比以前的其他型号更好。此外,三种不同类型的病例研究实施三种疾病,以证明 DBNMDA 的准确性。结果,前50位预测的 miRNA 中,84% (乳腺肿瘤)、100% (肺部肿瘤)和88% (食管肿瘤)得到了最新文献的验证。因此,我们可以得出结论,DBNMDA 是一种有效的方法来预测潜在的 miRNA 疾病的关联。
关键词: microRNA; disease; association prediction; deep-belief network; unsupervised pre-training; supervised fine-tuning
目录
1.引言
以上模型各有优势,但不可否认的是,这些模型也有其局限性,需要进一步完善。近年来,深度学习在需要高度抽象特征的任务中取得了良好的效果,如图像和语音识别[35]。现在,我们考虑应用深度学习来预测 miRNA 与疾病的关联。然而,在一般的多层感知机网络中,只能使用标签样本来训练网络。因此,我们提出了一个用于 MiRNA-disease 关联预测的深度信念网络模型(DBNMDA)。我们获得了所有 miRNA 疾病对的特征,并使用这些特征来预训练受限制的 Boltzmann 机器(RBM)。在 RBM 的预训练之后,我们保存了模型中的参数。然后,通过向 RBM 添加一个输出层来构建 DBN。最后,在预训练得到的参数的基础上,采用正样本和相同数量的随机选择的负样本对 DBN 进行微调。总之,DBNMDA 的训练过程可以分为两个过程: 贪婪分层无监督训练过程和监督微调过程。传统的机器学习方法利用正样本和负样本来训练预测模型。然而,在我们的数据集中,未标记的 miRNA 疾病样本的数量是非常大的。DBNMDA 的主要创新之处在于它能够通过引入无监督的预训练过程来利用所有 miRNA-disease 对的信息。此外,DBNMDA 采用有监督的微调过程,进一步提高了预测模型的准确性。在本研究中,我们采用不同的评估指标来验证模型的准确性。因此,DBNMDA 在全局留一交叉验证(LOOCV)中的 AUC 为0.9104,局部 LOOCV 中的 AUC 为0.8232。在5倍交叉验证中,平均 AUC 和标准差分别为0.9048和0.0026。此外,我们利用 DBNMDA 进行个案研究,进一步验证 DBNMDA 的预测能力。在三种不同类型的病例研究中,分别选择乳腺肿瘤(BN)、肺肿瘤(LN)和食管肿瘤(EN)作为研究疾病。结果表明,前50个预测的可能相关的 miRNA 中有42,50,44个已被数据库验证。
2.结果
2.1.预测性能评价
2.2.案例研究
3.讨论
对潜在疾病与 miRNA 关联的预测可以帮助我们了解疾病的发生机制,更好地治疗疾病。在本文中,我们开发了一个 DBNMDA 模型。DBNMDA 的实现过程可以分为两个部分。第一部分是构建特征向量,用于所有 miRNA 疾病对的 RBM 预训练。第二部分是用正样本和相同数量选择的负样本对 DBN 进行微调。在全局 LOOCV 和局部 LOOCV 中,DBNMDA 模型均表现出较高的性能,是全局 LOOCV 中 AUC 大于0.9的少数模型之一。另外,5折交叉验证的结果反映了 DBNMDA 的稳定性。此外,三个实例也验证了 DBNMDA 的准确预测性能。
DBNMDA 在以下方面表现良好。首先,我们将各种相似度信息作为特征进行集成。其次,DBNMDA 创造性地将无监督预训练与监督微调相结合。在我们的研究中,已知的关联对只占所有关联对的一小部分。预训练过程使模型能够了解所有配对的特征,这弥补了传统监督式学习只能训练标签样本的缺陷。因此,DBNMDA 适合我们的数据。最后,DBN 的微调过程是基于预训练得到的模型参数进行的,因此 DBN 可以使深度神经网络输入层附近的权值得到充分的训练。
然而,我们的模型也有一些局限性。首先,我们直接剪接 miRNA 与疾病的相似性作为 miRNA-疾病对特征。然而,这种方式并不是很容易理解。其次,在有监督的微调过程中,我们同时使用了正样本和负样本。然而,阴性样品是从未标记的样品中随机选择的,其中可能包括假阴性样品。因此,在 DBNMDA 中使用的阴性样本不是很可靠,这将影响 DBNMDA 的预测准确性。我们试图通过引入可靠的阴性样品的测量来改进它。最后,对于神经网络的超参数选择问题,如隐层数、每层神经元数等,目前还没有明确的解释。因此,获得多种类型的 miRNA 特征和疾病特征,并选择更可靠的阴性样本是今后的一项重要任务。此外,由于卷积网络已经引起了许多研究者的关注,在 DBNMDA 中可以考虑用卷积 RBM 来构建 DBN 模型。
4.材料和方法
4.1.材料和方法
HMDD v2.0


4.2.DBNMDA
在这项研究中,我们开发了一个名为 DBNMDA 的模型来预测潜在的 miRNA 疾病相关性。DBNMDA 的流程图如图2所示。经过数据收集和相似性计算,我们得到了 miRNA 疾病对的邻接矩阵 A (495 × 383)、整合 miRNA 相似性矩阵 SM (495 × 495)和整合疾病相似性矩阵 SD (383 × 383)。我们建立的预测模型基于这样的假设: 相似的 miRNA 往往与相似的疾病相关,反之亦然。因此,我们利用疾病相似性(miRNA 相似性)来表示疾病(miRNA)作为特征向量。从 miRNA 相似性矩阵和疾病相似性矩阵中,分别提取出每种 miRNA 和每种疾病的495和383个特征。具体而言,矩阵 SM 的第 i 行的495个元素被认为是 miRNA m (i)的495个特征,并且矩阵 SD 的第 j 行的383个元素被认为是疾病 d (j)的383个特征。将所研究疾病的特征向量和每个 miRNA 连接起来,每个 miRNA-疾病对产生878个特征。

接下来,我们构建了一个由 RBMs 组成的 DBN 模型。作为 DBN 的基本模块,RBM 主要由可见层和隐藏层组成。每个可见层单元连接到所有隐藏层单元,这被称为完全连接。隐层和可见层的神经元之间没有联系。RBM 是一个能量模型,其参数由权重和偏差组成。我们假设 RBM 的可见变量向量和隐藏变量向量分别是 v 和 h。RBM的能量定义为:

其中 ![]() 。W 是表示连接隐层和可见层单元之间权重的矩阵。b 和 c 分别代表可见层和隐藏层的偏差。基于
。W 是表示连接隐层和可见层单元之间权重的矩阵。b 和 c 分别代表可见层和隐藏层的偏差。基于 ![]() ,
,![]() 的联合概率分布可以定义如下:
的联合概率分布可以定义如下:
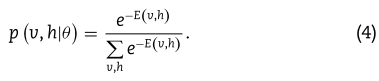
通过对所有可能的隐含向量求和,给出了可见层的边缘概率分布。

每个样本的分布经过RBM预测模型处理后,从可见层进行重构和输出。为了使样本的重构分布近似于样本的原始分布,我们将损失函数定义为负对数似然函数。

其中 T 是一组样本,它们是从所有对中随机选择的,用于随机梯度下降(SGD)。在参数调整中采用了 SGD 算法。
DBN 由多个堆叠的 RBM 和一个连接到最后一个 RBM 的输出层组成。DBN 的训练程序可分为两部分,即逐层贪婪无监督预训练和监督微调过程。贪婪的无监督预训练过程如下所示。首先,随机初始化参数 W,b,c。其次,我们将第一层和第二层作为 RBM,并将获得的特征值作为原始输入输入到可见层。第三,我们把第二层和第三层作为RBM,它的输入是以前RBM的输出。最终,我们迭代所需的层数。在预训练过程中,我们使用所有 miRNA 疾病对的特征进行预训练。监督微调过程如下。我们首先使用最后一层的输出作为 Logit模型分类器(LR)的输入。然后,我们将已知的关联作为正样本,并随机选择相同数量的未知对作为负样本。阳性样品的标签设置为1,阴性样品的标签设置为0。接下来,利用标记的正样本和负样本,我们通过 SGD 对所有 RBM 和 LR 的参数进行了微调。最后,我们可以通过将其特征向量输入到训练好的 DBN 模型中来获得候选 miRNA-疾病对的关联分数。