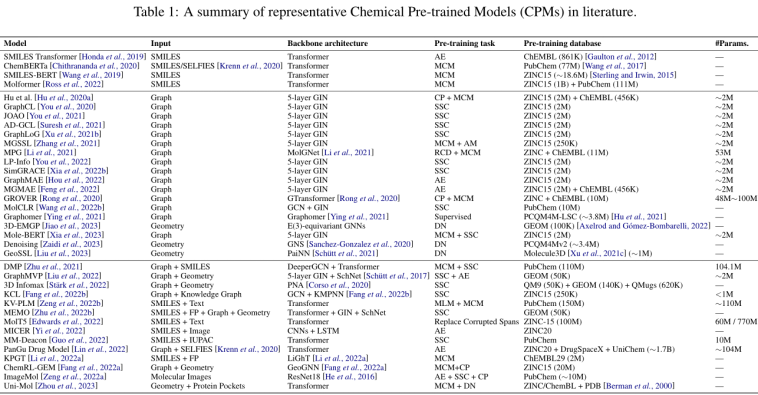
IJCAI_A Systematic Survey of Chemical Pre-trained Models
综述资料汇总(更新中,原文提供):GitHub - junxia97/awesome-pretrain-on-molecules: [IJCAI 2023 survey track]A curated list of resources for chemical pre-trained models
从分子描述符、编码器架构、预训练策略和应用描述文章。
直接使用DNN的问题:(1) 标记数据的稀缺性:分子的任务特异性标记可能极其稀缺,因为分子数据标记通常需要昂贵的湿实验室实验;(2) 分布外泛化能力差:在许多现实世界的情况下,学习具有不同大小或官能团的分子需要分布外泛化。
因此更加关注化学预训练模型(CMPs)【以从大量未标记的分子中学习通用分子表示,然后在特定的下游任务中进行微调】
一、Molecular Descriptors and Encoders
Fingerprints (FP). 描述具有二元串的分子的特定子结构的存在或不存在。
Sequences. 最常用的SMILES,
2D graphs. 原子作为节点,键作为边。
3D graphs. 表示分子的原子在3D空间中的空间排列,其中每个原子与其类型和坐标以及一些可选的几何属性(如速度)相关联。使用3D几何的优点是构象信息对许多分子性质,特别是量子性质至关重要。此外,在给定3D几何形状的情况下,还可以直接利用立体化学信息,例如手性。
二、Pre-training Strategies


1、AutoEncoding (AE) 图3a
【SMILES Transformer: Pre-trained Molecular Fingerprint for Low Data Drug Discovery.】:利用了基于转换器的编码器-解码器网络,并通过重建SMILES字符串表示的分子来学习表示。
【PanGu Drug Model: Learn a Molecule Like a Human. 】:预训练图来对不对称的条件变分自动编码器进行排序,以学习分子表示。
尽管自动编码器可以学习分子的有意义表示,但它们只关注单个分子,无法捕捉分子间的关系,这限制了它们在一些下游任务中的性能。(不是主流方法)
2、Autoregressive Modeling (AM) 图3b
将分子输入分解为子序列(通常为原子level结构),然后根据序列中前面的子序列逐个预测后面子序列。公式:

【MolGPT: Molecular Generation Using a Transformer-Decoder Model.2021】:使用transformer decoder,自回归方式预测SMILES字符串中的下一个token。
【GPT-GNN: Generative Pre-Training of Graph Neural Networks.KDD, 2020.】:重建分子图(图3b),给定一个节点和边被随机mask的graph,一次生成一个masked节点和边,并使每次迭代中生成的节点和边的可能性最大。然后,迭代地生成节点和边,直到生成所有masked节点为止。
【Motif-based Graph Self-Supervised Learning for Molecular Property Prediction. NeurIPS, 2021.】:生成分子图片段(子图),而不是单个原子或自回归键。
AM在生成分子表现更好。然而,AM在计算上更昂贵,并且需要预先设置原子或键的顺序,这可能不适合分子,因为原子或键不存在固有的顺序。
3、Masked Component Modeling (MCM) 图3c
MCM掩盖了分子的一些组成(如原子、键和片段),然后训练模型,在给定剩余成分的情况下预测它们。公式:

Sequence-base model:
【ChemBERTa: Large-Scale Self-Supervised Pretraining for Molecular Property Prediction. 2020】、【SMILES-BERT: Large Scale Unsupervised Pre-Training for Molecular Property Prediction. BCB, 2019.】、【Molformer: Large Scale Chemical Language Representations Capture Molecular Structure and Properties. Nat. Mach. Intell., 2022】:屏蔽SMILES strings中的随机字符,然后基于un-masked的SMILES string的transformer的输出来恢复它们。
Graph-based model:
【Strategies for Pre-training Graph Neural Networks. In ICLR, 2020.】:随机屏蔽输入的原子/化学键属性,并预训练GNN来预测它们
【Self-Supervised Graph Transformer on Large-Scale Molecular Data. NeurIPS, 2020.】:试图预测masked子图,以捕捉分子图中的上下文信息
【Mole-BERT: Rethinking Pre-training Graph Neural Networks for Molecules. ICLR,
2023】:由于自然界中的原子集极小且不平衡,掩盖原子类型可能会有问题。为了缓解这个问题,设计了一个上下文感知的tokenizer,将原子编码为具有化学意义的离散值,用于mask
基于其surrounding environments来预测masked components,而AM仅依赖于预定义序列中的先前分量。因此,MCM可以捕获更完整的化学语义。然而,由于MCM在BERT后的预训练过程中经常掩盖每个分子的固定部分,因此它不能对每个分子中的所有成分进行训练,这导致样本利用效率较低。
4、Context Prediction (CP) 图3d
以明确的、上下文感知的方式捕捉分子/原子的语义。

【Strategies for Pre-training Graph Neural Networks. ICLR, 2020.】:使用二元分类来判断分子和周围上下文结构中的子图是否属于同一节点。
虽然CP简单有效,但需要一个辅助神经模型将上下文编码为固定向量,从而为大规模预训练增加了额外的计算开销。
5、Contrastive Learning (CL) 图3e
最大化一对相似输入之间的一致性来预训练模型,例如同一分子的两个不同增强或描述符。
根据对比粒度(例如,molecule- or substructure-level),两类CL:Cross-Scale Contrast (CSC) and Same-Scale Contrast (SSC)
1)Cross-Scale Contrast (CSC)
【InfoGraph: Unsupervised and Semi-supervised Graph-Level Representation Learning via Mutual Information Maximization. ICLR, 2020.】:通过互信息最大化的无监督和半监督图级表示学习。
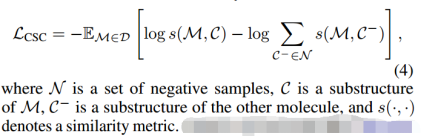
【Contrastive Multi-View Representation Learning on Graphs. ICML,2020.】:执行节点扩散以生成增广分子图,然后通过将一个视图的原子表示与另一个视图(反之亦然)的分子表示进行对比,最大限度地提高原始视图和增广视图之间的相似性。
2)Same-Scale Contrast (SSC).
通过将增强分子推向anchor分子(正对)和远离其他分子(负对),对单个分子进行对比学习。公式:

Graph-level的分子级预训练(各种增强策略)对比学习:
【Graph Contrastive Learning with Augmentations. In NeurIPS, 2020】、
【Graph Contrastive Learning Automated. In ICML, 2021.】
【MoCL: Contrastive Learning on Molecular Graphs with Multi-level Domain Knowledge. In
KDD, 2021.】
【Adversarial Graph Augmentation to Improve Graph Contrastive Learning. In NeurIPS,
2021】
【InfoGCL: InformationAware Graph Contrastive Learning. In NeurIPS, 2021.】
【Molecular Graph Contrastive Learning with Parameterized Explainable Augmentations.
In BIBM, 2021.】
【Molecular Contrastive Learning with Chemical Element Knowledge Graph. In AAAI,2022.】
【Improving Molecular Contrastive Learning via Faulty Negative Mitigation and Decomposed Fragment Contrast. J. Chem. Inf. Model., 2022.】
【SimGRACE: A Simple Framework for Graph Contrastive Learning without Data Augmentation. In WWW, 2022.】
最大限度地提高了相同分子的各种描述符之间的一致性,并排斥了不同的描述符:
【SMICLR: Contrastive Learning on Multiple Molecular Representations for Semisupervised and Unsupervised Representation Learning. J.Chem. Inf. Model., 2022.】:联合graph encoder 和SMILES string encoder
【Multilingual Molecular Representation Learning via Contrastive Pre-training. In ACL,2022.】:MM Deacon利用两个独立的transformers来编码SMILES和IUPAC,使用对比目标来促进同一分子的SMILES与IUPAC相似性
【3D Infomax Improves GNNs for Molecular Property Prediction. In ICML, 2022】:最大化所学习的3D geometry and 2D graph 之间的一致性;
【GeomGCL: Geometric Graph Contrastive Learning for Molecular Property Prediction. In AAAI, 2022.】:采用双视图几何消息传递神经网络(GeomMPNN)对分子的2D和3D图进行编码,并设计geometric contrastive目标,
一些关键问题阻碍了其更广泛的应用,在分子增强过程中很难保留语义。通过手动试验和错误来选择增强【Graph Contrastive Learning with Augmentations. In NeurIPS, 2020.】,繁琐的优化【Graph Contrastive Learning Automated. In ICML, 2021.】,或通过昂贵的领域知识指导【MoCL: Contrastive Learning on Molecular Graphs with Multi-level Domain Knowledge. In KDD, 2021.】,但仍然缺乏一种有效且原则性的方法来设计适合于分子预训练的化学增强。
CL背后拉近相似表征的假设对于分子表征学习来说可能并不总是成立的(分子活性悬崖:类似的分子具有完全不同的性质)。
CL目标随机选择一批中的所有其他分子作为阴性样本,而不管它们的真实语义如何,这将不期望地排斥具有相似性质的分子,并由于假阴性而破坏性能【ProGCL: Rethinking Hard Negative Mining in Graph Contrastive Learning. In ICML, 2022.】
6、Replaced Components Detection (RCD) 图3f
识别输入分子的随机替换成分。例如,【MPG:An Effective Self-Supervised Framework for Learning Expressive Molecular Global Representations to Drug Discovery. Briefings Bioinform 2021.】将每个分子分成两部分,通过组合两个分子的部分来改变其结构,并训练编码器检测组合的部分是否属于同一分子。

虽然RCD可以揭示分子结构中的内在模式,但编码器经过预训练,始终为所有天然分子产生相同的“非替换”标签,为随机组合的分子产生相同“替换”标签。然而,在下游任务中,输入分子都是天然分子,导致RCD产生的分子表征不太可区分。
7、DeNoising (DN) 图3g
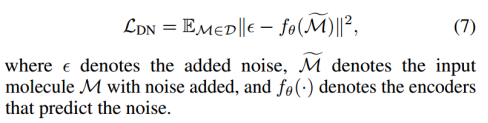
【Pre-training via Denoising for Molecular Property Prediction. ICLR, 2023.】:将噪声添加到3D分子几何的原子坐标中,并预训练编码器来预测噪声。
【Uni-Mol: A Universal 3D Molecular Representation Learning Framework. In ICLR, 2023.】:为原子坐标添加了噪声,其动机是在给定3D原子位置的情况下,可以很容易地推断出掩蔽的原子类型。
【Molecular Geometry Pretraining with SE(3)-Invariant Denoising Distance Matching. In ICLR, 2023.】:距离去噪预训练,以对3D分子的动态特性进行建模。
三、Extensions
1、Knowledge-enriched pre-training.
CPM通常从大型分子数据库中学习一般的分子表示。然而,他们往往缺乏特定领域的知识。
【GraphCL:Graph Contrastive Learning with Augmentations. In NeurIPS, 2020.】:指出键扰动(添加或删除键作为数据增强)在概念上与领域知识不兼容,并且在经验上对化合物的对比预训练没有帮助。因此,他们避免采用键扰动来增强分子图。
【MoCL: Contrastive Learning on Molecular Graphs with Multi-level Domain Knowledge. In KDD, 2021.】提出了一种基于领域的分子增强算子,称为子结构替换,其中分子的有效子结构被生物异构体所取代,生物异构体产生的新分子具有与原始分子类似的物理或化学性质。
【Molecular Contrastive Learning with Chemical Element Knowledge Graph. In AAAI, 2022.】:构建了一个化学元素知识图(KG)来总结化学元素之间的微观关联,并提出了一个新的用于分子表征学习的知识增强对比学习(KCL)框架。
【Motif-based Graph Self-Supervised Learning for Molecular Property Prediction. In NeurIPS, 2021.】:首先利用现有算法(On the Art of Compiling and Using ‘Drug-Like’ Chemical Fragment Spaces. ChemMedChem, 2008.)提取语义上有意义的基序,然后预处理神经编码器,以自回归的方式预测基序。
【Geometry-Enhanced Molecular Representation Learning for Property Prediction. Nat. Mach. Intell. 2022.】:提出利用分子几何信息来增强分子图预训练。设计了一种基于几何的GNN体系结构以及几种几何级的自监督学习策略(键长预测、键角预测和原子距离矩阵预测),以在预训练过程中获取分子几何知识。
尽管知识丰富(注入知识)的预训练有助于CPM获取化学领域知识,但它需要昂贵(计算代价、数据代价)的先验知识作为指导,当先验知识不完整、不正确或获取成本高昂时,这限制了更广泛的应用。
2、Multimodal pre-training.
使用包括图像和生物化学文本等。
【KV-PLM:A Deep-Learning System Bridging Molecule Structure and Biomedical Text with Comprehension Comparable to Human Professionals. Nat. Commun.,2022】:标记SMILES字符串和生物化学文本。然后,随机屏蔽部分令牌,并预训练神经编码器以恢复屏蔽的令牌。
【MolT5:Translation between Molecules and Natural Language. In EMNLP, 2022.】:mask大量SMILES字符串的一些跨度和分子的生物化学文本描述,然后预训练转换器来预测掩盖的跨度。通过这种方式,这些预先训练的模型可以生成SMILES字符串和生物化学文本,这对于文本引导的分子生成和分子字幕(生成分子的描述性文本)特别有效。
【Featurizations Matter: A Multiview Contrastive Learning Approach to Molecular Pretraining. In AI4Science@ICML, 2022.】:提出使用对比目标最大化四个分子描述符的嵌入与其聚合嵌入之间的一致性。通过这种方式,这些不同的描述符可以相互协作用于分子性质预测任务。
【MICER: A Pre-Trained Encoder–Decoder Architecture for Molecular Image Captioning. Bioinform., 2022.】:采用了一种基于自动编码器的预训练框架来进行分子图像字幕。将分子图像馈送到预先训练的编码器,然后解码相应的SMILES字符串。
上述多模式预训练策略可以促进各种模式之间的翻译。此外,这些模式可以协同工作,为各种下游任务创建更完整的知识库
四、Applications
1、Molecular Property Prediction (MPP)
标签可能非常稀缺,因为湿实验室实验通常既费力又昂贵。CPM利用大量未标记的分子,并作为下游分子性质预测任务的backbone。
与从头开始训练的模型相比,CPM可以更好地推断出分布外的分子【Strategies for Pre-training Graph Neural Networks. In ICLR, 2020.】
2、Molecular Generation (MG)
1)类似于MolGPT自回归在SMILES上生成。
2)通过将描述性文本转化为分子结构,进一步扩大了分子生成的可能性。
3)三维分子构象的产生,特别是用于预测蛋白质配体结合姿势。但是受计算限制。基于3D几何的CPM在构象生成任务中表现出显著的优势,因为可以在预训练过程中捕捉2D分子和3D构象之间的一些固有关系。
3、Drug-Target Interactions (DTI)
药物靶标相互作用预测(DTI)
4、Drug-Drug Interactions (DDI)
药物相互作用预测(DDI),相互作用可能导致不良反应,从而损害健康,甚至导致死亡。
五、Conclusions and Future Outlooks
1、Improving Encoder Architectures and Pre-training Objectives
CPM的理想功能和架构仍然难以捉摸,预训练目标仍有很大的改进空间,MCM中子组件的有效掩蔽策略就是一个主要例子。
2、Building Reliable and Realistic Benchmarks
尽管对CPM进行了大量研究,但由于所使用的评估设置不一致(例如,随机种子和数据集分割),它们的实验结果有时可能不可靠。例如,在包含几个用于分子性质预测的昂贵数据集的MoleculeNet上,同一模型的性能可能因不同的随机种子而显著不同,这可能是由于这些分子数据集的规模相对较小。同样至关重要的是,要为CPM建立更可靠、更现实的基准,将分布外的泛化考虑在内。一种解决方案是通过scafold split来评估CPM,这涉及到基于分子的亚结构来分裂分子。事实上,研究人员必须经常将从已知分子训练的CPM应用于新合成的未知分子,这些分子可能在性质上有很大差异,属于不同的结构域。
3、Broadening the Impact of Chemical Pre-trained Models
CPMs研究的最终目标是开发多功能分子编码器,可应用于大量与分子相关的下游任务。尽管如此,与NLP社区中PLM(pretraining language model)的进步相比,CPM的方法进步和实际应用之间仍然存在很大差距。一方面,CPM产生的表示尚未被广泛用于取代化学中的传统分子描述符,并且预先训练的模型尚未成为社区的标准工具。另一方面,对于这些模型如何使单个分子之外的更广泛的下游任务受益,如化学反应预测、虚拟筛选中的分子相似性搜索、逆转录合成、化学空间探索等,目前的探索有限。
4、Establishing Theoretical Foundations
CPM在各种下游任务中表现出了令人印象深刻的性能,但对这些模型的严格理论理解仍然有限。这种基础的缺乏阻碍了科学界和行业利益相关者寻求最大限度地发挥这些模型的潜力。必须建立CPM的理论基础,以便充分理解它们的机制以及它们如何在各种应用中提高性能。有必要进行进一步的研究,以更深入地了解不同分子预训练目标的有效性,从而为优化方法设计提供指导。