ABSTRACT
最新的联合模型通常依赖于外部自然语言处理(NLP)工具,性能好坏取决于工具在该领域的好坏
我们提出了一种神经网络,端到端模型,用于联合提取实体及其关系,该模型不依赖外部NLP工具,并且集成了一个大型的,经过预先训练的语言模型。由于我们模型的大部分参数都是经过预先训练的,并且避免重复进行自我关注,因此我们的模型可以快速进行训练。
INTRODUCTION
In the biomedical domain, NER and RE facilitate large-scale biomedical data analysis, such as network biology (Zhou et al., 2014), gene prioritization (Aerts et al., 2006), drug repositioning (Wang & Zhang, 2013) and the creation of curated databases (Li et al., 2015)
在生物医学领域,NER和RE可促进大规模生物医学数据分析,例如网络生物学(Zhou等人,2014),基因优先级排序(Aerts等人,2006),药物重新定位(Wang&Zhang,2013)和创建策展的数据库(Li等,2015)。
最常见的是,将NER和RE的任务作为管道来处理,而NER在RE之前。这种方法有两个主要缺点:(1)管道系统易于在NER和RE系统之间传播错误。 (2)一个任务无法利用另一任务的有用信息(例如,由RE系统标识的关系类型对于NER系统确定关系中涉及的实体类型可能有用,反之亦然)。最近,人们提出了一种同时学习提取实体和关系的联合模型,从而减轻了上述问题并实现了最先进的性能
最新联合抽取NER and RE进展
(2018a) proposes a neural, end-to-end system that jointly learns to extract entities and relations without relying on external NLP tools. In Bekoulis et al. (2018b), they augment this model with adversarial training. Nguyen & Verspoor (2019) propose a different, albeit similar end-to-end neural model which makes use of deep biaffine attention (Dozat & Manning, 2016). Li et al. (2019) approach the problem with multi-turn question answering, posing templated queries to a BERT-based QA model (Devlin et al., 2018) whose answers constitute extracted entities and their relations and achieve state-of-the-art results on three popular benchmark datasets.(2018a)提出了一种神经网络,端到端系统,该系统可以共同学习提取实体和关系,而无需依赖外部NLP工具。在Bekoulis等人中。 (2018b),他们通过对抗训练增强了该模型。 Nguyen&Verspoor(2019)提出了一个不同的端对端神经模型,尽管该模型利用了深的仿射流注意力(Dozat&Manning,2016)。 Li等。 (2019)通过多回合问题解答来解决该问题,将模板化查询呈现给基于BERT的QA模型(Devlin等人,2018),该模型的答案构成了提取的实体及其关系,并获得了最新的成果。三个流行的基准数据集。
end-to-end系统两个主要缺点
首先,是大多数模型参数都是从头开始训练的。对于大型数据集,这可能会导致较长的训练时间。For small datasets, which are common in the biomedical and clinical domains where it is particularly challenging to acquire labelled data, this can lead to poor performance and/or overfitting. 对于在生物医学和临床领域中很常见的小型数据集,在这些领域中获取标记数据特别具有挑战性,这可能导致性能不佳和/或过度拟合。
第二,这些系统通常包含RNN,这些RNN本质上是顺序的,不能在训练示例中并行化。
解决
Li等人提出的多通道QA模型。 (2019年)通过引入预训练的语言模型BERT(Devlin等人,2018年)缓解了这些问题,该模型避免了自我注意力的复用。他们方法的主要局限性在于它依靠手工制作的问题模板来获得最佳性能:
This may become a limiting factor where domain expertise is required to craft such questions (e.g., for biomedical or clinical corpora). Additionally, one has to create a question template for each entity and relation type of interest.在可能需要领域专业知识来提出此类问题的地方(例如,对于生物医学或临床语料库),这可能成为限制因素。另外,必须为每个感兴趣的实体和关系类型创建一个问题模板。
本篇论文贡献
- NER and RE基于端到端模型联合抽取
- 基于BERT模型微调,速度快
THE MODEL

NER部分由BERT实现
1.BERT模型产生下述序列:
![]()
2.送入全连接层并计算交叉熵损失,其中标签由BIOES表示
![]()
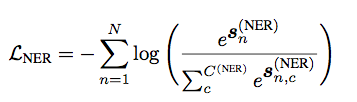
RE部分:
1.预测的标签被embedding,产生下述序列:
![]()
2.concat操作
![]()
3.

对应于第i个字用作关系的开头或结尾参数。
4.
Following Miwa & Bansal (2016) and Nguyen & Verspoor (2019), we incrementally construct the set of relation candidates, R, using all possible combinations of the last word tokens of predicted entities, i.e. words with E- or S- labels. An entity pair is assigned to a negative relation class (NEG) when the pair has no relation or when the predicted entities are not correct. Once relation candidates are constructed, classification is performed with a deep bilinear attention mechanism (Dozat & Manning, 2016), as proposed by Nguyen & Verspoor (2019).
继Miwa&Bansal(2016)和Nguyen&Verspoor(2019)之后,我们使用预测实体的最后单词标记的所有可能组合(即带有E-或S-标签的单词)逐步构建关系候选集R. 当实体对没有关联或预测的实体不正确时,会将实体对分配给负关联类(NEG)。 一旦建立了候选关系,就按照Nguyen和Verspoor(2019)提出的深双线性注意机制(Dozat&Manning,2016)进行分类。

5.为RE目标计算第二个交叉熵损失

6.端到端训练减少总loss

ENTITY PRETRAINING
因为NER的表现对整个模型非常重要,因此早期先训练NER,延迟训练RE
如今,我们没有将RE模块的训练延迟一些时间,而是权衡RE在训练的第一个时间段对总损失的贡献
其中λ在第一个epoch从0线性增加到1,其余时期设置为1。 我们之所以选择这种方案,是因为NER模块可快速为所有数据集(即在一个epoch内)取得良好的性能。 在早期的实验中,我们发现该方案的性能优于完整时期的延迟。
![]()
IMPLEMENTATION
PyTorch BERT_BASE model 、Apex3
DATASETS AND EVALUATION
ADE
The adverse drug event corpus was introduced by Gurulingappa et al. (2012) to serve as a benchmark for systems that aim to identify adverse drug events from free-text. It consists of the abstracts of medical case reports retrieved from PubMed6. There are two entity types, Drug and Adverse effect and one relation type, Adverse drug event.
Similar to previous work (Li et al., 2016; 2017; Bekoulis et al., 2018b), we remove ∼130 relations with overlapping entities and evaluate our model using 10-fold cross-validation, where 10% of the data within each fold was used as a validation set, 10% as a test set and the remaining data is used as a train set. We report the macro F1 score averaged across all folds.
不良药物事件语料库由Gurulingappa等人引入。 (2012年)作为旨在从自由文本中识别不良药物事件的系统的基准。 它包含从PubMed6中检索的医疗案例报告的摘要。 有两种实体类型,药物和不良反应,一种关系类型,药物不良事件。
与之前的工作类似(Li等人,2016; 2017; Bekoulis等人,2018b),我们删除了约130个与重叠实体的关系,并使用10倍交叉验证对我们的模型进行了评估,其中每个数据中的10% fold用作验证集,10%用作测试集,其余数据用作训练集。 我们报告宏观F1得分在所有折叠中的平均值。
Fei Li, Yue Zhang, Meishan Zhang, and Donghong Ji. Joint models for extracting adverse drug events from biomedical text. In IJCAI, volume 2016, pp. 2838–2844, 2016.
Fei Li, Meishan Zhang, Guohong Fu, and Donghong Ji. A neural joint model for entity and relation extraction from biomedical text. BMC bioinformatics, 18(1):198, 2017.
Giannis Bekoulis, Johannes Deleu, Thomas Demeester, and Chris Develder. Adversarial training for multi-context joint entity and relation extraction. arXiv preprint arXiv:1808.06876, 2018b.
HYPERPARAMETERS
除了批处理大小,学习率和训练时期数外,我们在所有实验中都使用了相同的超参数(请参阅表A.2)。 与Devlin等类似。 (2018),使用最小网格搜索为每个数据集选择学习率和批次大小(请参见表A.3)。
手动选择的一个超参数是用于初始化BERTBASE模型的预训练权重的选择。 对于一般领域语料库,我们从Devlin等人的案例中找到了套用的BERTBASE权重。 (2018)运作良好。
For biomedical corpora, we used the weights from BioBERT (Lee et al., 2019), which recently demonstrated state-of-the-art performance for biomedical NER, RE and QA.
对于生物医学语料库,我们使用了BioBERT的权重(Lee等人,2019),该权重最近展示了生物医学NER,RE和QA的最新性能。
同样,对于临床语料库,我们使用Peng等提供的权重。 (2019年),他对来自MIMIC-III7的PubMed摘要和临床笔记进行了BERTBASE的预培训。
Jinhyuk Lee, Wonjin Yoon, Sungdong Kim, Donghyeon Kim, Sunkyu Kim, Chan Ho So, and Jae- woo Kang. Biobert: pre-trained biomedical language representation model for biomedical text mining. arXiv preprint arXiv:1901.08746, 2019.
RESULTS


我们显示了四个易于理解的模式:注意下一个和上一个单词,注意单词本身,并注意句子的结尾。
DISCUSSION AND CONCLUSION
我们的模型以最大的幅度(6.53%)超越了ADE的最新技术性能。虽然令人兴奋,但我们认为该语料库特别容易学习。大多数句子(约68%)是针对两个实体(药物和不良作用,以及一个关联(药物不良事件))进行注释的,表面上,一个模型应该能够利用这种模式来获得对药物的近乎完美的性能。作为测试,我们再次运行模型,这次使用RE模块中的地面真实实体(与预测实体相对),发现该模型很快在RE上达到了几乎完美的性能。测试集(约98%),因此,ADE语料库的高性能不太可能转移到涉及各种生物医学文章的大规模注释的真实场景中。
在我们的实验中,我们仅考虑句子内的关系。但是,文档中的多个实体通常表现出复杂的句间关系。我们的模型当前无法提取此类句子间关系,因此我们对句子内关系的限制将限制其在某些下游任务(例如知识库创建)中的有用性。我们也忽略了在生物医学语料库中常见的嵌套实体的问题。将来,我们希望扩展我们的模型,以同时处理嵌套实体和句子间关系。最后,鉴于存在BERT的多语言,预训练权重,我们还希望模型的性能能够支持多种语言。我们把这个问题留给以后的工作。

