点击蓝字 关注我们
极端环境原核微生物培养策略

iMeta主页:http://www.imeta.science
方法论文
● 原文链接DOI: https://doi.org/10.1002/imt2.123
● 2023年6月12日, 中山大学李文均团队在 iMeta 在线发表了题为 “Cultivation strategies for prokaryotes from extreme environments ” 的文章。
● 本研究首先介绍了嗜极微生物的通用分离步骤,然后总结了多种从极端环境中获取纯培养原核微生物的策略,提出了一系列在微生物分离工作中容易被忽视但重要的技巧,并指出多组学指导的培养方式将在未来大有可为。
● 第一作者:杨子文
● 通讯作者:李文均 ([email protected]) 、焦建宇([email protected])
● 合作作者:连政汉、刘兰、房保柱
● 主要单位:中山大学、南方海洋科学与工程广东省实验室(珠海)、中国科学院新疆生态与地理研究所
亮 点
● 提供了一个从不同极端生境中分离原核微生物的直接有效的方案。
● 开发了一种生物信息学工具,可以轻松地将多条16S rRNA基因序列在EzBioCloud数据库中进行鉴定。
● 提出了多组学指导的微生物培养方法对于培养尚未培养的微生物的新观点。
摘 要
绝大多数的极端环境微生物目前仍尚未被培养。虽然高通量测序可以获得来自单细胞测序和宏基因组测序技术的微生物基因组,使得研究人员能够初探意想不到的“微生物暗物质”的遗传多样性。然而,从极端环境中培养微生物对于了解和利用极端微生物仍然是必不可少的。因此在本文中,通过之前的成功分离工作和在极端微生物资源挖掘方面的长期经验,我们提供了一种直接有效的方案,从而可以从不同的极端环境(高温、干旱、盐碱、酸性和低温环境等)中分离原核微生物。本文首先介绍了嗜极微生物的通用分离步骤,然后总结了多种从极端环境中获取纯培养原核微生物的策略,并提出了一系列在微生物分离工作中容易被忽视但重要的技巧。此外,我们提议使用多组学指导的微生物培养方法来培养这些尚未培养的微生物,并提供了两个例子来介绍这些方法是如何工作的。综上所述,该方案能帮助研究人员显著提高纯培养原核微生物和新类群的分离效率,从而为极端环境微生物资源的保护和利用铺平了道路。
视频解读
Bilibili:https://www.bilibili.com/video/BV1xh4y1377w/
Youtube:https://youtu.be/iAO-YORV0Rw
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
全文解读
引 言
极端生境(包括热泉、热液喷口、冰川、永久冻土带、沙漠、盐湖、盐田和酸性矿山废水等)是地球上重要的地质形态,它们孕育着各种各样的极端微生物。由于独特而恶劣的条件,生活在这些环境中的微生物通常会演化出不同的策略来应对极端的环境压力,从而定义了生命所能生存的边界。这些极端微生物不仅可以提供独特的产物(如Taq DNA聚合酶),而且还是研究微生物生态学、进化和环境适应的理想目标。不仅如此,极端微生物也被认为是研究生命起源和进化甚至地外生命的最佳模型。
近几十年来,随着测序技术的发展,多组学研究使研究人员无需获得微生物纯培养就能获得相应微生物遗传信息,极大地提高了我们对极端环境下微生物的认识,甚至改变了“生命之树”的结构。对微生物群落进行的免培养研究揭示了微生物群落与极端环境之间有趣的关联,但这些观察到的关联(包括代谢功能)只是基因组信息预测的潜在关联,而不是直接证据。相比之下,纯培养的方法不仅可以恢复微生物完整的参考基因组和开发新的遗传操作系统,还可以全面地对微生物进行生理和酶学的研究。因此,从极端环境中培养微生物是研究极端微生物多样性、功能和进化的必要条件。也正因如此,微生物培养工作目前也越来越受到人们的重视。尤其是近年来,一些令人振奋的培养成果极大地拓展了我们对极端环境下生存的微生物乃至真核生物起源的认识。不仅如此,根据一些案例可以发现,纯培养的方法还可以研究微生物群落中的极端稀有或那些难以提取DNA的微生物类群,而这些微生物却很难被免培养技术探测到。因此,在极端环境中培养微生物不仅重要而且必要。因此在本文中,我们希望与从事极端环境微生物的研究人员分享我们的经验和策略,从而促进极端环境微生物的分离工作。
极端微生物分离的通用策略
极端微生物分离的通用步骤可分为四个部分和九个步骤(图1)。尽管许多研究人员已经在极端环境中分离菌株方面有了一些经验,但仍然有许多经常被忽视的细节,而这些细节可能会最终影响分离的结果。
极端环境样品采集
材料和设备:无菌手术刀和铁铲、无菌离心管、无菌密封袋、亨盖特厌氧管、冰袋和冰箱。
样品采集
从盐湖、海洋沉积物、沙漠沙、酸性矿山废水(AMD)等常温生态位中采集不同种类的样品,需要使用无菌手术刀或铲子收集,并立即转移到无菌离心管或密封采样袋中。这类样品通常保存在低温(4°C)下,以便以后的分离工作和理化特性分析实验。从热泉中收集的样品通常保存在室温下,因为这有助于保持菌株的活性,并更好地用于后续的微生物分离。需要进行厌氧分离的样品需要用半胱氨酸或硫化钠等还原剂保存在无菌亨盖特厌氧管中。刃天青是一种氧化还原染料,需要加入刃天青来确认样品处于无氧状态。用于DNA提取的样品通常混合并收集在无菌的离心管中,并立即储存在液氮中,然后运回实验室并在DNA提取前保持在-80°C。注:对于4℃保存的样品,微生物群落会随着时间的推移而发生变化。因此,根据以往的实验和前期的微生物分离的工作经验,我们建议在4℃保存的样品最好在20天内进行微生物分离工作。
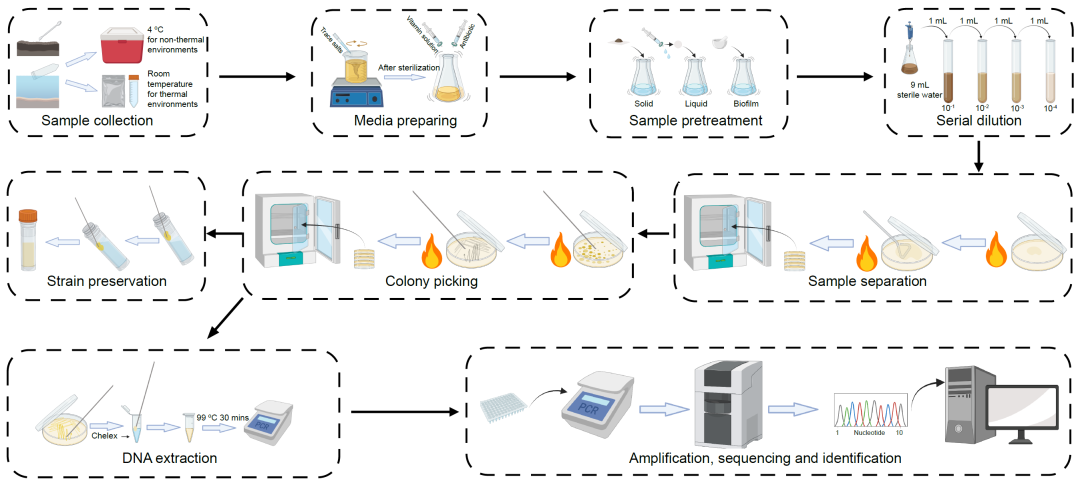
图1. 从极端环境中进行微生物分离的九个通用步骤
培养基准备与样品预处理
材料和设备:培养皿、酒精灯、涂布棒、移液器和枪头、15 mL离心管、100 mL锥形瓶、玻璃微珠、0.22 μm滤膜、10 mL注射器、研钵和杵、无机盐、复合维生素、制霉菌素和二甲基亚砜(DMSO)、琼脂、精密天平、高压灭菌器、摇床、恒温培养箱和超净台。
培养基准备
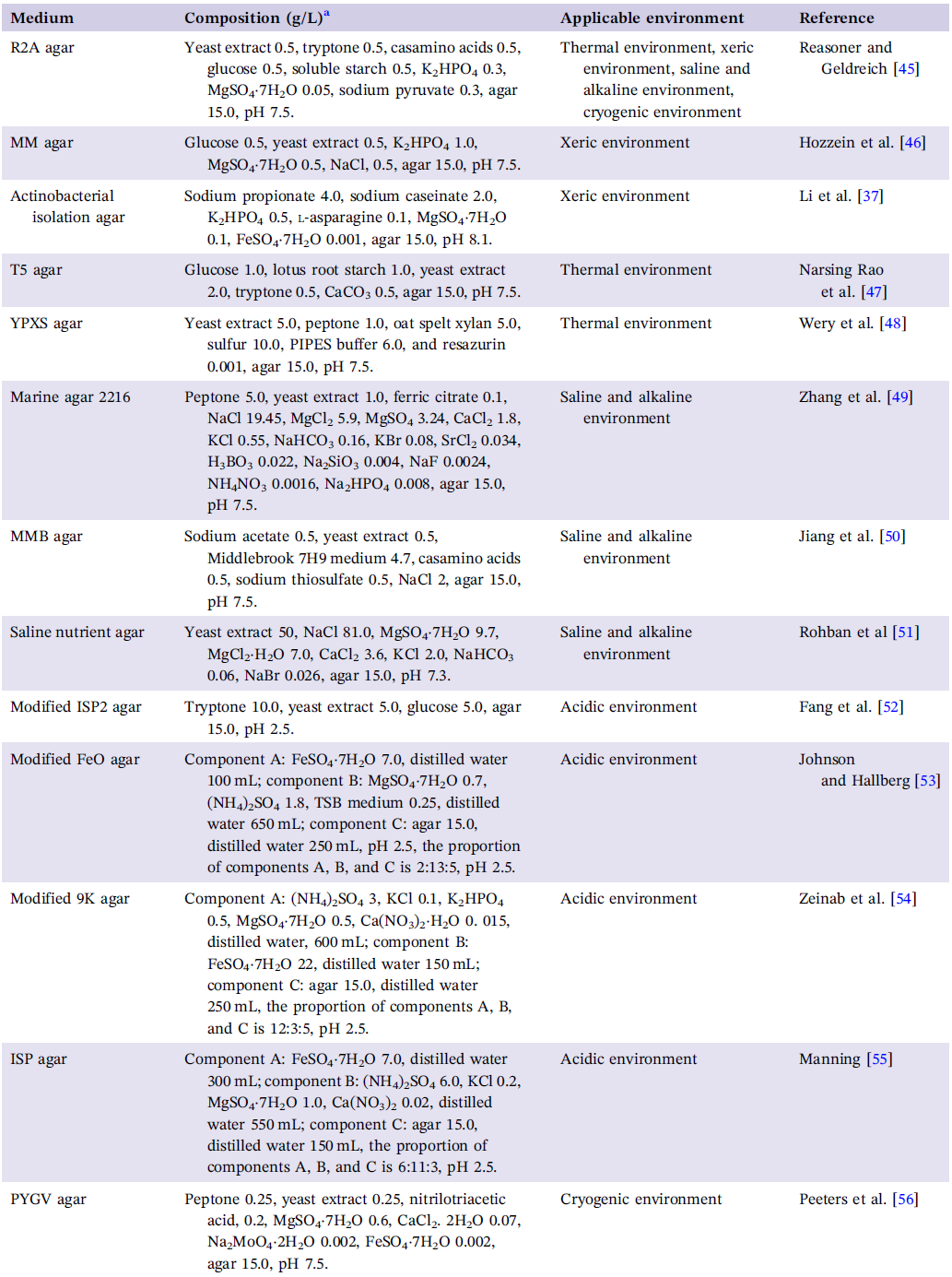
在微生物分离实验中,选择合适的培养基是实验成败的最关键步骤之一。表1列出了我们对相应的极端环境的微生物分离实验所推荐的培养基。除了培养基本身之外,我们还推荐加入微量盐和复合维生素到分离培养基中。微量盐的推荐配方为:MgSO4·7H2O 3 g/L、MnSO4·2H2O 0.5 g/L、NaCl 1 g/L、FeSO4·7H2O 0.1 g/L、CoCl2 0.1 g/L、CaCl2 2H2O 0.1 g/L、ZnCl2 0.1 g/L、CuCl2·2H2O 0.02 g/L、AlK(SO4)2 0.01 g/L、H3BO3 0.01 g/L、Na2MoO4·2H2O 0.1 g/L、NiCl2·6H2O 0.03 g/L、Na2WO4·2H2O 0.03 g/L。每升培养基应加入10毫升微量盐。复合维生素溶液的推荐配方为:生物素5 mg /L、叶酸5 mg /L、盐酸吡哆醇25 mg /L、核黄素12.5 mg/L、硫胺素12.5 mg/L、烟酸12.5 mg/L、泛酸12.5 mg/L、钴胺素5 mg/L、对氨基苯甲酸12.5 mg/L、硫辛酸12.5 mg/L。每升培养基应添加2毫升复合维生素溶液。此外,如果不需要对真菌或藻类进行分离,可以在分离培养基中加入制霉菌素(DMSO溶液,工作浓度0.05 mg/mL)来抑制快速生长的真核微生物。复合维生素溶液和制霉菌素均需经过0.22 μm滤膜过滤,待培养基灭菌后再加入。在制作分离平板时,倾倒的培养基的高度最好在培养皿的1/2,并应风干一夜以去除表面水分。注意:培养基pH值过高/过低或需要进行培养的温度过高时,不宜使用琼脂。这些因素会破坏琼脂的氢键使其难以固化。在这些情况下,吉兰胶仍具有凝固能力,并且可以获得更好的效果,因此是一个更加推荐的选择。当培养基含有磷酸盐时。最好将磷酸盐和琼脂培养基分开灭菌,从而可以通过减少过氧化氢的生成来大大提高微生物的可培养性。由于获得样品的来源千差万别,进行预实验以确定合适/最佳的分离培养基有时是必不可少的。先前的研究和我们的经验表明,与样品的原位环境相比,含有与样品相同物理和化学成分的培养基可能不是分离的合适/最佳条件,这意味着许多微生物在原始的极端环境中可能不活跃,或者需要额外的信号来激活生长。
样品预处理
极端环境下的样品大致可分为固体、液体和生物膜三种类型。对于固体样品可以将2 g样品悬浮在装有玻璃珠的18 mL生理盐水或1/10的R2A培养基的锥形瓶中,并在摇床中孵育(200转/分,1小时)。对于液体样品预处理:将2mL样品悬浮在装有18 mL生理盐水或1/10 R2A培养基的锥形瓶中。如果液体样品的微生物生物量明显较低,则最好先使用0.22 μm滤膜过滤样品,然后与固体样品一样的方式进行处理。生物膜样品首先需要进行完全研磨,然后处理与固体样品使用相同的方式进行处理。注:此处可以使用1/10 R2A培养基代替无菌生理盐水,因为它含有丙酮酸、氨基酸等物质,这些营养物质对于复苏休眠菌株有用的。然而,如果使用1/10 R2A培养基代替生理盐水,可能有加速易生长菌株生长的风险。在这种情况下,减少孵育时间将有效地减少这类微生物的生长。
梯度稀释
将1 mL悬浮液从锥形瓶转移到15 mL离心管中,并向管中加入9 mL无菌生理盐水。用移液器将管中的液体吹打混匀后,将1 mL悬浮液转移到另一根装有9 mL无菌生理盐水15 mL离心管并重复上述步骤从而将样品稀释至10−1、10−2、10−3等浓度并依此类推。合适的稀释浓度会根据每个样品的生物量而变化。因此,在进行大规模正式实验之前,最好先进行预实验以确定合适的稀释浓度。注意:将枪头的尖端剪掉并吹打混合悬浮液会有助于样品转移。
表1. 各种极端环境推荐培养基

样品稀释涂布
将100 μL的悬浮液加到到分离平板表面,使用涂布棒在平板表明均匀涂布。在整个过程中,平板需要与酒精灯的火焰保持3-5 cm的距离。涂布过程一直持续到倾斜平板后观察到没有流动的水体。每个培养皿需要记录培养基、样品类型、温度、稀释系数、接种日期等信息并标记在培养皿盖上。涂布完成的平板需要先在超净台正面朝上放置2小时。之后将培养皿倒置并放入孵育箱中。注:光对于一些重要的类群,如蓝藻的分离是必需的,光也可以提高铁氧化细菌等类群的分离效率。而这些类群恰好是某些极端环境中的常见类群,因此在对分离平板进行培养时,光也需要作为一个重要的参数来考虑。
微生物的分离与保存
材料和设备:培养皿、酒精灯、接种针、甘油和2 mL冻存管、无机盐、复合维生素、制霉菌素和DMSO、琼脂、精密天平、高压灭菌器、冰箱、超低温冰箱、恒温培养箱和超净台。
菌落挑取
分离平板应在孵育3、7和14天后观察其生长情况(某些样品,如永久冻土,可能需要更长的时间)。待离散菌落清晰可见并均匀分布于培养基表面后,即可开始挑取菌落。挑取前,最好在平板底部圈出目标菌落,并标注序列号。目标菌落最好与其它菌落分开,避免挑取时被其它细菌污染。菌落挑取时应尽可能基于菌落的颜色、大小、透明度、边缘特征和表面特征进行选择,以避免重复挑取。将单个菌落转移到初步分离平板上并用平板划线法进行纯化。分离菌株在初步分离平板上完成生长后,可将其接种到统一培养平板上(盐环境所分离菌株一般选择Marine Agar 2216E培养基,其他环境所分离菌株一般选择R2A培养基),以减少工作量。初步分离平板上菌体可用于DNA提取和初步鉴定,培养平板上的菌体可用于微生物保存。注意:虽然非常小的菌落可能看起来非常相似,但它们可能还没有完全成熟,这意味着不同的菌株或物种可能形成相似的菌落。因此,适当多挑取这些很小的菌落是有必要的。同时,外形相似的大型菌落可能不是同一株菌种,但它们在某些代谢功能上可能存在差异,因此在去除重复的步骤中需要对此更加小心。从分离平板上在不同的孵育时间进行挑取工作可以得到更多种类的分离菌株。此时,平板底部的数字将有助于识别最近生长的菌株。我们还发现一些古菌或其他生长缓慢的菌株会很晚才形成菌落。因此,我们建议在菌落挑取工作后仍将分离平板保存1至2个月,并观察是否有生长缓慢的菌落形成。
微生物保存
使用无菌的装有1.5 mL甘油(20%,v/v)的2 mL冻存管来保存分离得到的纯培养微生物。首先用无菌接种针采摘适量的新鲜菌体(约绿豆大小),将菌体均匀地涂布在管壁上,然后倾斜试管,使甘油覆盖扩散区域,再使用接种针轻轻搅动以形成悬浮液。悬浮液应先在-20℃预冻2小时,然后在-80℃保存。注意:在涂抹和搅动时避免剧烈的动作否则可能会引入外来污染。另外,液体培养的原核生物也可以在甘油(20%,v/v)中形成悬浮液后用于菌株保存。
微生物的初步鉴定
材料和设备:移液器和枪头、PCR管、剪刀、Chelex‐100、Tris‐EDTA缓冲液和PCR仪。
DNA提取
对于大规模的DNA提取实验,我们推荐使用10%的Chelex悬浊液进行DNA提取(10克Chelex-100加到90 mL Tris‐EDTA缓冲体系中)。具体步骤如下:(1)摇动Chelex悬浊液,然后用一个剪短尖端的枪头转移50 μL悬浊液到PCR管中。(2)将少量菌体加入到PCR管中,与悬浊液均匀混合。(3)将PCR管放入PCR仪或水浴锅中,并在99°C加热30分钟。(4)短暂离心使细胞碎片和Chelex沉淀到PCR管的底部,而上清液可用于PCR扩增实验。如果某些含有过多色素或细胞外聚合物质的菌株无法使用此方式提取DNA,则建议使用苯酚-氯仿法提取DNA。
DNA扩增、测序及菌株鉴定
16S rRNA基因通常被用作来对原核微生物进行初步鉴定。用于细菌16S rRNA基因扩增的引物为27F(5′‐CAGAG TTTGATCCTGGCT‐3′)和1492R(5′‐AGGAGGTGATC CAGCCGCA‐3′),用于古菌的引物为P1(5′‐ATTCCGGTT GATCCTGCCGGA‐3′)和P2(5′‐AGGAGGTGATCCA GCCGCAG‐3′)。PCR扩增按照以下程序进行:94°C初始变性5分钟;然后在94°C变性45秒,在56°C退火30秒,在72°C延伸1分钟30秒,持续32个循环;最后在72°C延伸10分钟。得到的PCR产物可以使用Sanger测序技术进行测序。获得的序列可以使用EzBioCloud数据库和NCBI Basic Local Alignment Search Tool(blast)进行鉴定。值得注意的是,EzBioCloud是提供经过质量控制的16S rRNA基因序列数据库,从而可以提供可靠的基于相似性的搜索,但是需要我们进行逐一鉴定。为了减少重复提交单个序列的繁重工作量,我们开发了一个生物信息学工具,可以轻松地分析16S rRNA基因序列,并与EzBioCloud数据库进行比对,从而实现自动提交、识别序列(https://github.com/lianzhh/easy_EzBioCloud)。每个纯培养所鉴定的最相似菌株的信息将被保存,并在鉴定完成后生成一个饼状图,用于分析可培养微生物的多样性。一般来说,如果一株分离物的16S rRNA基因序列与已有效发表的菌株相似性低于98.65%,我们认为该分离物为潜在的的新物种。注:由于Sanger测序技术的特点,该技术获得的序列的起始和末端是不可靠的。Sanger测序的数据应通过MEGA或BioEdit等工具进行可视化和检查并切割不可信的碱基序列。
不同极端微生物分离的特定策略
不同极端环境由于其独特的限制因素而具有独特的微生物群落。因此,在进行不同极端环境可培养微生物分离工作时,需要采取不同的分离策略以及关注到不同的注意事项(图2)。
高温环境
热环境主要包括陆地热泉和深海热液喷口。高温是这些地方限制生命存在的主要因素之一。为了成功分离热环境微生物,提高分离温度是必要的。但高温会使琼脂变性液化,因此如果分离培养温度高于60℃,应使用0.8%的吉兰胶代替琼脂。高温也会使培养基变干,因此需要将分离平板装在塑料袋里,并将一个装满水的培养皿放入塑料袋,以保持湿润。此前的研究表明,热泉样品经过低温保存后,其生物多样性会迅速下降。因此,热泉样品的分离时间应在样品新鲜时进行;如果样品需要保存,常温保存比低温保存效果更好。作为热泉环境中的优势类群,Thermus属的成员引起了科研工作者的广泛关注,而在培养基中添加硫代硫酸钠会提高该类群的分离效率。热液喷口内及周围微生物的营养需求非常复杂,因此单一类型的培养基往往不能很好地发挥作用,所以若是想从这些生境中分离原核微生物,那么分离培养基应考虑提供各种营养需求。此外,热环境通常同时生存着好氧菌株和厌氧菌株,因此在进行微生物分离工作时也需要考虑氧气的作用。

图2. 从不同极端环境中分离微生物的策略和建议
干旱环境
沙漠和雅丹地貌是典型的干旱环境,由于干旱和太阳的紫外辐射,而一直被认为是不毛之地。然而,即使是自然条件最恶劣的阿塔卡马沙漠,也存在着数量庞大、种类繁多的微生物。与其他极端环境相比,沙漠样品的前处理策略可以更加激进。一般情况下,45 kHz超声处理2 min是一种可靠的样品预处理策略,可以有效抑制快速生长菌株的生长。另外,我们在超净台中使用紫外线照射10分钟处理沙漠样品,与正常培养方法相比,这种方法获得了更高的纯培养微生物多样性。此外,尽管沙漠环境中的盐浓度通常不高,但一些从沙漠环境分离的菌株却可以忍受极端的盐条件。例如,我们分离并描述了一个物种Saccharopolyspora deserti,它可以耐受高达22%的NaCl (w/v;≈ 3.8 M)。因此,在分离培养基中添加不同浓度的氯化钠可能有助于分离更多独特的菌株。干旱样品中放线菌门成员特别是链霉菌的比例和多样性非常高,表明干旱生境可能是次生代谢物生物勘探的宝库。分离培养基中添加萘啶酮酸(25 mg/L)、重铬酸钾(25 mg/L)和放线菌酮(25 mg/L)可以对竞争菌的生长有明显抑制作用,从而促进放线菌门成员的分离。
盐碱环境
高盐环境如盐湖、盐沼和盐碱地中生活着大量的嗜盐菌和嗜碱菌。通常,盐碱样品在分离前不需要风干,但是风干确实是获得更高比例的嗜盐或嗜碱放线菌和古菌的有效方法。还可以注意到,不同的盐环境具有不同的盐成分,因此可能需要根据样品的物理化学性质在培养基中添加其他无机盐,如KCl或MgCl2等。此外,高浓度的盐在灭菌时可能会与培养基中的有机成分形成沉淀,因此当这种现象发生时,盐和培养基应分开灭菌。在盐环境中,嗜盐古菌在微生物群中占有相当大的比例。添加25 mg/L的氯霉素可有效抑制盐单胞菌等嗜盐细菌的生长,提高嗜盐古菌的分离效率。此外,生活在这些环境中的微生物通常需要特定的盐浓度来维持其正常生理所需的渗透压。因此,除了在培养基中,还应在样品预处理溶液和甘油保存溶液中加入5%-10%的NaCl来维持这些菌株的活性。
酸性环境
酸性矿山废水是一个典型的酸性环境,它同时有着pH值低以及重金属离子浓度高的特点。因此,酸性矿山废水环境中的微生物会受到这些条件的限制导致丰富度和生物量很低,这意味着在样品预处理步骤中,酸性矿山废水样品需要使用0.22 μm的滤膜进行富集,并降低稀释倍数。通常,用于从酸性矿山废水样品中分离菌株的培养基含有很少或不含有机营养素。然而,我们测试了一种营养丰富的培养基:改良的ISP2培养基(表1),使用这种培养基从酸性矿山废水沉积物样品中分离得到多种放线菌,取得了意想不到的效果。此外,一种被称为“覆盖技术”的方法已被用于极端酸性环境的微生物分离。简而言之,该方法首先将选定的异养型嗜酸菌接种到培养基并倒入无菌培养皿中,在底部培养基固化后加入第二层无菌培养基。这样,这种异养型嗜酸菌既可以清除培养基中的有毒物质,又不会干涉到上层微生物的生长。这种方法的具体实施步骤可以在Johnson和Hallberg所写的书中找到。此外,值得注意的是,由于高温和低pH值可能使琼脂变性,使其难以固化。因此,分离嗜酸菌的培养基和琼脂必须分别灭菌,必要时还需使用吉兰胶代替琼脂。
低温环境
冰川和永久冻土是典型的低温环境,它们约占地球表面积的10%,且温度常年保持在0℃以下。因为极低的温度导致这两种环境的微生物的生物量偏低,因此在样品预处理步骤中,最好将冰样融化然后使用0.22 μm滤膜富集微生物。与冰川分为极地冰川和高山冰川类似,永久冻土也分为高海拔永久冻土和高纬度永久冻土。它们的理化性质和营养条件以及微生物群落组成都是不同的,这意味着在进行微生物分离时需要考虑这些差异。例如,与中国东北的永久冻土相比,青藏高原的永久冻土含有较低的有机质和无机盐从而导致微生物生物量更低。因此,从青藏高原冻土样品中分离菌株的培养基建议用1/4 R2A或1/10 TSA这些更寡营养的培养基进行培养。从前期的分离结果来看,大多数从低温环境中培养的微生物似乎主要是耐寒菌而不是嗜冷菌,这些菌株的最佳生长温度一般为12°C至25°C之间。因此除了低温(如0°C或4°C)外,还应选择中等温度(如16°C或25°C)进行微生物分离。出于同样的原因,冻土样品可在4℃下保存数周,以增加可培养细菌的数量。我们还发现一些嗜冷菌生长非常缓慢,在低温下需要几个月的时间才能形成菌落,因此在较低的温度下,分离平板的培养时间需要延长到至少2个月。与来自沙漠环境的菌株类似,从冰川或永久冻土中分离出来的许多嗜冷菌被发现可以耐受一定的盐浓度。因此,在分离培养基中添加3% ~ 5% (w/v)的NaCl可以提高可培养菌株的多样性。
厌氧极端环境微生物
厌氧微生物分布广泛,在极端环境中发挥重要作用。然而,因为分离必须在缺氧条件下进行,因此与好氧微生物相比,厌氧微生物的分离具有更大的挑战。分离这些菌株的关键是去除培养基中的氧,从而保持一个厌氧的环境。亨盖特厌氧滚管技术是分离厌氧菌的传统方法,适用于培养严格厌氧菌,如产甲烷菌。另外,厌氧培养箱技术的优点是可以使用培养皿,并在培养许多厌氧菌方面获得成功。简而言之,厌氧培养箱技术是在好氧条件下制备培养基,灭菌后立即转移到含有N2:H2:CO2(8:1:1)气相的厌氧工作站。然后在厌氧条件下将培养基与无菌还原剂一起倒入培养皿中。最后,将样品匀浆,在无菌生理盐水中连续稀释至10−3,取100 μL稀释液涂于分离板上进行厌氧培养。值得强调的是,样品也可以通过富集的方式获得特殊的厌氧群落。例如使用带丁基橡胶盖的血清瓶,来减少长时间富集过程中氧气的影响,同时也可以在达到纯培养后扩大微生物的生物量。此外,针对厌氧菌株生长缓慢的特点,微流控系统或其他单细胞培养方法将有助于将不同细胞隔离开来,从而使缓慢生长的菌株不会受到快速生长菌株的影响。
多组学指导下的微生物纯培养
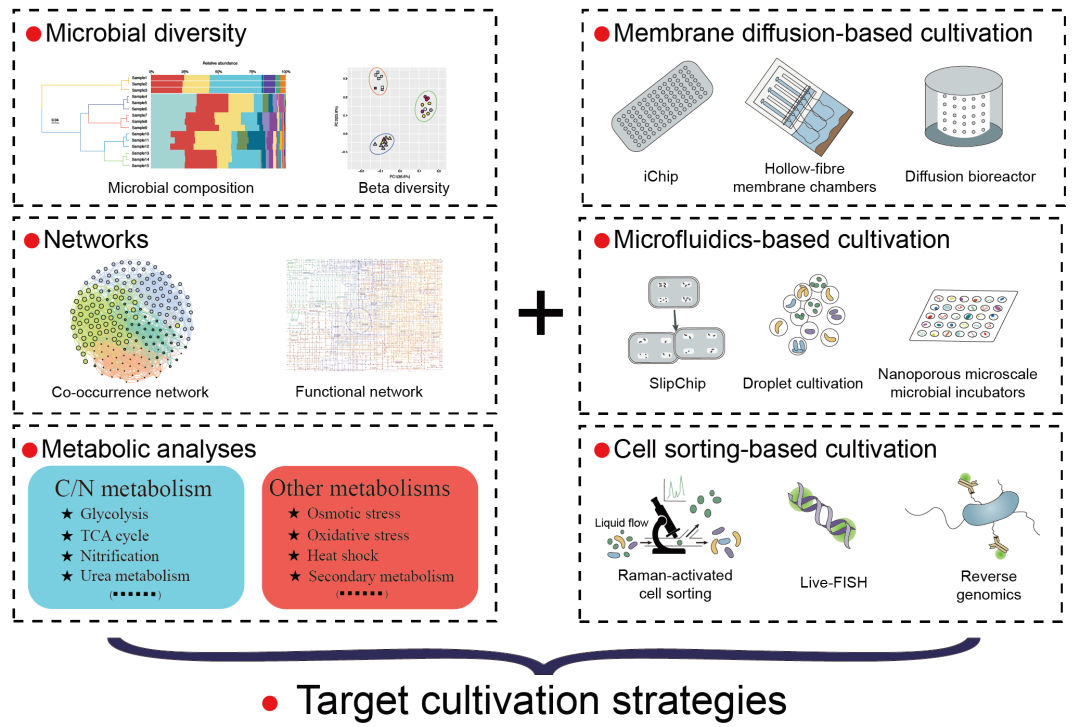
我们上面介绍的培养策略是非靶向性的,适用于生活在极端环境中的所有原核微生物。随着各种高通量测序技术和算法的发展,通过对宏基因组组装基因组(MAGs)、单细胞测序基因组(SAGs)或其他组学方法的分析,发现了一系列具有特定功能或在系统发育分析中具有重要意义的未培养微生物。这些从MAGs和SAGs描述的分类群现在还可以使用SeqCode来命名。但是尽管各种高通量测序技术取得了进步,微生物纯培养仍始终需要来为微生物(例如生理学)的研究提供材料。这是一项非常具有挑战性的任务。幸运的是,来自SAGs、MAGs和其他方法的信息可以与其他技术(放射自显影技术、荧光原位杂交、质谱、拉曼光谱等)和创新培养方法(膜扩散/细胞分选/微流控)相结合,为合理设计靶向培养策略提供必要的框架(图3)。例如Chen等人利用Micrarchaeota和Parvarchaeota的39个SAGs /MAGs来推断它们的代谢潜力,然后成功设计了低氧培养策略来确认它们的微氧/厌氧生活方式。此外,创新的培养方法(图3),如分离芯片技术,允许微生物与环境和其他物种交换代谢物,可以增加可培养微生物的数量。此外,据报道,还有一种免疫磁珠富集培养组学方法能够特异性地分离出特定疾病的潜在病原体。值得注意的是,在传统的培养方法设计中,微生物的相互作用被忽略了,这可能是许多尚未培养的微生物仍然无法获得培养的一个关键原因。因此,来自16S rRNA基因数据集的共现网络,以及来自多组学数据的功能网络的网络分析技术,能够为指导培养策略的设计提供很大的机会。在这里,我们分享了两种利用多组学指导微生物进行靶向分离的方法(图4)。
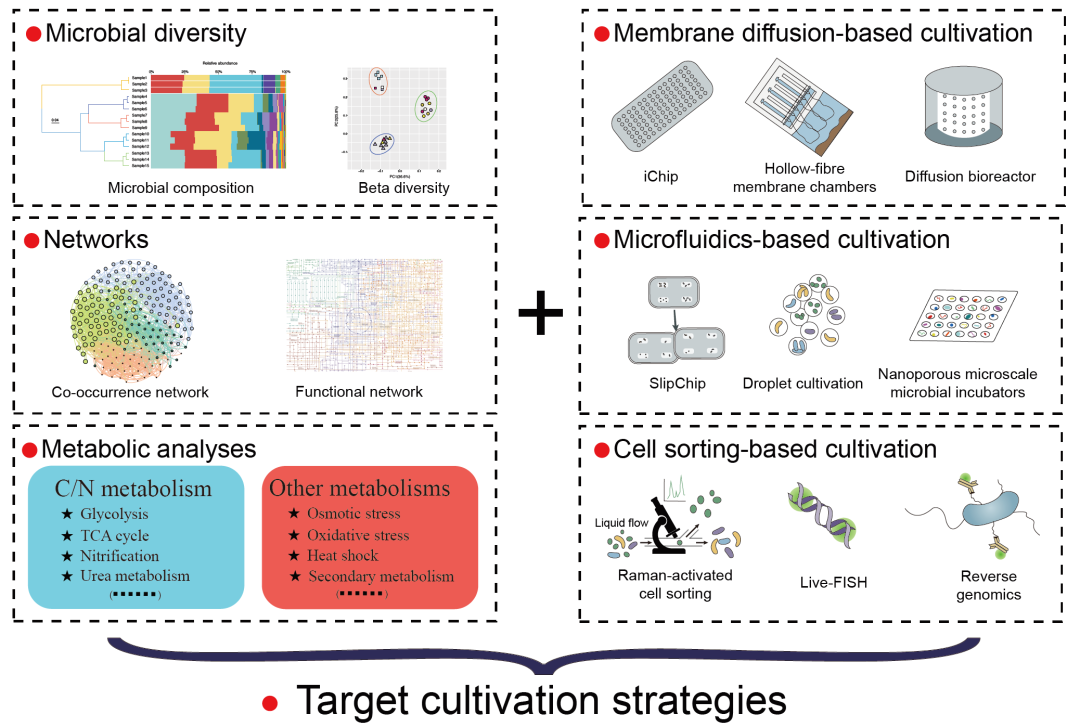
图3. 靶向微生物培养策略示意图
放线菌门是细菌域中最大的门类之一,其成员以其广泛的次级代谢而闻名。到目前为止,我们对这一群体的大部分认识都是基于纯培养的,未经培养的放线菌的生态功能仍然不清楚。通过分析宏基因组数据,我们发现了35个高质量的MAGs,它们代表了三种新的放线菌新纲(Ca. Geothermincolia, Ca. Humimicrobiia和Ca. Aquicultoria)。对这些MAGs的基因组推断表明,它们含有编码氢化酶、产乙酸和Wood-Ljungdahl途径的基因,表明它们是潜在的同型产乙酸菌。基因组学研究表明,不同处理的厌氧富集(G55H: N2:H2:CO2的比例为5:4:1;G55HB: N2:H2:CO2的比例为5:4:1,并添加20mM的2‐溴乙烷磺酸钠)均富集了这些类群。在富集过程中,对H2、CO2和乙酸的浓度进行了测试,以验证是否发生了同型产乙酸过程。经过120天的富集,使用扩增子测序检测群落结构发现,目标放线菌的比例急剧增加,表明我们的多组学指导富集策略对Ca. Geothermincolia的成功。一般来说,通过多组学数据指导的菌株富集方式在功能微生物类群富集时会有良好的效果(图4A)。然而,它在获得纯培养物方面仍然面临困难,需要新的培养方法,如基于细胞分选的培养或基于微流体的培养和基于膜扩散的培养等方法。
另一个案例时通过网络来指导极端微生物分离(图4B)。在前期研究中,通过对西藏和云南温泉样品的高通量测序和共现网络分析,发现温单胞菌属(相对丰富度为0.014%)的可操作分类单元(OTUs)具有高度中心性(关键节点),而绿弯菌属(相对丰富度为13.9%)的OTUs形成了外围节点。基于这一结果,我们假设微生物群落是通过具有高中心性的稀有成员促进丰度更高的成员的基本代谢而来维持的。在验证我们的假设之前,我们根据基因组数据的功能网络分析证实了这两个成员具有代谢交换的潜力。因此,我们使用加入10%温单胞菌发酵上清液的R2A来分离优势的绿弯菌。经分离和测序,发现从这些样品中分离到的绿弯菌大部分为未培养类群,其中包括36个潜在新种和1个潜在新纲。这些结果表明,利用高通量测序和共现/功能网络寻找关键物种,并根据它们的相互作用关系分离微生物,可能是一种有效的极端微生物培养方法。
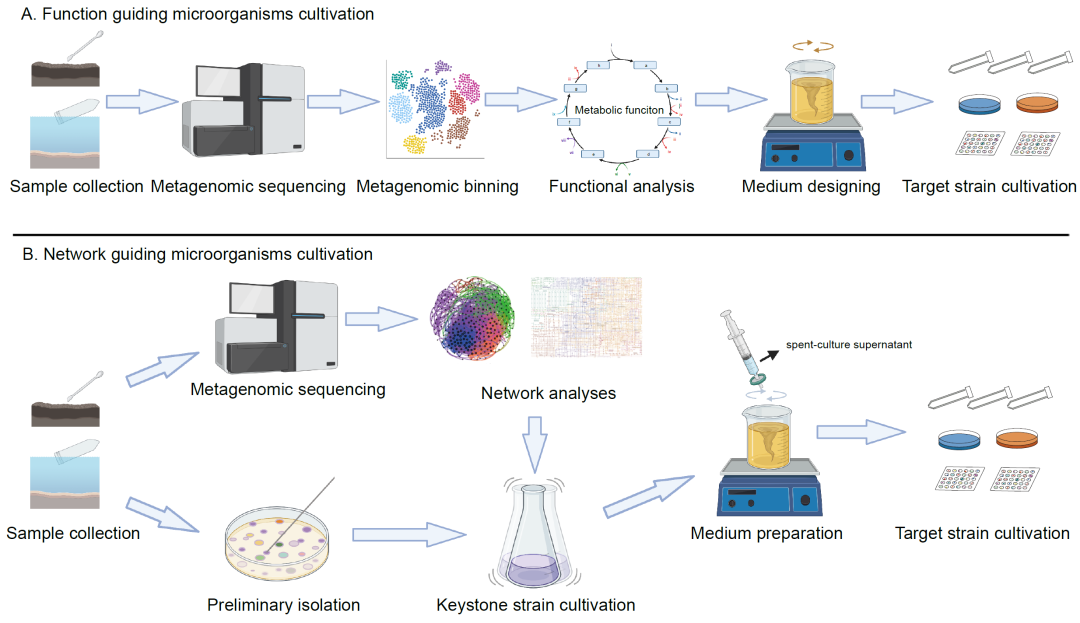
图4. 多组学指导微生物培养方法的两个例子
(A)功能引导微生物培养和(B)网络引导微生物培养
结 论
与免培养方法相比,微生物的分离和培养无疑是一种需要花费更多时间和精力的方法,但纯培养微生物在研究微生物生理学、验证微生物互作,以及生物技术应用等方面更有实际价值。极端环境下的微生物由于它们特殊的适应能力和代谢潜能,与普通环境的微生物相比更难分离。为此,本文提供了一个直接且简便的方案来有效的分离极端环境微生物,并提出了使用多组学指导的微生物培养方法来培养那些尚未培养的微生物,希望以此来突破传统微生物分离方法的瓶颈。本文最终希望能够促进极端环境微生物分离培养工作,使得从极端环境中分离微生物的工作变得更容易和高效。
引文格式:
Yang, Zi‐Wen, Zheng‐ Han Lian, Lan Liu, Bao‐Zhu Fang, Wen‐Jun Li, and Jian‐Yu Jiao. 2023. “Cultivation strategies for prokaryotes from extreme environments.” iMeta e123. https://doi.org/10.1002/imt2.123
作者简介

杨子文(第一作者)
● 中山大学博士后。
● 开展微生物资源与生态等相关领域的工作接近8年,在微生物的分离、分子生态学及分子生物学等研究方面积累了丰富的研究经验;对特殊功能微生物的富集、分离及培养具有丰富的实践经验和独特的技术。建立并生效发表了新属1个,新种7个,参与发表微生物新属或新种十几个。目前以第一作者或并列第一作者的身份发表在iMeta、Microbial Ecology等杂志发表了SCI论文10篇,担任了Microbiology Spectrum, Frontiers in Microbiology以及International Journal of Systematic and Evolutionary Microbiology等杂志审稿人。

李文均(通讯作者)
● 中山大学生命科学学院百人计划/珠江学者、逸仙学者特聘教授,中国科学院新疆生态与地理研究所兼职特聘研究员(百人计划B类),博士生导师。
● 长期从事高温、高盐碱、海洋等极端或特殊生境(动物肠道、植物内生、洞穴)微生物分类及系统学、生态学研究。现任国际原核微生物系统学委员会(International Committee on Systematics of Prokaryotes,ICSP)国际委员,伯杰氏国际系统微生物学会(BISMiS)创始会员及秘书长。同时兼任中国微生物学会微生物教学工作委员会副主任委员、国际交流工作委员会委员、普通微生物专业委员会委员、地质微生物专业委员会委员、微生物资源专业委员会委员。《Microbiome》《Environmental Microbiome》《Frontiers in Microbiology》《International Journal of Systematic and Evolutionary Microbiology》《Antonie van Leeuwenhoek》《Archives of Microbiology》《BioMed Research International》《微生物学报》杂志副主编或编辑,《mLife》《Syst Appl Microbiol》《Journal of Arid Land》《微生物学杂志》等期刊编委及多个国内外期刊或基金会的通讯评审专家。先后承担并顺利完成了包括国家科技部“973”子课题、重点研发课题、国际合作专项、国家科技基础性工作专项,以及国家自然科学基金(青年、面上、重点、国际合作共10项)等在内的40余项国家级或省级课题的研究。以第一或通讯作者已在Natl Sci Rev、Nat Commun、Microbiome、ISME J、Sci Total Environ、Environ Pollut、NPJ Biofilms Microbiomes、Chemosphere、mSystems、Org Lett、Environ Microbiol、Bioresource Technol、Genome Biol Evol、Mol Phylogenet Evol、Front Microbiol、Appl Environ Microbiol、Syst Appl Microbiol、Extremophiles、Int J Syst Evol Microbiol、ANTO等刊物上发表学术论文500余篇,参与发表Nature、Nature Microbiol、Nat Commun、ISME J及其它期刊论文近400余篇,主编或参编专著10余部,受邀撰写新版《伯杰氏系统细菌学手册》20余章,专利40余件。先后建议并命名、发表包括细菌、古菌2个新门,5个新纲,23个目,40余个新科,100个新属,超600余新种。2014-2022连续九年均入选由世界著名出版公司爱思唯尔(Elsevier)发布的中国高被引学者榜单。

焦建宇(通讯作者)
● 中山大学生命科学学院特聘副研究员。
● 主要研究方向为极端环境微生物资源与生态,自2013年起长期从事极端环境微生物资源挖掘、生态功能与适应性进化等相关领域的研究工作。主持或参与国家级项目6项;参与建立并发表原核微生物新纲3个、新目21个、新科23个、新属30个、新物种79个;以第一(含并列第一)或通讯作者(含共通通讯)在iMeta、National Science Review、The ISME Journal、mLife、Environmental Microbiome、Microbiological Research、Frontiers in Microbiology、International Journal of Systematic and Evolutionary Microbiology、Antonie van Leeuwenhoek等期刊发表论文二十余篇,并以其他作者身份参与发表学术论文六十余篇,谷歌学术总引用1000多次,H指数为14,i10指数为25(Google Scholar截止至2023.06);参编专著2部;参与撰写新版《伯杰氏古菌和细菌系统学手册》3节。现为iMeta杂志青年编委,Frontiers in Microbiology客座副主编和审稿编辑,以及多个微生物资源与生态相关期刊审稿人。
更多推荐
(▼ 点击跳转)
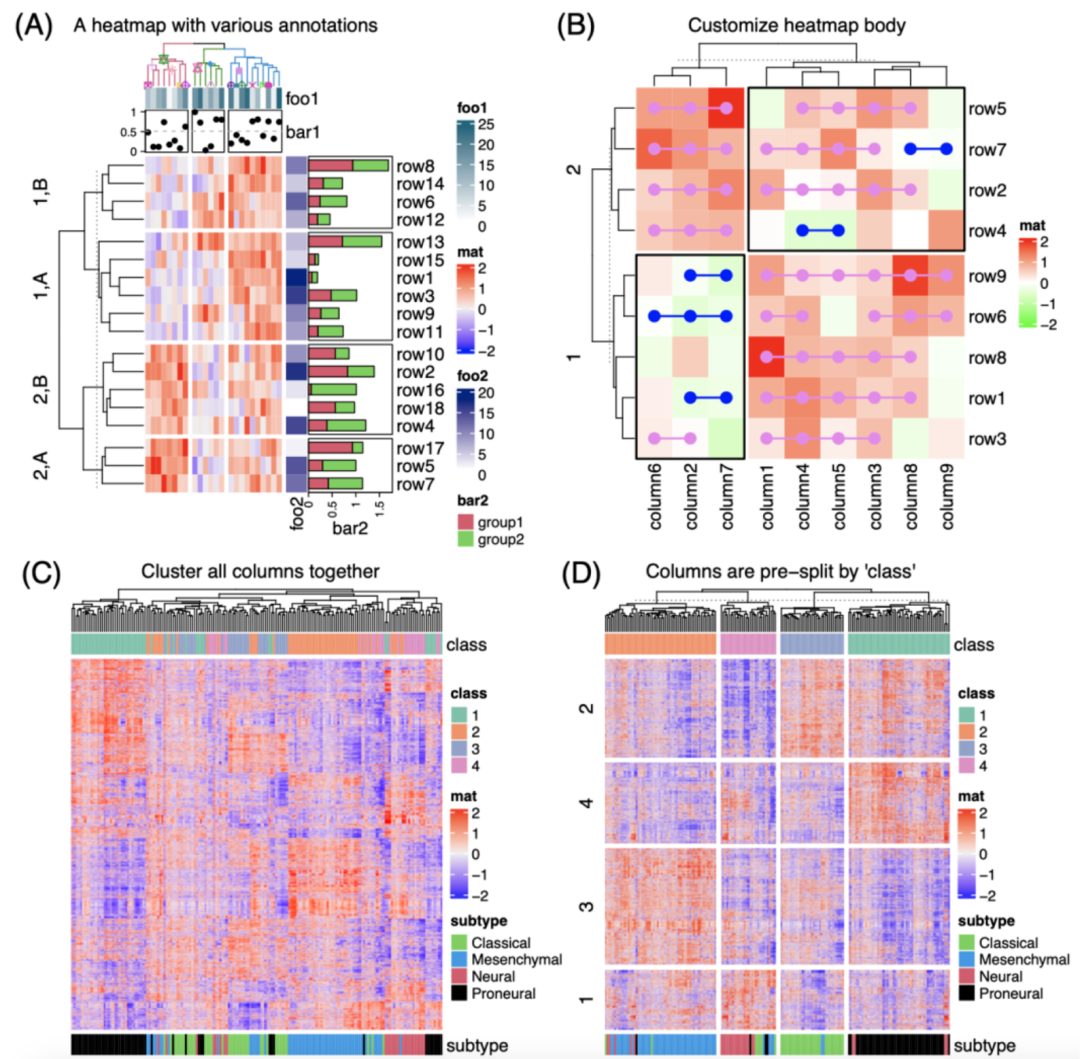
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
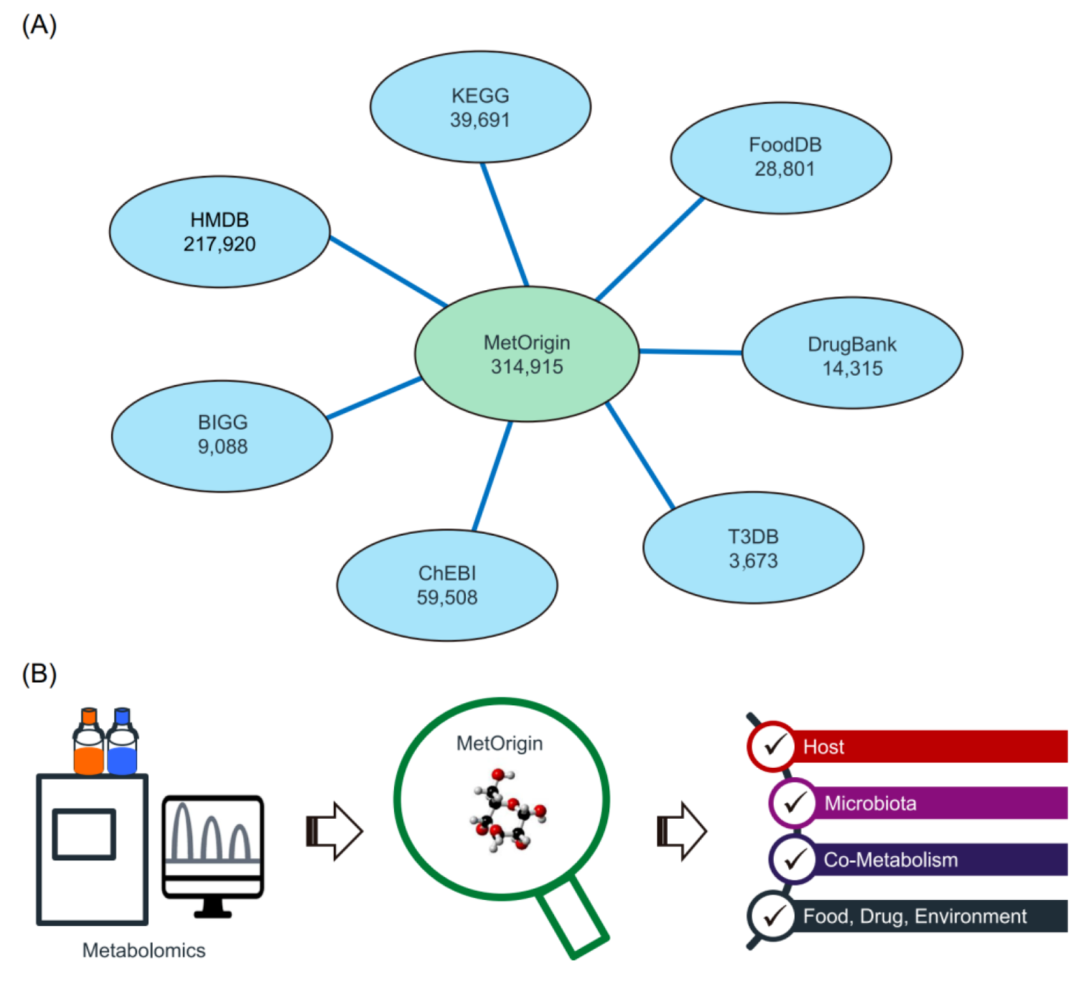
iMeta | 浙大倪艳组MetOrigin实现代谢物溯源和肠道微生物组与代谢组整合分析
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF > 15)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、前3年免出版费、50万用户的社交媒体宣传等。2022年2月正式创刊发行!
联系我们
iMeta主页:http://www.imeta.science
出版社:https://onlinelibrary.wiley.com/journal/2770596x
投稿:https://mc.manuscriptcentral.com/imeta
邮箱:[email protected]