点击蓝字 关注我们
编码胆盐水解酶的乳酸杆菌的分类鉴定:调节肝肠胆汁酸谱
iMeta主页:http://www.imeta.science

研究论文
● 原文链接DOI: https://doi.org/10.1002/imt2.128
● 2023年7月16日, 中国药科大学李萍、李菁团队在 iMeta 在发表了题为 “Taxonomic identification of bile salt hydrolase-encoding lactobacilli: Modulation of the enterohepatic bile acid profile ” 的文章。
● 本研究发现促进了人类肠道微生物组中编码BSH的关键乳酸杆菌的分类鉴定,并阐明了它们对BAs肝肠循环调节的贡献,这将有助于未来编码BSH的益生菌应用于治疗以改善人类健康。
● 第一作者:宋紫薇、封硕
● 通讯作者:李萍 (liping2004@126. com)、 李菁 ([email protected])
● 合作作者:周星辰、宋正兴
● 主要单位:中国药科大学天然药物活性组分与药效国家重点实验室、中国药科大学生命科学与技术学院、北京放射医学研究所生物学部传染病分子诊断新技术北京市重点实验室
亮 点
● 乳酸杆菌被鉴定为编码胆盐水解酶(bile salt hydrolases,BSHs)的关键菌属,并根据编码的BSH系统型将16个主要乳酸杆菌菌种分为4类;
● 编码BSH‐T3的菌株具有最高的体外解离活性;
● 给予编码BSH‐T3的罗伊氏乳酸杆菌可以改变小鼠的肝肠胆汁酸谱。
摘 要
胆盐水解酶(bile salt hydrolases,BSHs)是胆汁酸(bile acids,BAs)肝肠代谢所必需的酶。BSH催化游离BA的产生并调节BA池的动态平衡。本研究鉴定乳酸杆菌为编码BSH的关键菌属,并利用人类肠道微生物组数据库的宏基因组数据筛选获得了16个主要菌种。然后,将16种乳酸杆菌按所编码BSH系统型分为4类,其中编码BSH‐T0的有5种,编码BSH‐T2的有6种,编码BSH‐T3的有4种,同时编码BSH‐T0和BSH‐T3的只有唾液乳酸杆菌(Ligilactobacillus salivarius)。对7种结合BAs体外解离活性最高的乳酸杆菌是编码BSH‐T3的菌株。此外,在小鼠体内的研究表明,给予编码BSH-T3的罗伊氏乳酸杆菌可以改变肠道微生物组和代谢组的结构,并显著增加游离BAs和总BA排泄水平。我们的发现促进了人类肠道微生物组中编码BSH的关键乳酸杆菌的分类鉴定,并阐明了它们对BAs肝肠循环调节的贡献,这将有助于未来编码BSH的益生菌应用于治疗以改善人类健康。
视频解读
Bilibili:https://www.bilibili.com/video/BV138411U7qy/
Youtube:https://youtu.be/beBxGQ84sko
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
全文解读
引 言
胆汁酸(bile acids,BAs)最初由肝细胞中的胆固醇合成,然后与甘氨酸或牛磺酸共价结合并储存在胆囊中。胆囊中的结合BAs响应膳食摄入转移到肠道,通过肠道菌群中一系列酶的催化作用生成游离BAs和次级BAs。肠道中大约95%的BAs(包括初级和次级BAs;结合的和游离BAs)被重新吸收,通过门静脉进入肝脏,其余的随粪便排出体外。这种代谢特性能够形成一个动态的BA池并促进体内的肝肠循环。这些代谢过程通过激活回肠和肝脏中的法尼醇X受体(farnesoid X receptor, FXR)而受到负反馈调控。
肠道菌群中的胆盐水解酶(bile salt hydrolases,BSHs)负责断裂结合BAs中的酰胺键,并增加BA池的复杂性。BSHs产生的游离BAs不仅可以作为信号分子调节BAs的代谢和运输,还可以促进各种代谢活动,这些代谢活动对平衡体内糖脂代谢、胰岛素敏感性和先天免疫至关重要。肝脏BA池内稳态的破坏可能导致胆汁淤滞和其他几种肝脏疾病的发展,包括非酒精性脂肪肝、肝癌和胆管癌。BA稳态受损可导致肠易激综合征、炎症性肠病、结直肠癌等胃肠道疾病。早期对细菌中BSHs的研究主要集中在不同细菌中BSHs的鉴定和纯化、体外BSHs活性菌株的筛选以及新型BSHs的发现。近年来,由于技术的进步和对精准治疗的追求,研究开始考虑BSHs的分类和宿主代谢的功能差异。Jia等人收集了5,790个BSH同源物,并基于序列相似性网络将其划分为7个聚类。在我们之前的研究中,基于系统发育分析,我们系统地鉴定了编码BSH的菌株,并将这些BSHs分为8个系统型,在体外证明它们具有不同的酶活性。
据报道,在编码BSH的细菌中,乳酸杆菌产生了体内大部分的BSH活性。我们之前的工作提供了类似的证据,表明仅由乳酸杆菌编码的BSH‐T3具有最高的比酶活。然而,乳酸杆菌也编码属于其他系统型的BSH,这表明它们的活性存在差异。一项对170种不同乳酸杆菌中BSH的研究也推测,非同源BSH基因在体内的活性可能存在差异。因此,有必要进一步研究编码BSH的乳酸杆菌的分类和功能评价。此外,乳酸杆菌属基于全基因组测序技术和多相法被重新分类为25各菌属,其中包括修订后的乳酸杆菌属(Lactobacillus)。
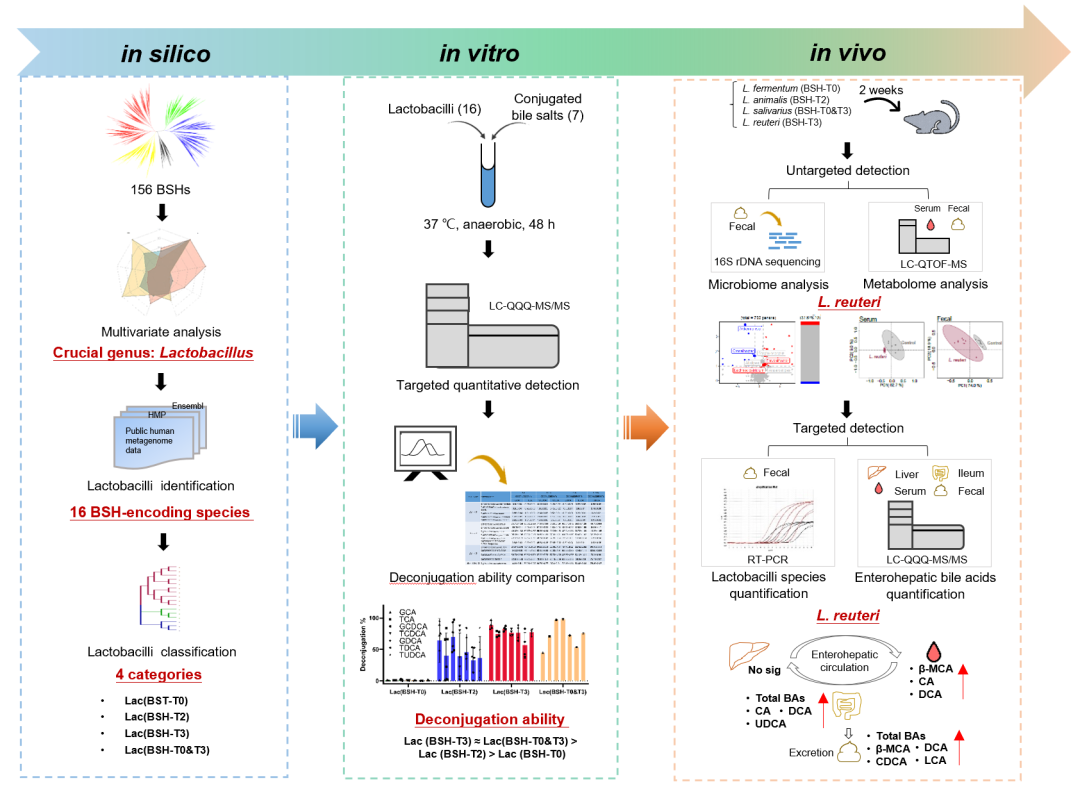
基于我们之前的发现,BSHs分布在人类微生物组的117个菌属的591个菌株中,我们对这些编码BSH的细菌进行了深入分析,以:(1)揭示人类肠道微生物群中关键的BSH编码菌属;(2)根据系统发育树对编码BSH的乳酸杆菌进行分类,并在体外评估其解离活性;(3)研究代表性乳酸杆菌菌株对小鼠肠道微生物组、代谢组和肝肠BA池结构的影响。
结 果
编码BSH的细菌特征
构建了8种BSH系统型(包括来自90个物种的156种BSH)的系统发育树,并根据先前的结果确定了它们在人类肠道微生物组中的丰度(图S1)。8种系统型中最丰富的菌属包括肠球菌属(占BSH‐T0总相对丰度的18.32%)、真杆菌属(占BSH‐T1总相对丰度的18.53%)、链球菌属(占BSH‐T2总相对丰度的43.96%)、乳酸杆菌属(在2020年乳杆菌分类重组之前)、双歧杆菌属(占BSH‐T3总相对丰度的50.97%)、拟杆菌属(分别占BSH‐T5和BSH‐T6总相对丰度的82.13%和89.89%)和布劳特氏菌属(占BSH‐T7总相对丰度的73.14%;图1A,图S1)。接着,依据丰度、多样性(香农指数)、解离能力(反应48小时后100 μM结合胆盐被BSHs水解为游离胆盐的比例,通过液相色谱-三重四级杆质谱检测)、比酶活(茚三酮法检测中BSHs每分钟从20 mM结合胆盐中释放1 μmol甘氨酸或牛磺酸的量)、编码的BSH系统型数量和相关疾病数目对这7个菌属进行多变量分析(图1B)。结果表明,编码BSH的乳酸杆菌菌株具有最高的平均解离能力(97.13%)、比酶活(124 μM min−1 mg−1)、香农指数(1.4)和系统型数量(7个系统型中的3个)。雷达图清楚地显示,乳酸杆菌是编码BSH的代表性菌属中最关键的菌属(图1C,图S2)。
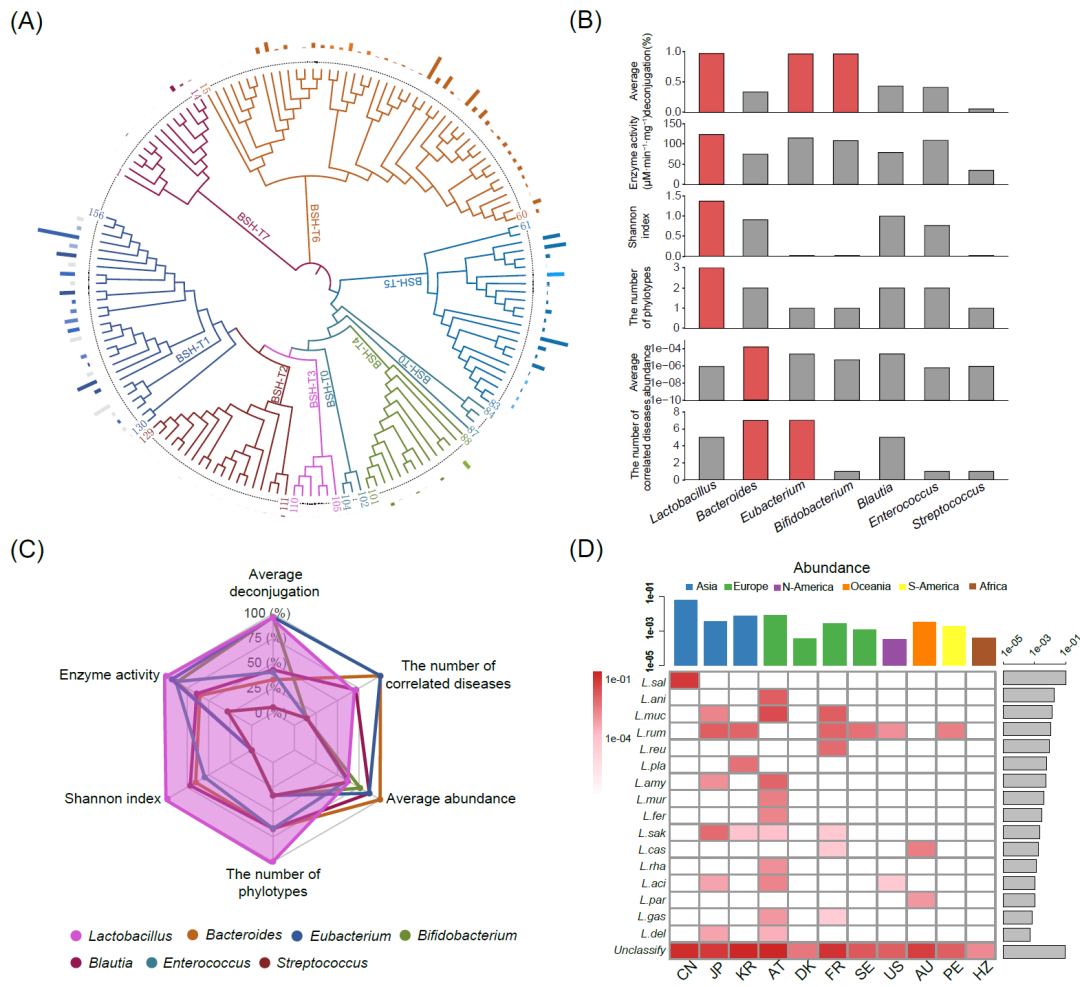
图1. 胆汁盐水解酶(bile salt hydrolase,BSH)编码菌的综合分析
(A) 156个BSHs的系统发育树,以及它们在人类微生物组中的相对丰度。相同颜色的柱表示来自同一菌属的BSHs。在同一系统型中,柱色越浅,表示该菌属编码的BSH占总相对丰度的比例越低。丰度小于5%的菌属用灰色柱表示。(B)代表性BSH编码菌属的多因素分析。多样性用香农指数来表示,解离能力用反应48 h后BSHs将100 Μm结合胆盐水解为游离胆汁酸的比例(液相色谱-三重四极杆质谱联用仪检测)来定义,比酶活性用茚三酮法测定,以BSHs每分钟从20Mm结合胆盐中释放1 μmol甘氨酸或牛磺酸的量来确定。红色柱表示相应因子分析时的最优属。(C)BSH编码菌属的雷达分析。对每个因素的数据进行了归一化处理。(D)不同人群中编码BSH乳酸杆菌菌株的相对丰度。
人类肠道菌群中编码BSH的乳酸杆菌菌种
从Ensembl数据库下载166种581株乳酸杆菌的基因组序列(表S1)。然后,根据人类宏基因组计划(Human Metagenome Project,HMP)数据库获得25种源自人类宏基因组的乳酸杆菌(表S2)。此外,从11个人群的581名健康个体的肠道微生物组数据中鉴定出17种乳酸杆菌(表S3)。以156个BSH序列和KEGG数据库中6个具有代表性的BSH序列作为参考(表S4),共从健康个体的肠道微生物组中获得16种编码BSH的乳酸杆菌,包括唾液乳杆菌、动物乳杆菌、粘膜乳杆菌、瘤胃乳杆菌、罗伊氏乳杆菌、植物乳杆菌、淀粉状乳杆菌、鼠乳杆菌、发酵乳杆菌、清酒乳杆菌、干酪乳杆菌、鼠李糖乳杆菌、嗜酸乳杆菌、副干酪乳杆菌、格氏乳杆菌、德氏乳杆菌(图1D,按相对丰度降序排序)。在上述菌种中,瘤胃乳杆菌存在于大多数人群的肠道微生物群中(11个人群中有6个,表S5)。乳酸杆菌种类最多的样本来自奥地利(AT;10 / 16,表5)。
乳酸杆菌的分类及体外解离能力的比较
16种具有代表性的编码BSH的乳酸杆菌菌株根据前期研究建立的基于系统发育的方法进行分类。这些乳酸杆菌主要编码三种BSH:BSH‐T0、BSH‐T2和BSH‐T3。发酵乳杆菌、清酒乳杆菌、干酪乳杆菌、鼠李糖乳杆菌和副干酪乳杆菌编码BSH‐T0。动物乳杆菌、粘膜乳杆菌、瘤胃乳杆菌、植物乳杆菌、鼠乳杆菌和德氏乳杆菌编码BSH‐T2。罗伊氏乳杆菌、淀粉状乳杆菌、嗜酸乳杆菌和格氏乳杆菌编码BSH‐T3。然而,特别的是,唾液乳杆菌同时编码BSH‐T0和BSH‐T3(图2A,表S6)。因此,16株乳酸杆菌可分为Lac (BSH‐T0)、Lac (BSH‐T2)、Lac (BSH‐T3)和唾液乳杆菌。
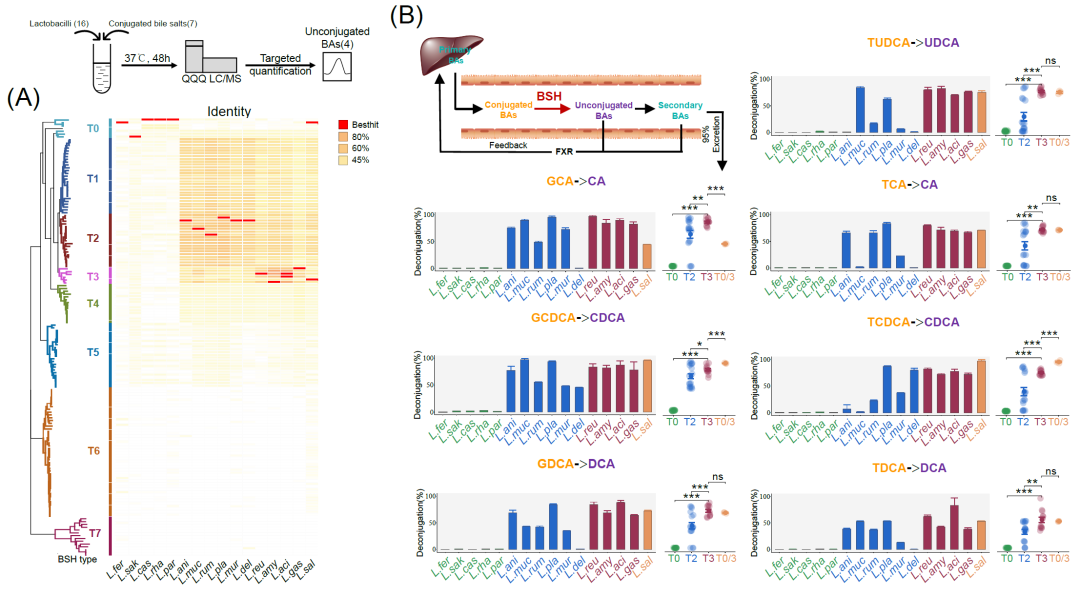
图2. 编码乳酸菌的胆汁盐水解酶(BSH)的解毒活性
(A)编码BSH的代表性乳酸杆菌菌株的分类特征(表S6)。(B)具有代表性的编码BSH的乳酸杆菌菌株的体外解离活性(表S7)。*p<0.05,**p < 0.01,***p < 0.001。在本实验中,游离胆汁酸包括CA、CDCA、DCA和UDCA。DCA和UDCA属于次级胆汁酸,也可以从CA和CDCA转化而来。CA,胆酸;CDCA,鹅去氧胆酸;DCA,脱氧胆酸;GCA,甘氨胆酸钠;GCDCA,甘氨鹅去氧胆酸钠;GDCA,甘氨脱氧胆酸钠;TCA,牛磺酸胆酸钠水合物;TCDCA,牛磺鹅去氧胆酸钠;TDCA,牛磺脱氧胆酸钠水合物;TUDCA,牛磺熊去氧胆酸钠;UDCA,熊去氧胆酸。
在水解48 h后,对相应的游离BAs进行定量检测,包括胆酸(cholic acid,CA)、鹅去氧胆酸(chenodeoxycholic acid,CDCA)、脱氧胆酸(deoxycholic acid,DCA)和熊去氧胆酸(ursodeoxycholic acid,UDCA)(图S3)。与其他结合胆盐相比,除嗜酸乳杆菌ATCC 4796(83.13%;图2B,表7)外,乳酸杆菌在水解牛磺酸脱氧胆酸钠(sodium taurodeoxycholate hydrate,TDCA)过程中普遍表现出较弱的解离能力。而除TDCA外,与Lac (BSH‐T3)菌株孵育后,约80%-100%的其他结合胆盐可转化为游离BAs,但与Lac (BSH‐T0)菌株孵育后不到3%。对于同时编码BSH‐T0和BSH‐T3的唾液乳杆菌 BNCC 138618菌株,只有对于甘氨胆酸钠水合物(sodium glycocholate hydrate,GCA)、甘氨鹅去氧胆酸钠(sodium glycochenodeoxycholate,GCDCA)和牛磺鹅去氧胆酸钠(sodium taurochnodeoxycholate,TCDCA)的解离能力与Lac (BSH‐T3)菌株有显著差异(图2B的散点图,表S7)。总的来说,乳酸杆菌菌株的解离能力可以表示为:Lac (BSH‐T3) ≈ 唾液乳杆菌 > Lac (BSH‐T2) > Lac (BSH‐T0)(图2B的散点图)。
有趣的是,分类为Lac (BSH‐T2)的菌株表现出对底物的偏好性。例如,德氏乳杆菌保加利亚亚种ATCC 11842能有效水解GCDCA(45.64%)和TCDCA(79.85%),但对其他胆盐的解离能力小于1%(柱状图见图2B,表S7)。鼠乳杆菌DSM 13345对GCA(89.76%)、 GCDCA(97.34%)和牛磺熊去氧胆酸钠(TUDCA;84.46%)具有较强的解离能力,但对牛磺酸胆酸钠(TCA;2.18%)和TCDCA解离能力较差(1.83%;柱状图见图2B,表S7)。
给予乳酸杆菌改变了小鼠的肠道微生物群
为了进一步探索乳酸杆菌在体内的作用,我们将编码BSH‐T0的发酵乳杆菌 BNCC 194390、编码BSH‐T2的动物乳杆菌BNCC 134981、编码BSHT3的罗伊氏乳杆菌 ATCC 27755和同时编码BSHT0和BSH‐T3的唾液乳杆菌 BNCC 138618共 4株乳酸杆菌(每种乳酸杆菌类别中相对丰度最高的菌种)给雄性C57BL/6J小鼠灌胃2周。
给药2周后用小鼠粪便基因组DNA进行16S rDNA测序。与对照组相比,只有罗伊氏乳杆菌给药组的α多样性水平显著高于对照组(Wilcoxon秩和检验;p < 0.05,图3A)。扩增子测序数据也显示了各组微生物结构的不同(置换多元方差分析;R2 = 0.2599, p = 0.001)。除动物乳杆菌组外,其他乳杆菌组与对照组有显著差异(图3B)。此外,与对照组(B:F = 3.4)相比,所有乳杆菌组拟杆菌门与厚壁菌门的比例(B:F)都有所下降,其中罗伊氏乳杆菌给药组下降幅度最大(B:F = 2.2,图S4A)。
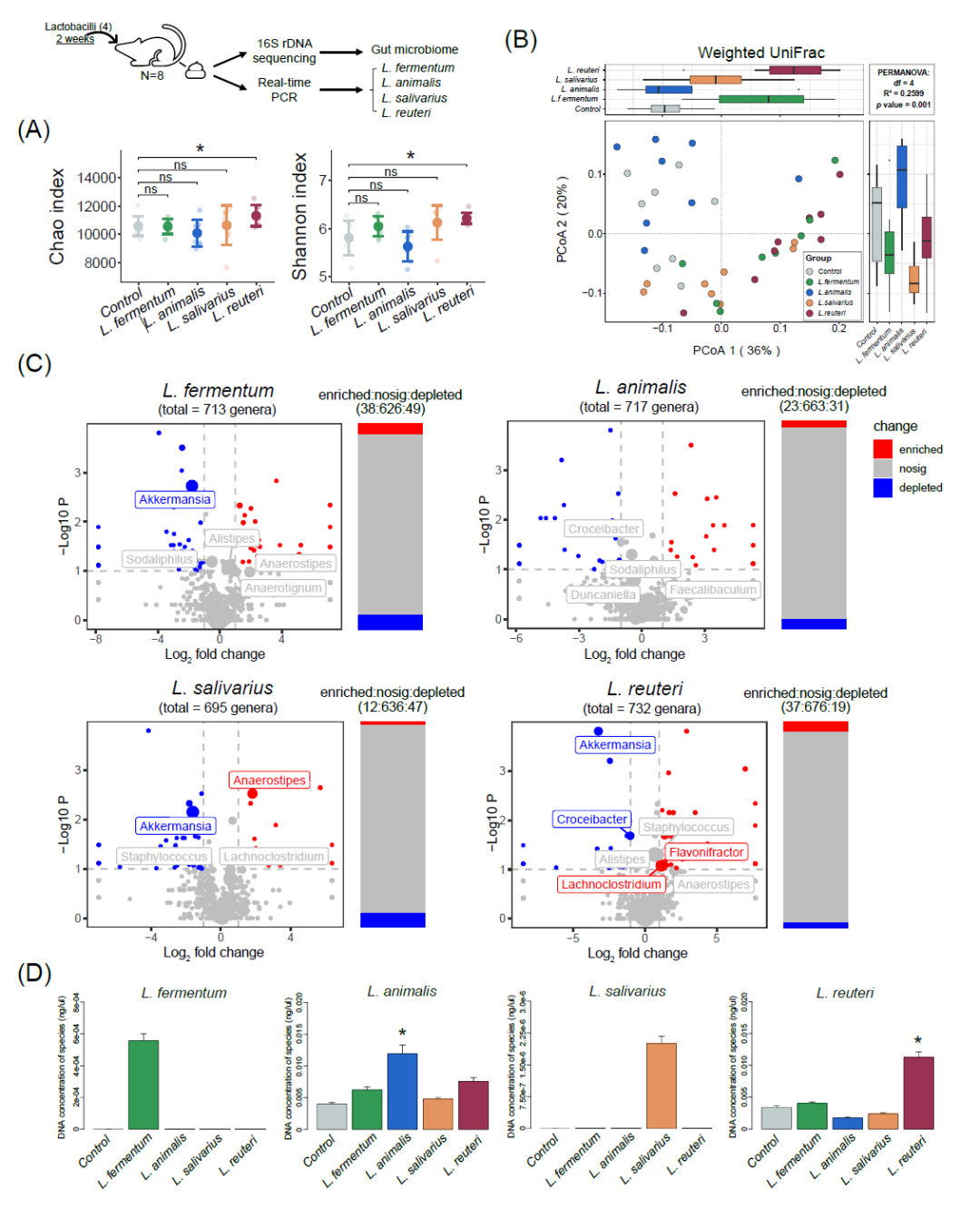
图3. 乳酸菌处理改变了小鼠粪便微生物群的结构
(A)粪便微生物群落的α多样性(Chao丰富度和Shannon多样性指数)。(B)基于WUniFrac的粪便微生物组PCoA。β多样性的统计学显著性采用PERMANOVA;p < 0.05为差异有统计学意义。(C)火山图说明了粪便微生物群的属富集分析。采用Wilcoxon秩和检验进行分析。标记代表前15个属,标记和点的大小与每组中平均属的丰度成正比。(D)不同组小鼠粪便微生物群中发酵乳酸杆菌、动物乳酸杆菌、唾液乳酸杆菌和罗伊氏乳酸杆菌四种乳酸杆菌的浓度。PCoA,主坐标分析。*p < 0.05, **p < 0.01, ***p < 0.001。
通过绝对定量分析(见材料和方法部分),发现罗伊氏乳杆菌给药组显示出最高富集和最少减少的属(|log2(fold change)| > 1,−log10(p值)> 1)。值得注意的是,在前15个属中,Lachnoclostridium(平均丰度 = 0.0699;log2(fold change) = 1.17)和Flavonifractor(平均丰度= 0.0180;log2(fold change) = 1.05)在罗伊氏乳杆菌给药组中被发现富集(图3C)。此外,在罗伊氏乳杆菌给药组中,观察到另枝菌属和葡萄球菌属的相对丰度显著增加(图S4B)。此外,与对照组相比,除了动物乳杆菌组外,所有乳杆菌组的阿克曼菌丰度都显著降低。因此,给予编码BSH的乳酸杆菌,特别是罗伊氏乳杆菌,改变了小鼠肠道微生物群的结构。
尽管给予了乳酸杆菌,但各组间乳酸杆菌属的丰度没有显著差异(图S4B)。因此,我们设计了四种乳酸杆菌的特异性引物,用于在小鼠粪便中靶向定量(图S5,表S8)。与对照组相比,动物乳杆菌和罗伊氏乳杆菌的相对丰度显著增加(图3D)。由于发酵乳杆菌和唾液乳杆菌不是小鼠肠道微生物群的原生物种,因此除了给予这两种乳酸杆菌菌株的组外,在任何组中都没有观察到信号(图3D)。上述结果表明,四种乳酸杆菌在小鼠体内的浓度显著增加。
给予乳酸杆菌改变了小鼠的血清和粪便代谢组
然后我们研究了乳酸杆菌对小鼠代谢组的影响。在血清和粪便中分别检测到573和1630个差异离子。通过血清样本获得的总离子色谱分析显示,与对照组相比,罗伊氏乳杆菌给药组显示出最多的差异离子(图4A,表S9)。各组差异离子的主成分分析显示,发酵乳杆菌和罗伊氏乳杆菌给药组小鼠的血清代谢物与对照组存在显著差异(图4B)。值得注意的是,乳酸杆菌处理改变了血清中50种代谢物(上调),而罗伊氏乳杆菌处理导致了其中49种代谢物改变(图4C,表S10)。上调的代谢物主要是脂质和类脂分子,包括27-去胆固醇内己醇(一种胆固醇代谢的中间体)和一些肉碱代谢物,如L‐乙酰肉碱(表S10)。在粪便中,罗伊氏乳杆菌给药组仍然具有最多的差异离子,并且是唯一与对照组有显著差异的组(图4D,E,表S9)。此外,乳酸杆菌处理改变了粪便中的33种代谢物(24种上调,9种下调),而罗伊氏乳杆菌给药组显示22种显著差异代谢物(13种上调,9种下调)(图4F,表S10)。上调的代谢物主要是脂质分子、有机酸及其衍生物。下调的代谢物包括一些类固醇衍生物,如3‐氧‐4,6‐胆酸(表S10)。这些结果表明,给予罗伊氏乳杆菌对小鼠的血清和粪便代谢组有显著影响,特别是在促进脂质和胆固醇代谢方面,这可能在预防脂肪堆积方面发挥了潜在的作用。

图4. 乳酸杆菌处理改变了小鼠的血清和粪便代谢组
(A)血清中差异代谢离子富集分析热图。红色表示给药乳酸菌组的差异离子相对浓度较高,蓝色表示对照组的差异离子相对浓度较高;灰色表示该差异离子的相对浓度在两组之间没有差异。(B)基于血清差异代谢物(VIP > 1, pFDR≤0.05)的主成分分析评分图。(C)血清中差异代谢物(pFDR≤0.05)热图。(D)粪便中差异代谢离子富集分析热图。(E)基于粪便差异代谢物(VIP > 1, pFDR≤0.05)的主成分分析评分图。(F)粪便中差异代谢物(pFDR≤0.05)热图。星号代表罗伊氏乳杆菌组的代谢物差异。带正电的离子以ESI+模式收集,带负电的离子以ESI−模式收集。
乳酸杆菌对小鼠肝肠BA池的调节作用
为了确定编码不同BSH系统型的乳酸杆菌对肝肠BA谱的影响,我们在小鼠的肝脏、血液、回肠内容物和粪便中对BA进行了靶向定量检测(图S6-S9,表S11)。结果显示,肝脏BA池变化较小,只有发酵乳杆菌给药组β-鼠胆酸(β -MCA)和CDCA浓度升高(图5A,图S10A)。肝脏中总BA水平和游离/结合BA比值均无显著差异(图5B,C)。在血清中,给予罗伊氏乳杆菌导致β-MCA、CA和DCA浓度显著增加,总BA水平和游离BA百分比呈上升趋势(图5D-F)。在回肠远端内容物和粪便排泄中,给予罗伊氏乳杆菌后,BA谱和总BA水平都发生了显著变化(图5H,K)。具体来说,在罗伊氏乳杆菌给药组,回肠远端内容物中CA、DCA和UDCA的浓度以及粪便中β-MCA、CDCA、DCA和石胆酸(lithocholic acid,LCA)的浓度都显著增加(图5G,J)。然而,罗伊氏乳杆菌给药组粪便中游离/结合BA比例增加(图5L),但回肠远端内容物中BA比例下降(图5I),这可能是由于结合BA水平增加(图S10)。这些结果表明,乳酸杆菌菌株是肝肠BA代谢的多效调节剂。罗伊氏乳杆菌给药小鼠游离BA水平的显著升高和BA谱的变化进一步证实了编码BSH‐T3的乳酸杆菌是BA代谢的重要调节剂。
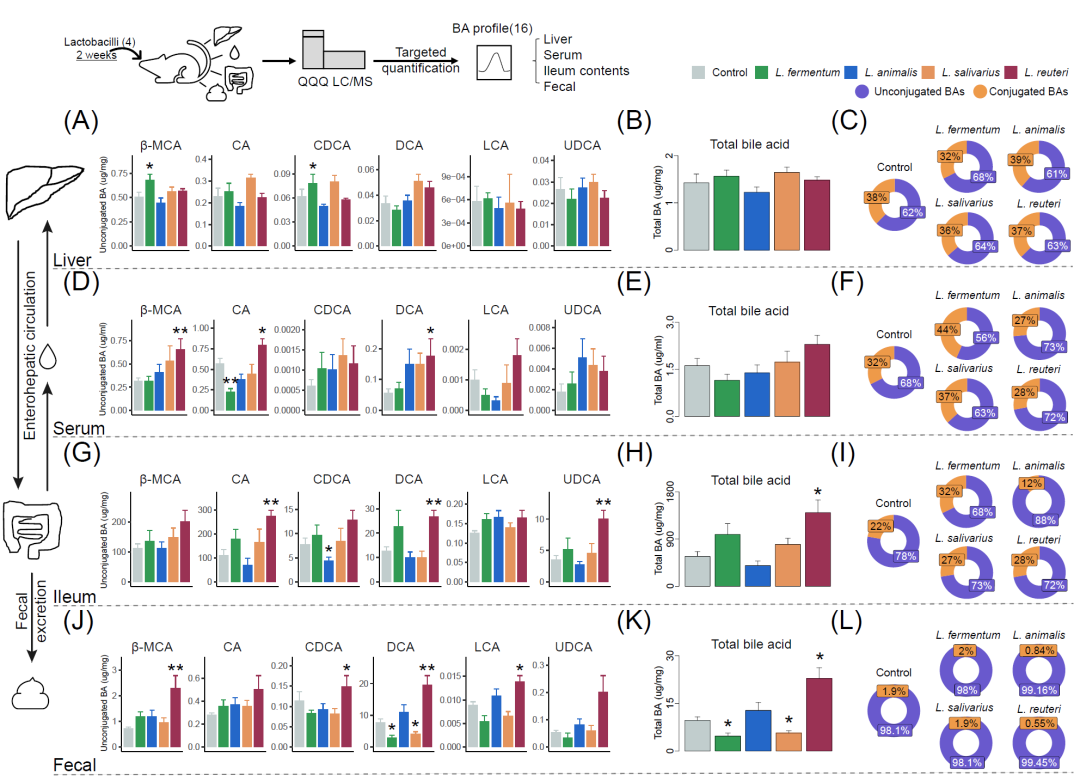
图5. 乳酸杆菌处理改变了小鼠胆汁酸池的组成
不同组小鼠肝脏中(A)游离BAs;(B)总BA;(C)游离BAs和结合BAs的比例。不同组小鼠血清中(D)游离BAs;(E)总BA;(F)游离BAs和偶联BAs的比例。不同组小鼠回肠远端内容物中(G)游离BAs;(H)总BA;(I)游离BAs和偶联BAs的比例。不同组小鼠粪便样品中(J)游离BAs;(K)总BA;(L)游离BAs和偶联BAs的比例。使用配对t检验确定两组之间的统计学显著差异。*p < 0.05, **p < 0.01, ***p < 0.001。游离BAs包括CA、CDCA、DCA和UDCA。结合BAs包括GCA、GCDCA、GDCA、TCA、TCDCA、TDCA和TUDCA。总BA是检测到的所有BAs(包括游离BAs和结合BAs)的总和。CA,胆酸;CDCA,鹅去氧胆酸;DCA,脱氧胆酸;LCA,石胆酸;UDCA:熊去氧胆酸;β-MCA,β-鼠胆酸。
讨 论
先前的一项研究首次使用功能驱动的宏基因组学来鉴定微生物群落中的BSHs,并基于环境来源对其进行聚类,但样本量较小。我们之前的工作揭示了基于大量人类肠道微生物组数据的BSHs分类和丰度分析,并通过将目标序列分类为特定的系统型来评估BSH活性。基于之前的工作,本研究通过多元分析和广泛的宏基因组数据挖掘,全面评估了编码BSH的肠道细菌,发现了编码BSH的关键乳酸杆菌菌种。与其他编码BSH的属相比,报道最多的就是乳酸杆菌(图S11,表S12),包括格氏乳杆菌、罗伊氏乳杆菌、唾液乳杆菌等。先前的一项研究报道了大约39种编码BSH的乳酸杆菌,但包括一些不是肠道原生的物种,如敏捷乳杆菌和阴道粘液乳杆菌,它们通常分别存在于唾液和阴道中。本研究利用健康人类肠道宏基因组,系统鉴定了16种编码BSH的乳酸杆菌,并根据所编码BSH的系统型进行了分类。而如前所述,根据核心基因组系统发育、成对平均氨基酸特征、进化支特异性特征基因、生理标准和生物生态学对乳杆菌属进行了重新分类。但基于BSH型的乳酸杆菌分类似乎与新的分类学没有足够的相关性。具体来说,虽然有三种Lacticaseibacillus菌株都编码BSH‐T0,但属于同一新乳酸菌属的发酵乳酸杆菌和罗伊氏乳杆菌分别编码BSH‐T0和BSH‐T3。动物乳杆菌、瘤胃乳杆菌和鼠乳杆菌属于Lac (BSH‐T2),而同属于这个新乳酸菌属的唾液乳杆菌不编码BSH‐T2(图2A)。
Jimmy等为了检测乳酸杆菌菌株的体外BSH活性,比较了5种乳酸杆菌对4种BAs的解离活性,发现植物乳杆菌DGIA1具有出色的解离能力。相反,我们的研究系统地比较了16种BSH系统型分类的乳酸杆菌菌株对7种结合BAs的解离活性,发现编码BSH‐T3的乳酸杆菌菌株,包括罗伊氏乳杆菌ATCC 27755、淀粉乳杆菌BNCC 135321、嗜酸乳杆菌 ATCC 4796和格氏乳杆菌 BNCC 135057,具有最高的解离活性。然而,值得注意的是,一些BSH-T2编码菌株表现出对胆盐底物的偏好(图2B,表S7)。同样,Matthew等也发现嗜酸乳杆菌NCK1909和产酸乳杆菌NCK2253编码的BSHs具有底物偏好,并可能影响菌株的生长。并且,他们最近在结构上确定了一个环,预测BSH对甘氨酸或牛磺酸底物的偏好。具体来说,牛磺酸偏好BSHs只含有Gly‐Val/Thr‐Gly序列,而甘氨酸偏好酶只含有Ser‐Arg‐Gly/Ser序列。另外有一篇综述总结了已报道的底物特异性BSH,并根据底物偏好对其进行分类,并探索了可能参与决定底物偏好的关键残基和二级结构元件。还有一项研究发现,唾液乳杆菌偏爱牛磺酸结合底物,主要是因为其去羟基化的甾醇环易于水解,并表明Asp19、Asn79、Asn171、Arg224、Gln257、Asn262和Glu270对底物的氨基酸头结合的重要性。然而,本研究中编码BSH‐T2的乳酸杆菌对底物的偏好更为复杂,不能完全分为偏好甘氨酸或偏好牛磺酸的乳酸杆菌。我们推测这些特性可能与不同BSH-T2的结构有关,需要进一步深入研究BSH-T2与特定底物的结构分析以及全面的氨基酸突变诱导,以了解底物偏好的基础。
与人类相比,小鼠肠道细菌可以产生更多样化的次级BAs。此外,甘氨酸结合BAs在人中占主导地位,而牛磺结合BAs在小鼠中占主导地位,这与我们的结果中肝肠甘氨酸BAs的低浓度是对应的(图5,图S10)。虽然模型动物和人类的肠道微生物群和BAs的组成差异很大,但在小鼠体内的研究仍然是该领域不可或缺的(图S12,表S13)。然而,大多数研究都集中在证明单一乳酸杆菌菌株对BA代谢的调节。例如,据报道,植物乳杆菌 CCFM8661可显著增加小鼠肝脏BA合成、胆汁和胆道谷胱甘肽输出以及粪便BA排泄。补充鼠李糖乳杆菌 GG可以抑制BA合成,降低肝脏BA,增加BA排泄。我们的研究检测了编码不同BSH种型的四种乳酸杆菌菌株对小鼠肝肠BA谱的影响,发现编码BSH‐T3的罗伊氏乳杆菌ATCC 27755显著改变了小鼠肠道微生物组和代谢组的结构,改变肝肠BA谱和增加BA排泄的良好能力。此外,一些研究表明,高脂肪摄入可能通过改变BA谱引发慢性疾病。对小鼠代谢组的分析显示,给予BSH编码的乳酸杆菌改变了许多与脂质和固醇代谢相关的代谢物(图4),这表明可能对与高脂肪饮食相关的代谢性疾病有益。
我们之前的研究也报道过BSHs 的旁系同源。据报道,发酵乳杆菌MTCC 8711编码2个bsh基因,植物乳杆菌WCFS1编码4个bsh基因。本研究中的一些乳酸杆菌菌株也编码了多个bsh基因,包括嗜酸乳杆菌ATCC 4796编码了两个属于BSH‐T3的基因,淀粉乳杆菌BNCC 135321编码了三个属于BSH‐T3的基因,副干酪乳杆菌BNCC 189800编码了两个属于BSH‐T0的基因,唾液乳杆菌BNCC 138618分别编码了属于BSH‐T0和BSH‐T3的基因(表S6)。据我们所知,很少有研究调查编码BSH旁系同源的细菌对BAs代谢功能的影响。有趣的是,我们的研究结果表明,尽管唾液乳杆菌 BNCC 138618在体外具有与Lac (BSH‐T3)菌株相当的解离能力,但在体内研究中,给予唾液乳杆菌 BNCC 138618对肝肠BA池没有显著影响。因此,仍然需要进一步的机理研究。
本研究的主要不足是:(1)所使用的公开的人类肠道宏基因组数据可能会对乳酸杆菌属和所选菌种产生偏差;(2)乳酸杆菌菌株调节BA代谢的机制需要通过适当的体外和体内模型进一步探索。我们将继续收集宏基因组数据,为进一步的基因组分析提供更好的数据集,并尝试构建bsh敲除乳酸杆菌和BA代谢相关的疾病动物模型,探索乳酸杆菌在疾病治疗中的临床应用可能性。
材料与方法
序列数据收集
从Ensembl数据库(https://asia.ensembl.org/)下载乳酸杆菌基因组序列。从HMP数据库(https:// www.hmpdacc.org/)获取参考基因组,对乳酸杆菌基因组中的基因和蛋白质进行预测和分类。元基因组序列数据来源于以往研究构建的数据集,其中包括来自6大洲11个人群的581名健康个体的数据,包括非洲的坦桑尼亚(HZ),亚洲的中国(CN)、日本(JP)和韩国(KR),欧洲的奥地利(AT)、丹麦(DK)、法国(FR)和瑞典(SE),大洋洲的澳大利亚(AU),北美洲的美国(US)和南美洲的秘鲁(PE)。使用BLASTP (版本 2.2.29+)对氨基酸序列进行配对比对。
系统发育树
使用ClustalW对BSH蛋白序列进行比对,并在MEGA软件(版本7.0)中使用最大似然法(Jones-Taylor-Thornton模型)构建系统发育树。使用Interactive Tree Of Life (iTOL,https://itoL.embL.de/)将156个BSH序列的丰度数据导入系统发育树,并根据需要填充颜色。使用Dendroscope(版本3.4.7)通过调整标签和填充颜色来修饰系统发育树。
材料和菌株
购买水合甘氨胆酸钠(CAS: 863‐57‐0)、甘氨鹅去氧胆酸钠(CAS: 16564‐43‐5)、甘氨脱氧胆酸钠(CAS: 16409‐34‐0)、牛磺胆酸钠(CAS: 345909‐26‐4)、牛磺鹅去氧胆酸钠(CAS: 6009‐98‐9)、牛磺脱氧胆酸钠(CAS: 207737‐97‐1)、牛磺熊去氧胆酸钠(CAS: 35807‐85‐3)用于在体外对乳酸杆菌的解离水平进行靶向定量。GCA、GCDCA、GDCA、GLCA、GUDCA、TCA、TCDCA、TDCA、TLCA、TUDCA、CA、CDCA、DCA、LCA、UDCA、β-MCA等标准品及相应的同位素标准品用于定量分析。16株乳酸杆菌用于BA解离试验及动物实验(表S6)。
乳酸杆菌解离结合胆盐
将乳酸杆菌菌株在新鲜制备的MRS培养基中稀释至对数生长期(OD600nm = 0.1, 107 CFU/mL)水平。向菌株培养液中加入结合胆盐(终浓度为4 μM)。根据先前的工作,我们对16株乳酸杆菌的生长进行了监测,确认所有菌株在48 h后均达到生长稳定期。所以,将菌株和结合胆盐培养物在37℃厌氧室中培养48 h。
动物研究
购入8周龄C57BL/6J雄性小鼠(20-22 g)。试验前让小鼠适应环境1周,自由进食和饮水,然后随机分为不同组,饲喂标准饲料。收集孵育48 h后的乳酸杆菌培养物,洗涤后用无菌磷酸盐缓冲盐水(PBS)稀释至5 × 109 CFU/mL(200 μL /只小鼠/天)灌胃。最初每组10只小鼠,但考虑到个体小鼠的代谢存在较大差异,我们将每组总BA浓度最高和最低的小鼠排除在外,这些样本也被排除在肠道微生物组和代谢组的分析之外。按最小试验要求选择各组大小,即每组8只。
16S rDNA测序及数据分析
每天给予乳酸杆菌2周后,收集每只小鼠的粪便样本。使用试剂盒提取基因组DNA,并加入外源菌株(Sporosarcina pasteurii ATCC 11859)的DNA(1%),以进行绝对定量测序。然后,使用30至40 ng的DNA混合物来产生扩增子。选取16S rDNA的V3和V4高变区片段(F:5′‐ACTCCTACGGGAGGCAGCA‐3′;R:5′‐GGACTACHVGGGTWTCTAAT‐3′)进行扩增和DNA文库构建,使用Illumina NovaSeq进行测序。
采用双端测序,并通过鉴定外部标准菌株计算绝对丰度水平。序列数据分析主要采用QIIME 2进行。采用R(版本3.3.2)进行统计分析。为了比较样本间菌属的丰富度和均匀度,利用Chao和Shannon指数可视化不同类群间的α多样性值。Beta多样性是基于加权UniFrac(WUniFrac)距离矩阵,使用主坐标分析来测量。根据总菌属的绝对丰度对菌属进行富集分析。根据以下标准筛选富集属:log2(fold change) > 1和- log10(p值)> 1。同样,使用以下标准筛选减少的属:log2(fold change)< - 1和- log10(p值)> 1。
小鼠粪便中乳酸杆菌菌种的定量检测
采用AceQ qPCR SYBR Green Master Mix试剂盒通过QPCR检测乳酸杆菌菌种含量。用于细菌扩增的引物集合见表S8。用对应的乳酸杆菌菌株稀释后的DNA构建标准曲线。
BAs的测定
采用安捷伦1290液相色谱仪联用安捷伦 6470三联四极杆质谱仪定量检测细菌培养物及小鼠血清、肝脏、回肠内容物和粪便中的BAs,包括GCA、GCDCA、GDCA、GLCA、GUDCA、TCA、TDCA、TLCA、TUDCA、CA、CDCA、DCA、LCA、UDCA和β - MCA(仅在小鼠中)。CA‐d4、GCDCA‐d4、GDCA‐d4、GLCA‐d4、GUDCA‐d4、TCA‐d4、TDCA‐d4、TLCA‐d4、TUDCA‐d4、CA‐d4、CDCA‐d4、DCA‐d4、LCA‐d4、UDCA‐d4、β‐MCA‐d5作为内标。采用多种反应监测模式在负离子模式下对BAs进行检测。使用安捷伦MassHunter软件(版本B.08.00)进行仪器控制和数据采集。值得注意的是,由于其浓度低于检测限,未对小鼠血清和肝脏中的GLCA进行定量分析。样品制备方法和详细的色谱条件在附加信息中描述。
代谢组检测
非靶向代谢组检测使用安捷伦1290 液相系统与安捷伦6545四极杆飞行时间质谱仪进行,该质谱仪配备电喷雾电离源,可在正离子和负离子模式下工作。用于非靶向代谢组学分析的血清和粪便的处理方法与靶向分析类似,只是同位素标准工作溶液被0.1 μg/mL L‐2‐氯苯丙氨酸和1 μg/mL酮洛芬代替并混合。从每个血清和粪便匀浆液样品的混合等分中获得QC样品,并使用相同的程序进行预处理。使用安捷伦 MassHunter软件(版本B.08.00)进行仪器控制和数据采集,运行R中的XCMS包进行数据预处理,包括峰检测判别、基线校正、非线性保留时间对齐。通过检索人类代谢组数据库,根据准确的质量和MS/MS片段进行差异代谢物特征鉴定。详细的色谱条件在附加信息中描述。
统计分析
所有数值均以平均值±SEM表示。使用Wilcoxon秩和检验检测两组之间的统计学显著差异,随后进行FDR校正。相关分析采用Spearman系数。采用线性回归模型进行多变量调整分析,进行多重比较。所有分析均使用R(版本3.3.2)进行,p < 0.05被认为具有统计学意义。数据采用ImageGP进行可视化处理。应用OPLS - DA来确定组间代谢表型的差异。在OPLS - DA模型中,调整后的p值(pFDR)< 0.05和VIP值> 1.0的代谢特征被定义为差异代谢物。
引文格式:
Ziwei Song, Shuo Feng, Xingchen Zhou, Zhengxing Song, Jing Li, Ping Li. 2023. “Taxonomic identification of bile salt hydrolase-encoding lactobacilli: Modulation of the enterohepatic bile acid profile.” iMeta. e128. https://doi.org/10.1002/imt2.128
作者简介

宋紫薇(第一作者)
● 中国药科大学生药学博士研究生。
● 研究方向:胆汁酸的肠道代谢,细菌抗性基因的分布,中药多糖与肠道菌群互作。

封硕(第一作者)
● 中国药科大学药物生物信息学博士研究生。
● 研究方向:药物与肠道菌群的相互作用。

李萍(通讯作者)
● 中国药科大学教授,天然药物活性组分与药效国家重点实验室主任。
● 江苏省教学名师,现任教育部“长江学者”特聘教授,天然药物活性组分与药效国家重点实验室主任。国家自然科学基金委创新研究群体学术带头人、教育部创新团队带头人、国家杰出青年基金获得者、江苏省第四期“333高层次人才培养工程”第一层次培养对象人选,《生药学》国家重点学科首席学科带头人、全国优秀科技工作者、全国优秀博士学位论文指导教师。担任Journal of Chromatography B、Journal of Pharmaceutical and Biomedical Analysis、Evidence-Based Complementary and Alternative Medicine、中国天然药物、药学学报等杂志编委。兼任中国人民政治协商会议第十二届全国委员会委员、国家重大新药创制责任专家和项目评审专家、国家自然科学基金委评审专家、国家科技奖评审专家、国家药典委员会委员、美国USP-HMC东亚专家组成员等职。

李菁(通讯作者)
● 中国药科大学教授。
● 江苏省“青蓝工程”中青年学术带头人,中国生物物理学会肠道菌群分会理事,中国药理学会药物基因组学专业委员会委员。现承担国家自然科学基金委多项研究基金,并为国家自然科学基金委项目评审人和PloS Genetics等国际著名杂志的审稿人。主要研究方向为“群体药物基因组学”和“微生物宏基因组学”的交叉领域,即通过新一代DNA高通量测序技术从不同人群的遗传多样性、环境适应机制、和人体伴生微生物菌群结构等多方面探索人体药物基因组学和微生物宏基因组学的基本机制和机理。
更多推荐
(▼ 点击跳转)
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
iMeta | 浙大倪艳组MetOrigin实现代谢物溯源和肠道微生物组与代谢组整合分析
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF > 15)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、前3年免出版费、50万用户的社交媒体宣传等。2022年2月正式创刊发行!
联系我们
iMeta主页:http://www.imeta.science
出版社:https://onlinelibrary.wiley.com/journal/2770596x
投稿:https://mc.manuscriptcentral.com/imeta
邮箱:[email protected]