学习《scikit-learn机器学习》时的一些实践。
常用参数
参数C
SVM分类器svm.SVC()中的参数C即SVM所优化的目标函数
中,松弛系数
求和项的系数
。
松弛系数 表征了数据样本 违反最大间距规则的程度。对大部分满足约束条件的样本,其松弛系数 为0;而对不满足约束条件的样本,其松弛系数 是大于0的。
所以松弛系数的求和项系数 就是对违反最大间距规则的样本的惩罚力度,惩罚越大越不能容忍有样本不满足约束条件。这一项类似于Logistic回归中引入正则化项,都是为了减少overfitting。
参数degree
在多项式核中使用,表示使用的多项式的阶数。
参数gamma
官方文档里的原话是:
Kernel coefficient for ‘rbf’, ‘poly’ and ‘sigmoid’. If gamma is ‘auto’ then 1/n_features will be used instead.
对于多项式核,它是多项式核函数
中的
。
对于高斯核,它是径向基函数
中的
。
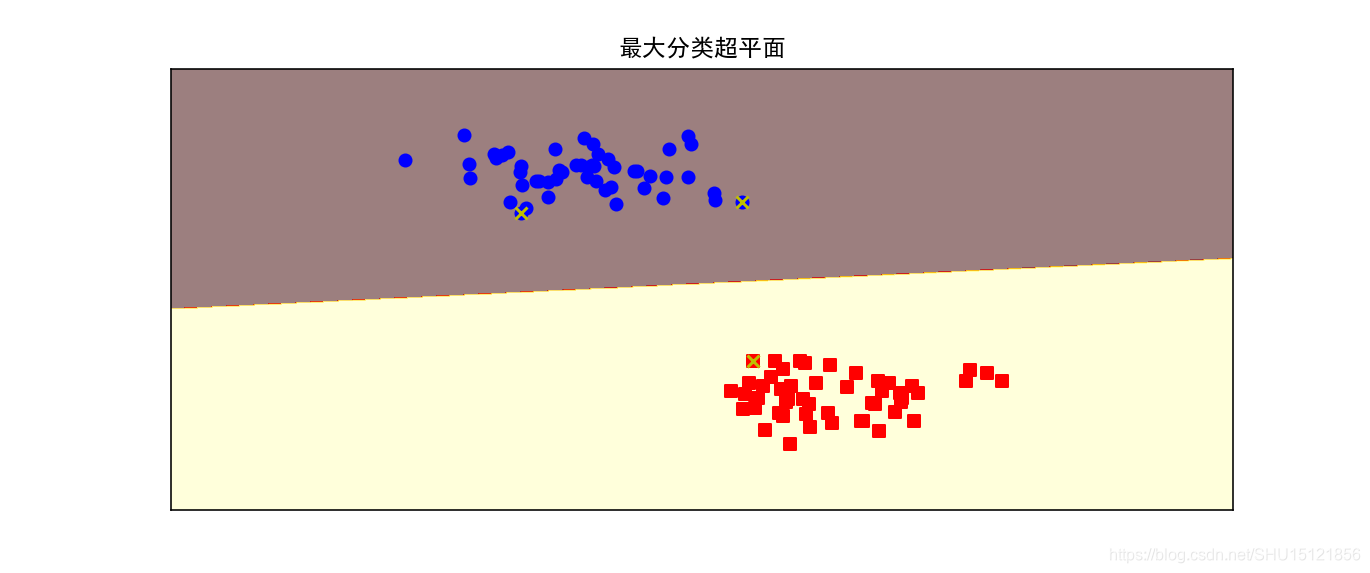
绘制最大分类超平面
打x号的点是支持向量。
from sklearn import svm
from sklearn.datasets import make_blobs
from matplotlib import pyplot as plt
import numpy as np
# 绘制分类超平面,其中X是有两个特征的,所以实际绘制出来就是在平面上的不同类别区域用不同颜色标记
# 这里h是采样步长,draw_sv指示是否绘制支持向量
def plot_hyperplane(clf, X, y,
h=0.02,
draw_sv=True,
title='hyperplan'):
# 绘制的区间,x轴和y轴都从最小值-1到最大值+1
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
# 使用np.meshgrid()扩充为两轴的所有可能取值的组合
xx, yy = np.meshgrid(np.arange(x_min, x_max, h),
np.arange(y_min, y_max, h))
plt.title(title)
plt.xlim(xx.min(), xx.max())
plt.ylim(yy.min(), yy.max())
plt.xticks(())
plt.yticks(())
# np.ravel()将采样点的xy坐标摊平,使用np.c_将其按列组合成[[x y][x y]...]的坐标点形式
Z = clf.predict(np.c_[xx.ravel(), yy.ravel()])
# 用绘制等高线图的方式来绘制不同类别为不同颜色(等高线图上同一高度为同一颜色)
Z = Z.reshape(xx.shape)
plt.contourf(xx, yy, Z, cmap='hot', alpha=0.5)
# 不同类(标签)展示在图上的的标记和颜色
markers = ['o', 's', '^']
colors = ['b', 'r', 'c']
# 可能的类(标签)取值
labels = np.unique(y)
# 对于每种标签取值
for label in labels:
# 绘制相应的样本点,使用自己的标记和颜色
plt.scatter(X[y == label][:, 0],
X[y == label][:, 1],
c=colors[label],
marker=markers[label])
# 绘制支持向量
if draw_sv:
# 用该方式可以直接取出支持向量是哪些点
sv = clf.support_vectors_
# 绘制为白色'x',这样就会贴在之前的有色点上了
plt.scatter(sv[:, 0], sv[:, 1], c='y', marker='x')
if __name__ == '__main__':
# 生成聚类样本100个,特征数为2(默认n_features=2),类别数为2,标准差0.3,随机种子设为0
X, y = make_blobs(n_samples=100, centers=2, random_state=0, cluster_std=0.3)
# print(X,y)
clf = svm.SVC(kernel='linear', C=1.0)
clf.fit(X, y)
plt.figure(figsize=(12, 4), dpi=144)
plot_hyperplane(clf, X, y, h=0.01, title="最大分类超平面")
plt.show()
运行结果:
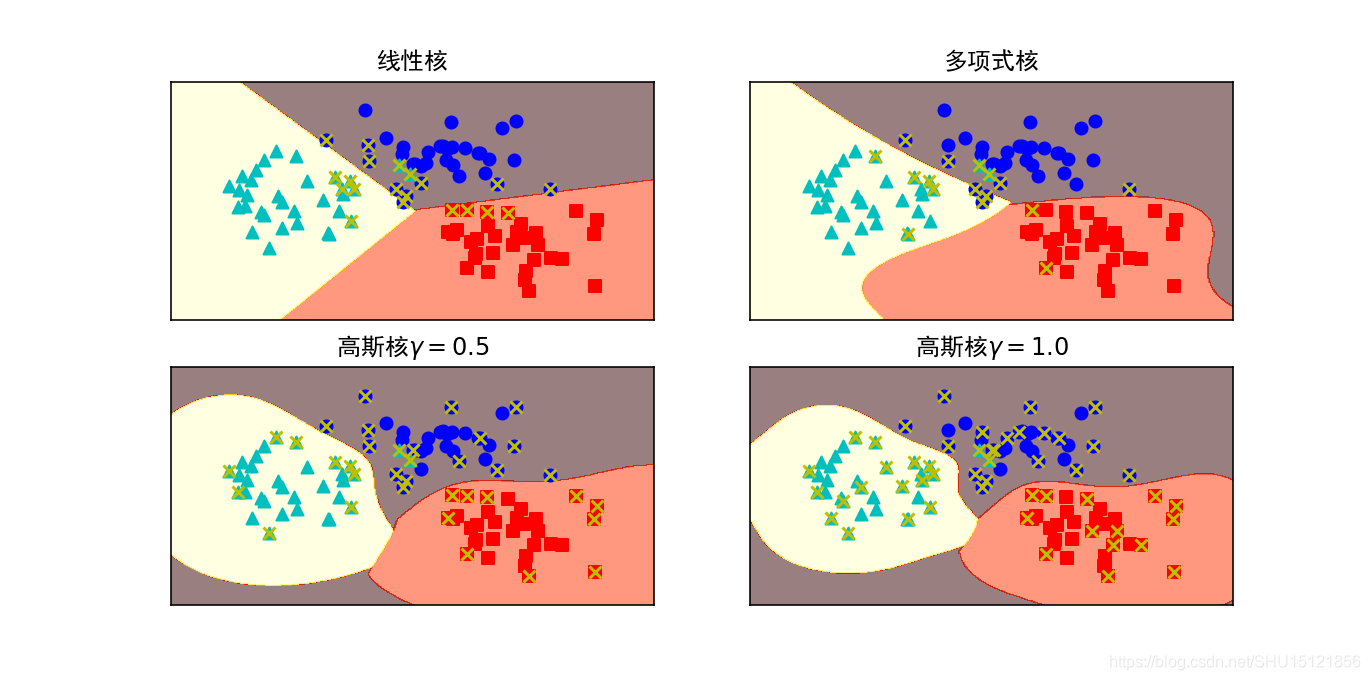
比较三种核SVM的分类面
from sklearn import svm
from sklearn.datasets import make_blobs
from matplotlib import pyplot as plt
from z8.svc2 import plot_hyperplane
X, y = make_blobs(n_samples=100, centers=3, random_state=0, cluster_std=0.8)
# 线性核
clf_linear = svm.SVC(C=1.0, kernel='linear')
# 多项式核
clf_poly = svm.SVC(C=1.0, kernel='poly', degree=3)
# 高斯核(RBF核)
clf_rbf = svm.SVC(C=1.0, kernel='rbf', gamma=0.5)
clf_rbf2 = svm.SVC(C=1.0, kernel='rbf', gamma=1.0)
plt.figure(figsize=(10, 10), dpi=144)
clfs = [clf_linear, clf_poly, clf_rbf, clf_rbf2]
titles = ["线性核", "多项式核", "高斯核$\gamma=0.5$", "高斯核$\gamma=1.0$"]
# 分别训练模型并绘制分类面.注意这里用zip组成一个个元组组成的对象
for clf, i in zip(clfs, range(len(titles))):
clf.fit(X, y)
plt.subplot(2, 2, i + 1)
plot_hyperplane(clf, X, y, title=titles[i])
plt.show()
运行结果:
预测乳腺癌数据集
这里在最后一部分(循环中调用plot_learning_curve())总是会有反复调用这个文件自己的奇怪现象,全部放到函数里就好了,还不清楚为什么会有这种情况。
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from sklearn.svm import SVC
from z7.titanic import plot_curve # 绘制score随参数变化的曲线
import numpy as np
from sklearn.model_selection import GridSearchCV
from matplotlib import pyplot as plt
from z3.learning_curve import plot_learning_curve # 绘制学习曲线
from sklearn.model_selection import ShuffleSplit
'''
SVM对乳腺癌数据集做分类
这里全部需要放在函数里调用,不然会来回调用这个文件自己(还不清楚为什么,这次不是文件重名的问题)
'''
def init():
global X, y, X_train, y_train, X_test, y_test
cancer = load_breast_cancer()
X = cancer.data
y = cancer.target
print("shape:{},阳性样本数:{},阴性样本数{}".format(X.shape, y[y == 1].shape[0], y[y == 0].shape[0]))
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2)
if __name__ == '__main__':
init()
# 高斯核的SVM模型很复杂,在如此小的数据集上造成了过拟合
clf = SVC(C=1.0, kernel='rbf', gamma=1.0)
clf.fit(X_train, y_train)
train_score = clf.score(X_train, y_train)
test_score = clf.score(X_test, y_test)
print("train score:{},test score:{}".format(train_score, test_score))
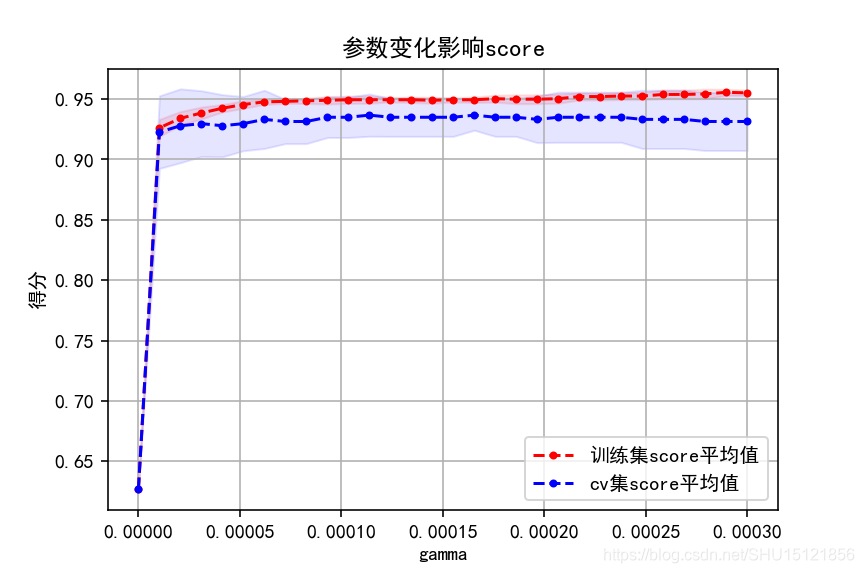
# 获取gamma参数在一个范围集合中的最优值
gammas = np.linspace(0, 0.0003, 30)
param_grid = {'gamma': gammas}
clf = GridSearchCV(SVC(), param_grid, cv=5, return_train_score=True)
clf.fit(X, y)
print("最优参数:{},对应score:{}".format(clf.best_params_, clf.best_score_))
# 绘制score随着参数变化的曲线
plot_curve(gammas, clf.cv_results_, xlabel='gamma')
plt.show()
# 绘制学习曲线以观察模型拟合情况
cv = ShuffleSplit(n_splits=10, test_size=0.2, random_state=0)
title = "高斯核SVM的学习曲线"
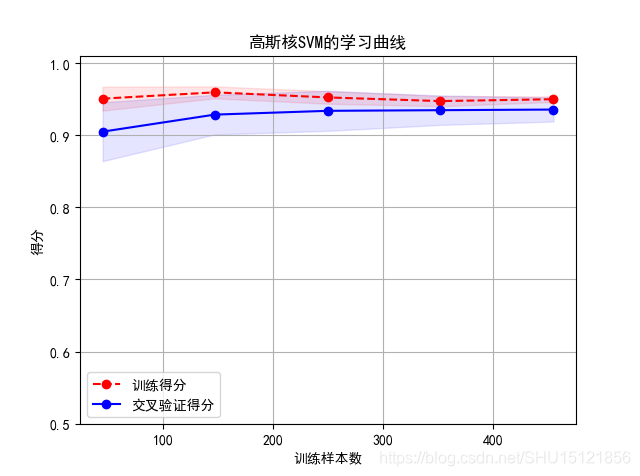
plot_learning_curve(SVC(C=1.0, kernel='rbf', gamma=0.0001), title, X, y, ylim=(0.5, 1.01), cv=cv)
plt.show()
# 使用二阶多项式核
clf = SVC(C=1.0, kernel='poly', degree=2)
clf.fit(X_train, y_train)
train_score = clf.score(X_train, y_train)
test_score = clf.score(X_test, y_test)
print("train score:{},test score:{}".format(train_score, test_score))
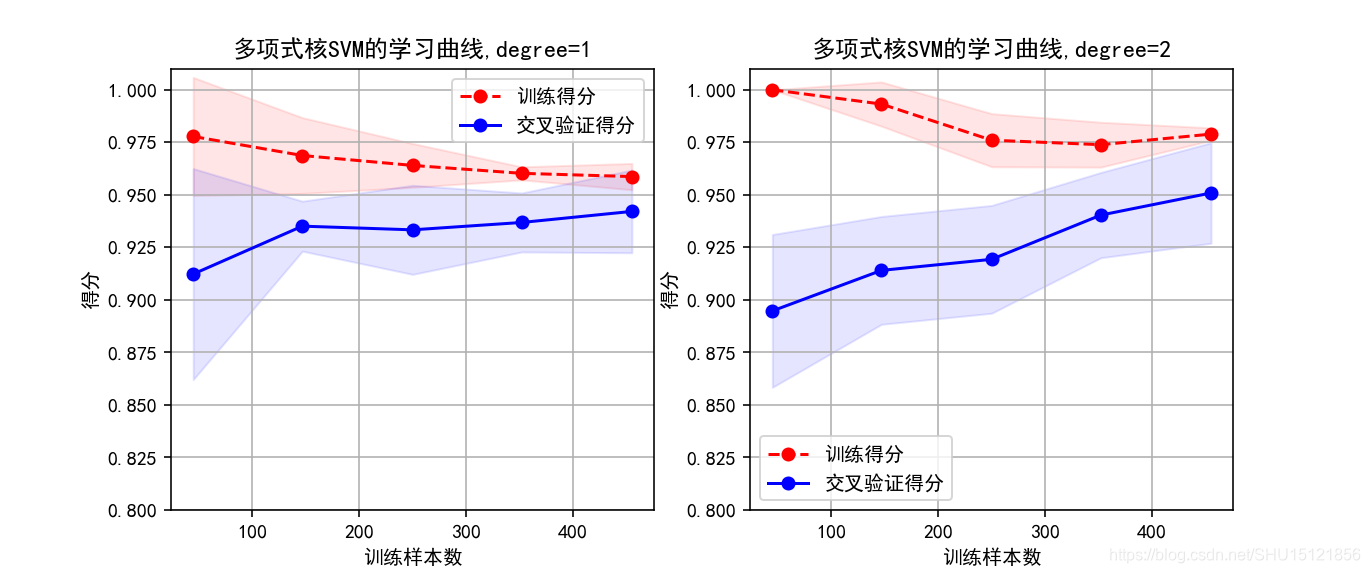
# 对比一阶多项式和二阶多项式的学习曲线
cv = ShuffleSplit(n_splits=5, test_size=0.2, random_state=0)
title = "多项式核SVM的学习曲线,degree={}"
degrees = [1, 2]
plt.figure(figsize=(12, 4), dpi=144)
for i in range(len(degrees)):
plt.subplot(1, len(degrees), i + 1)
# 这里n_jobs将传入learning_curve(),为并行运行的作业数
plt = plot_learning_curve(SVC(C=1.0, kernel='poly', degree=degrees[i]), title.format(degrees[i]), X, y,
ylim=(0.8, 1.01), cv=cv, n_jobs=4)
plt.show()
运行结果:
shape:(569, 30),阳性样本数:357,阴性样本数212
train score:1.0,test score:0.6052631578947368
最优参数:{'gamma': 0.00011379310344827585},对应score:0.9367311072056239
从下面的图中可以看到,从gamma=0.0001之后逐渐发生了过拟合,因为训练集score在提升,而cv集score在下降。
从下面这张学习曲线图上可以看到用上面找到的参数 gamma=0.0001效果就比较好,试改成gamma=0.1可以看到明显的过拟合。
train score:0.9692307692307692,test score:0.9385964912280702
二阶多项式核SVM的拟合效果更好,相比一阶多项式核,其训练集和cv集上的score都有提升,不过训练代价很高。