一、数据集简介
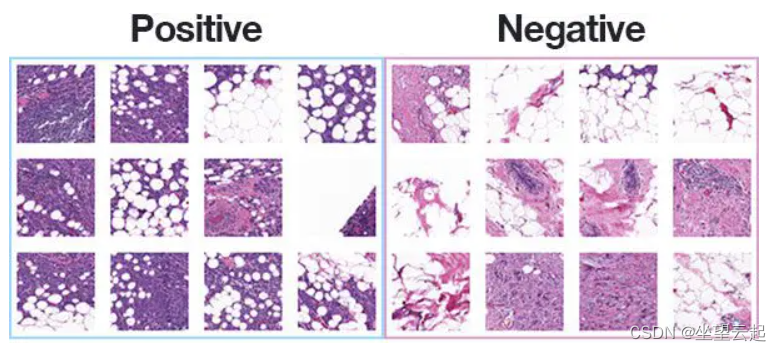
乳腺组织病理学图像
浸润性导管癌 (IDC) 是所有乳腺癌中最常见的亚型。 为了给整个样本分配侵袭性等级,病理学家通常关注包含 IDC 的区域。 因此,自动侵略性分级的常见预处理步骤之一是在整个安装载玻片内描绘 IDC 的确切区域。
乳腺癌是女性最常见的癌症形式,浸润性导管癌 (IDC) 是最常见的乳腺癌形式。 准确识别和分类乳腺癌亚型是一项重要的临床任务,可以使用自动化方法来节省时间和减少错误。
原始数据集包含 162 张以 40 倍扫描的乳腺癌 (BCa) 标本的整体载玻片图像。从中提取了 277,524 个大小为 50 x 50 的图像块(198,738 个 IDC 阴性和 78,786 个 IDC 阳性)。
数据集是kaggle提供,下面是链接地址

kaggle乳腺组织病理学图像数据集由Janowczyk、Madabhushi和Roa 等人策划。
数据集中的每个图像都有一个特定的文件名结构。数据集中的图像文件名示例如下所示:
10253_idx5_x1351_y1101_class0.png
我们可以将此文件名解释为:
患者编号: 10253_idx5
x - 坐标: 1,351
y -坐标: 1,101
类标签: 0(0表示无IDC,1表示IDC)
二、编写代码
1、配置文件
创建config.py文件
import os
# 初始化图像的原始目录的路径
ORIG_INPUT_DATASET = "datasets/orig"
# 在计算训练和测试拆分后,初始化新目录的基本路径,该目录将包含我们的图像
BASE_PATH = "datasets/idc"
# 训练、验证和测试目录
TRAIN_PATH = os.path.sep.join([BASE_PATH, "training"])
VAL_PATH = os.path.sep.join([BASE_PATH, "validation"])
TEST_PATH = os.path.sep.join([BASE_PATH, "testing"])
# 定义将用于训练的数据量
TRAIN_SPLIT = 0.8
# 验证数据量将是训练数据的百分比
VAL_SPLIT = 0.12、构建数据集
创建名为build_dataset.py的文件,执行该脚本将会划分训练、验证、测试数据集。
import config
from imutils import paths
import random
import shutil
import os
# 获取原始输入目录中所有输入图像的路径并将它们打乱
imagePaths = list(paths.list_images(config.ORIG_INPUT_DATASET))
random.seed(42)
random.shuffle(imagePaths)
# 计算训练和测试分割
i = int(len(imagePaths) * config.TRAIN_SPLIT)
trainPaths = imagePaths[:i]
testPaths = imagePaths[i:]
# 我们将使用部分训练数据进行验证
i = int(len(trainPaths) * config.VAL_SPLIT)
valPaths = trainPaths[:i]
trainPaths = trainPaths[i:]
# 定义我们将要构建的数据集
datasets = [
("training", trainPaths, config.TRAIN_PATH),
("validation", valPaths, config.VAL_PATH),
("testing", testPaths, config.TEST_PATH)
]
# 循环数据集
for (dType, imagePaths, baseOutput) in datasets:
# 打印我们正在创建的数据拆分
print("[INFO] building '{}' split".format(dType))
# 如果输出基本输出目录不存在,则创建它
if not os.path.exists(baseOutput):
print("[INFO] 'creating {}' directory".format(baseOutput))
os.makedirs(baseOutput)
# 循环输入图像路径
for inputPath in imagePaths:
# 提取输入图像的文件名并提取类标签(“0”表示“负”,“1”表示“正”)
filename = inputPath.split(os.path.sep)[-1]
label = filename[-5:-4]
# 构建标签目录的路径
labelPath = os.path.sep.join([baseOutput, label])
# 如果标签输出目录不存在,则创建它
if not os.path.exists(labelPath):
print("[INFO] 'creating {}' directory".format(labelPath))
os.makedirs(labelPath)
# 构建目标图像的路径,然后复制图像本身
p = os.path.sep.join([labelPath, filename])
shutil.copy2(inputPath, p)3、创建模型
这里使用了SeparableConv2D,看过 MobileNet 架构的人都会遇到可分离卷积的概念。可分离卷积主要有两种类型:空间可分离卷积和深度可分离卷积。可分离卷积可以减少了卷积中的参数数量。
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import BatchNormalization
from tensorflow.keras.layers import SeparableConv2D
from tensorflow.keras.layers import MaxPooling2D
from tensorflow.keras.layers import Activation
from tensorflow.keras.layers import Flatten
from tensorflow.keras.layers import Dropout
from tensorflow.keras.layers import Dense
from tensorflow.keras import backend as K
class CancerNet:
@staticmethod
def build(width, height, depth, classes):
# 将模型与输入形状一起初始化为“通道最后”和通道尺寸本身
model = Sequential()
inputShape = (height, width, depth)
chanDim = -1
# 如果我们使用“通道优先”,更新输入形状和通道维度
if K.image_data_format() == "channels_first":
inputShape = (depth, height, width)
chanDim = 1
# CONV => RELU => POOL
model.add(SeparableConv2D(32, (3, 3), padding="same",
input_shape=inputShape))
model.add(Activation("relu"))
model.add(BatchNormalization(axis=chanDim))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
# (CONV => RELU => POOL) * 2
model.add(SeparableConv2D(64, (3, 3), padding="same"))
model.add(Activation("relu"))
model.add(BatchNormalization(axis=chanDim))
model.add(SeparableConv2D(64, (3, 3), padding="same"))
model.add(Activation("relu"))
model.add(BatchNormalization(axis=chanDim))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
# (CONV => RELU => POOL) * 3
model.add(SeparableConv2D(128, (3, 3), padding="same"))
model.add(Activation("relu"))
model.add(BatchNormalization(axis=chanDim))
model.add(SeparableConv2D(128, (3, 3), padding="same"))
model.add(Activation("relu"))
model.add(BatchNormalization(axis=chanDim))
model.add(SeparableConv2D(128, (3, 3), padding="same"))
model.add(Activation("relu"))
model.add(BatchNormalization(axis=chanDim))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
# first (and only) set of FC => RELU layers
model.add(Flatten())
model.add(Dense(256))
model.add(Activation("relu"))
model.add(BatchNormalization())
model.add(Dropout(0.5))
# softmax classifier
model.add(Dense(classes))
model.add(Activation("softmax"))
# return the constructed network architecture
return model4、训练模型
创建train_model.py文件。
import matplotlib
matplotlib.use("Agg")
from tensorflow.keras.preprocessing.image import ImageDataGenerator
from tensorflow.keras.callbacks import LearningRateScheduler
from tensorflow.keras.optimizers import Adagrad
from tensorflow.keras.utils import to_categorical
from sklearn.metrics import classification_report
from sklearn.metrics import confusion_matrix
from pyimagesearch.cancernet import CancerNet
from pyimagesearch import config
from imutils import paths
import matplotlib.pyplot as plt
import numpy as np
import argparse
import os
# 构造参数解析器并解析参数
ap = argparse.ArgumentParser()
ap.add_argument("-p", "--plot", type=str, default="plot.png", help="path to output loss/accuracy plot")
args = vars(ap.parse_args())
# 初始化我们的时期数、初始学习率和批量大小
NUM_EPOCHS = 40
INIT_LR = 1e-2
BS = 32
# 确定训练、验证和测试目录中的图像路径总数
trainPaths = list(paths.list_images(config.TRAIN_PATH))
totalTrain = len(trainPaths)
totalVal = len(list(paths.list_images(config.VAL_PATH)))
totalTest = len(list(paths.list_images(config.TEST_PATH)))
# 计算每个类中训练图像的总数并初始化一个字典来存储类权重
trainLabels = [int(p.split(os.path.sep)[-2]) for p in trainPaths]
trainLabels = to_categorical(trainLabels)
classTotals = trainLabels.sum(axis=0)
classWeight = dict()
# 遍历所有类并计算类权重
for i in range(0, len(classTotals)):
classWeight[i] = classTotals.max() / classTotals[i]
# 初始化训练数据增强对象
trainAug = ImageDataGenerator(
rescale=1 / 255.0,
rotation_range=20,
zoom_range=0.05,
width_shift_range=0.1,
height_shift_range=0.1,
shear_range=0.05,
horizontal_flip=True,
vertical_flip=True,
fill_mode="nearest")
# 初始化验证(和测试)数据增强对象
valAug = ImageDataGenerator(rescale=1 / 255.0)
# initialize the training generator
trainGen = trainAug.flow_from_directory(
config.TRAIN_PATH,
class_mode="categorical",
target_size=(48, 48),
color_mode="rgb",
shuffle=True,
batch_size=BS)
# initialize the validation generator
valGen = valAug.flow_from_directory(
config.VAL_PATH,
class_mode="categorical",
target_size=(48, 48),
color_mode="rgb",
shuffle=False,
batch_size=BS)
# initialize the testing generator
testGen = valAug.flow_from_directory(
config.TEST_PATH,
class_mode="categorical",
target_size=(48, 48),
color_mode="rgb",
shuffle=False,
batch_size=BS)
# 初始化我们的模型并编译它
model = CancerNet.build(width=48, height=48, depth=3, classes=2)
opt = Adagrad(lr=INIT_LR, decay=INIT_LR / NUM_EPOCHS)
model.compile(loss="binary_crossentropy", optimizer=opt, metrics=["accuracy"])
# fit the model
H = model.fit(x=trainGen,
steps_per_epoch=totalTrain,
validation_data=valGen,
validation_steps=totalVal,
class_weight=classWeight,
epochs=NUM_EPOCHS)
# 重置测试生成器,然后使用我们经过训练的模型对数据进行预测
print("[INFO] evaluating network...")
testGen.reset()
predIdxs = model.predict(x=testGen, steps=(totalTest // BS) + 1)
# 对于测试集中的每张图像,我们需要找到具有对应最大预测概率的标签的索引
predIdxs = np.argmax(predIdxs, axis=1)
# 显示格式化的分类报告
print(classification_report(testGen.classes, predIdxs, target_names=testGen.class_indices.keys()))
# 计算混淆矩阵并使用它来推导原始准确度、灵敏度和特异性
cm = confusion_matrix(testGen.classes, predIdxs)
total = sum(sum(cm))
acc = (cm[0, 0] + cm[1, 1]) / total
sensitivity = cm[0, 0] / (cm[0, 0] + cm[0, 1])
specificity = cm[1, 1] / (cm[1, 0] + cm[1, 1])
# 显示混淆矩阵、准确性、敏感性和特异性
print(cm)
print("acc: {:.4f}".format(acc))
print("sensitivity: {:.4f}".format(sensitivity))
print("specificity: {:.4f}".format(specificity))
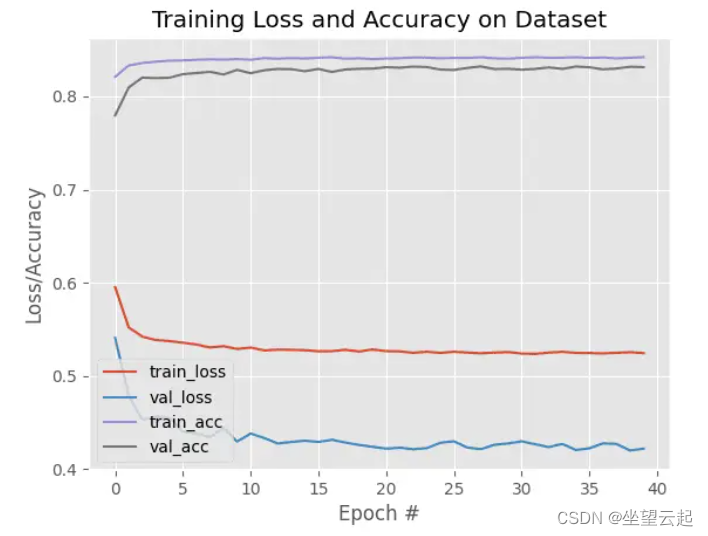
# 绘制训练损失和准确率
N = NUM_EPOCHS
plt.style.use("ggplot")
plt.figure()
plt.plot(np.arange(0, N), H.history["loss"], label="train_loss")
plt.plot(np.arange(0, N), H.history["val_loss"], label="val_loss")
plt.plot(np.arange(0, N), H.history["accuracy"], label="train_acc")
plt.plot(np.arange(0, N), H.history["val_accuracy"], label="val_acc")
plt.title("Training Loss and Accuracy on Dataset")
plt.xlabel("Epoch #")
plt.ylabel("Loss/Accuracy")
plt.legend(loc="lower left")
plt.savefig(args["plot"])