Detecting Cancer Metastases on Gigapixel Pathology Images
在高分辨率的病理图片上检测癌症转移
Liu Y, Gadepalli K, Norouzi M, et al. Detecting cancer metastases on gigapixel pathology images[J]. arXiv preprint arXiv:1703.02442, 2017.
总结
使用Camelyon16的数据集,目标是检测病理切片中的乳腺癌区域,并对该病人做出诊断。模型是Inception-v3,裁剪图片的时候裁剪299,间隔128,数据集标注是pixel-level,patch的标注根据patch中心128x128中是否有癌症来确定。用等概率选取正常和癌症的采样来解决癌症和正常patch的数据不平衡;数据扩增来解决癌症patch少的问题。预测的时候滑窗,选取有组织的部分,间隔128,裁剪大小299,只预测中间128区域。模型融合,同一张patch旋转翻转总共8张取平均,独立的训练模型,大于3个收益少。最后合成一张热力图。
ImageNet预训练模型能够加快收敛速度,但是不能提高FROC。多尺度40X,20X结合还好,多了效果没有提升。颜色标准化其他的论文都有效,但是本文没有效果,可能是因为数据扩增学到了足够的颜色信息。
non-maxima suppression方法将热力图转化为掩膜,maximum Function来预测病人是否患有乳腺癌。
摘要
美国每年有超过23万病例需要根据癌症转移程度来指定治疗方案,依据的病理切片诊断耗时而且误差大。本文提出了一种框架来自动检测和定位高像素图片中的癌症。使用CNN在Camelyon2016数据集上,检测出了92.4%的癌症区域,而人只有73.2的敏感度(recall)。我们方法在图片级别的AUC是97%,在camelyon2016测试集和另外一个110张切片的数据集上。另外还找出了2个camelyon16数据集上的标注错误。本文方法可以显著减小假阴性。
1. 引言
乳腺癌治疗方案依赖于癌症阶段,根据前哨淋巴切片需要有经验的医师和时间,并且误差大。计算机帮助诊断。
CNN在计算机视觉领域很好,同样可以用在医疗方面。
本文提出了CNN框架来辅助检测淋巴节点的乳腺癌转移。主要用最近的Inception结构,仔细的图片patch采样和数据扩增。即使使用步长为128(原来是4,预测裁剪pach的步长?),使假阳性FP减半。同时也发现了几种方法没有好处:一是模仿病理学家诊断生物组织的多尺度方法;二是ImageNet的预训练模型;三是颜色标准化。最后我们启用了随即森林和构造的特征,发现最大函数是一种高效的wsi步骤。
相关工作。Camelyon16的冠军得到了敏感度(recall)75%,wsi的AUC是92.5%。作者用pre-sample(欠采样?过采样?还是预先选好训练集?)构建数据集,训练了Inception-v1,手动构造了28个特征训练随机森林分类器来预测大图的标注。后来又训练了一个Inception模型,取了平均。这个队伍提高分数到了82.7%和99.4%使用颜色标准化,另外的数据增强,降低inference步长从64到4。Camelyon组织者也在小数据集上训练了CNN,来检测乳腺癌,也用它来做细胞核、上皮组织等的分割或者检测,F1score较高,准确率也还好。在ICPR12和AMIDA13中也用来检测细胞有丝分裂。其他也用奇迹学习来预测癌症,包括在肺部的。
2. 方法
给定一张大图,目标是识别图片中的癌症并定位。癌症的检测和定位比基于像素的分类更加重要。因为病理切片很大,而且切片很少(270),所以从切片中裁剪出小的patch。同样地在预测的时候,用滑窗把切片裁剪,然后合成一张概率热力图。对于每张切片,把热力图中最大的值作为切片的预测值。
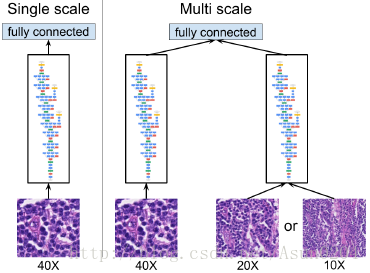
使用Inception-v3模型,输入的切片大小为299,只预测中心128x128区域的标注,只要中间128区域中出现了癌症标注,那么就认为这张切片是癌症。尝试过通过减少卷积核数量来开发参数数量的影响,也实验了多尺度方法,使用在不同倍率下的切片来裁剪patch。结果发现使用4个倍率没有好处,所以我们的结果中只有2个倍率。
癌症比正常少的数据不平衡给训练和评估带来难处,通过采样来解决。首先等概率选择正常和癌症,其次选择一张类别均匀的切片,从这张切片中采样(当作验证还是测试?)。现有的方法中都是通过pre-sample(预先选好训练集?),会限制训练的时候patch的宽度。
癌症patch少,使用数据扩增来解决。90度*4的旋转,左右翻转,再重复旋转(有必要吗?),就可以有8个方向,因为病理切片的方向没有一个权威的方向。其次使用tensorflow的图像库来实现64/255的亮度,max_delta0.25的饱和度,max_delta0.04的色度,max_delta0.75的对比度。最后在裁剪patch的时候有一个8像素的抖动。验证集中用了少量的颜色和抖动(验证集也用扩增?)。像素值限定在[0,1]之间,然后scaled到[-1,1]。
预测的时候使用滑窗,间隔128裁剪来得到中心区域。对于每个patch,通过旋转和左右翻转来获得8个预测,平均得到最后的预测结果。
实现细节。batch_size32,使用PMSProp,momentum动量因子0.9,decay衰减0.9, 初始学习率0.05,每2百万衰减0.5。预训练ImageNet的初始学习率是0.002。
要点
- 用Inception-v3模型,输入图片299,只要中间128区域有癌症,就标记为癌症,裁剪间隔128
- 癌症比正常少的数据不平衡,均匀取癌症和正常patch
- 癌症patch少,使用90度旋转、左右翻转、对比度色度亮度饱和度、裁剪时随机抖动,数据扩增来解决
- 预测的时候间隔裁剪128来覆盖中间区域,对于每个patch旋转左右翻转得到8张,分别预测取平均作为最后预测的结果
3. 评价和数据集
用了2个Camelyon16的评价指标。用AUC(ROC曲线的面积,定积分)来评价切片级别的分类。因为每张切片都有100000左右张patch,导致了潜在的假阳性FP,所以AUC提升有难度。我们用自举bootstrap(通过部分样本反映整体的方法,和假设检验类似https://en.wikipedia.org/wiki/Bootstrapping_(statistics))方法获得了95%置信区间的值。
第二个指标是FROC,用来评价癌症检测和定位。首先生成一系列的坐标,然后从各自的热力图中得到预测值。在每个带标注的肿瘤区域内的所有坐标中,保留最大的预测。在肿瘤区域外的坐标就是假阳性。用这些值去计算ROC。每个非肿瘤切片平均假阳性敏感度0.25、0.5、1、2、4、8来定义FROC。这个指标有难度,因为假阳性区域的点能够很快拉低分数。用FROC和AUC,来提高评价指标的可靠性。和AUC一样,95%的执行区间,通过2000+的bootstrap样本来计算FROC。另外用8FP的敏感度来评估假阴性(FN)率。
camelyon冠军用阈值,将概率热力图转化为二值掩膜。本文用non-maxima suppression方法,阈值t来将热力图转化为掩膜。重复1. report最大值和最大的坐标;2. 最大值为中心,r为半径中的点最大值设为0;直到热力图中没有值。其中r是128个像素,t控制report的点数量,对FROC没有影响,除非曲线的plateaus在8FP之前。为了防止降低癌症的预测,使用保守的阈值t=0.5。
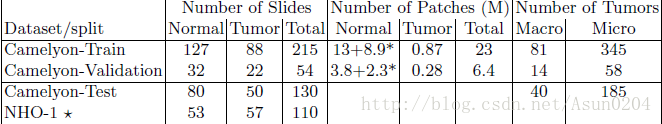
数据集。使用Camelyon16的数据集,包含400张切片,270张像素标记的切片,130张没有标注的切片作为测试集。
为了减少计算,用灰度阈值0.8排除背景patch,手动检查组织是否有遗弃。
另外的评价NHO-1。用了另外的从20个病人的110张切片,57张包含癌症,由权威的病理专家来判断。
4. 实验和结果
现有的方法从预测的热力图中提取特征,用随机森林来进行切片级的分类。不幸的是我们不能训练切片级的分类器,因为验证集的AUC是100%,没有提高的可能。除非使用每张切片热力图取得的AUC>97%的最大值,统计上来说和现在最好的结果没有区别。
为了癌症级别的分类器,发现连通分支方法connected component方法在FROC比较小的时候(<80%)能够提高1%-5%。但是这种方法对阈值很敏感,会混淆通过癌症多种提升模型的评价。相反本文的方法non-maxima suppression相对r4-6不敏感,除非验证集上准确率很低,调整r到8。最后得到100%FROC在大的癌症,表明大多数的FN都是由小的癌症组成的。
预训练模型在不同的领域能够提高性能。但是发现本文中可以提高收敛速度,但是不能提高FROC。可能是因为ImageNet的自然图像和病理图像差别太大。另外,本文的数据集大,数据扩增都能够在没有预训练的情况下提高模型训练时候的准确率。
其次检测了模型规模。当初只是为了加快试验时间,减少了3%的参数,但是和完全版本的效果一样,因此后面都是用缩小版的。
受病理学家检测的启发,使用多分辨率来检测。然而发现用40X和低分辨率性能没有提高,但是整合这些输出能够平滑热力图,可能是因为CNN的平移不变性和邻近patch的重叠。这些视觉上的提高是迷惑性的:40X的模型显示小的非癌症区域被癌症包围着。
现有的方法中显示颜色标准化可以提高性能,但是本文的实验发现没有,可能是因为额外的数据增强让模型能够学习颜色不变的特征。
最后用两种方式实现了模型融合。第一,在预测的时候平均8个方向能够有一定的提高。第二,融合独立的模型能够额外提高一点分数,但是融合3个以后提升就很少了。
另外的验证。在110张切片上测试,AUC是97.6(93.6, 100),和在Camelyon16上的分数差不多。
定性评估。086和144切片的病人没有感染,但是标注是癌症,都在训练集中,说明我们的模型抗噪能力强。另外的,发现7张标注不完全的癌症切片,5张在训练集中2张在验证集中。预测和对应的patch都在附录中。
局限性。错误发生在一些不需要关注的组织(巨噬细胞、基质)和组织准备。更好的扫描质量、组织准备和更完整的不同组织类型标注能够改善这些错误。另外,模型的超参数都是为了能够提高验证的FROC和AUC。
5. 总结
本文的方法在高分辨率的病理切片上检测小的癌症的敏感度超过的现有水平,减少了FN率。在两个独立的测试集上的AUC超过病理学家水平。本文方法能够提高准确率、乳腺癌症评估的一致性和潜在地提高病人检测。未来的工作是在大数据集上的提升。