Classification of breast cancer histology images using transfer learning

一、数据集:
BACH 2018 grand challenge
(ICIAR 2018 Grand Challenge on BreAst Cancer Histology images)
https://iciar2018-challenge.grand-challenge.org/home/
其实算得上Camelyon 16和17 的后继版本。
所不同的是,camelyon 16的数据只有WSI(whole slide images),比赛有两个任务,分别是WSI分类(是否含有肿瘤)和肿瘤位置的定位。总体来说是二类任务的分类与分割。camelyon 17 数据与16有相似之处,任务也不同,不再赘述,感兴趣可以去官网看看。
BACH也有两个任务,第一个任务是相对比较小的图片的四个阶段的分类,第二个任务是WSI中不同阶段的定位(算是四类的分割)。
BACH中第二个任务的图片分辨率极高,本文没有涉及。本文所涉及的第一个任务的图片尺寸为2048*1536 pixels(论文中写的是2040*1536,我去官网看了一下,应该是2048)。训练图片分为四类normal,benign,in situ caicinoma, invasive carcinoma。每个类别有100张图片。(这四百张其实是官方放出来的训练数据,后续还公布了测试数据,只是作者没有使用。)官网竞赛结果已经出来了,官网即可查看。本文的结果只是训练集上的结果。
作者采用train:validation:test = 60:20:20 的比例来随机划分每类图片。
二、预处理:
1.染色归一化:
训练图片均为Hematoxylin和Eosin染色再由光学显微镜呈现,所以色彩上会有一定的差异。需要归一化来校正载玻片制备过程中产生的误差。
以往的论文也会采用归一化的手段来处理色彩差异,本文采用了不一样的归一化方式,呈现出的效果的确不错,不知道最后的好结果是不是与特定的归一化手段有关。作者关于归一化方法是这么说的:
In this paper, we used the approach proposed by Reinhard et al. [11] which matches the statistics of color histograms of a source and target image, following transformation of the RGB images to the de-correlated LAB color space.
其中所涉及是这篇文章:
Reinhard, E., Adhikhmin, M., Gooch, B., Shirley, P.: Color transfer between im- ages. IEEE Computer Graphics and Applications 21(5) (Sep 2001) 34-41
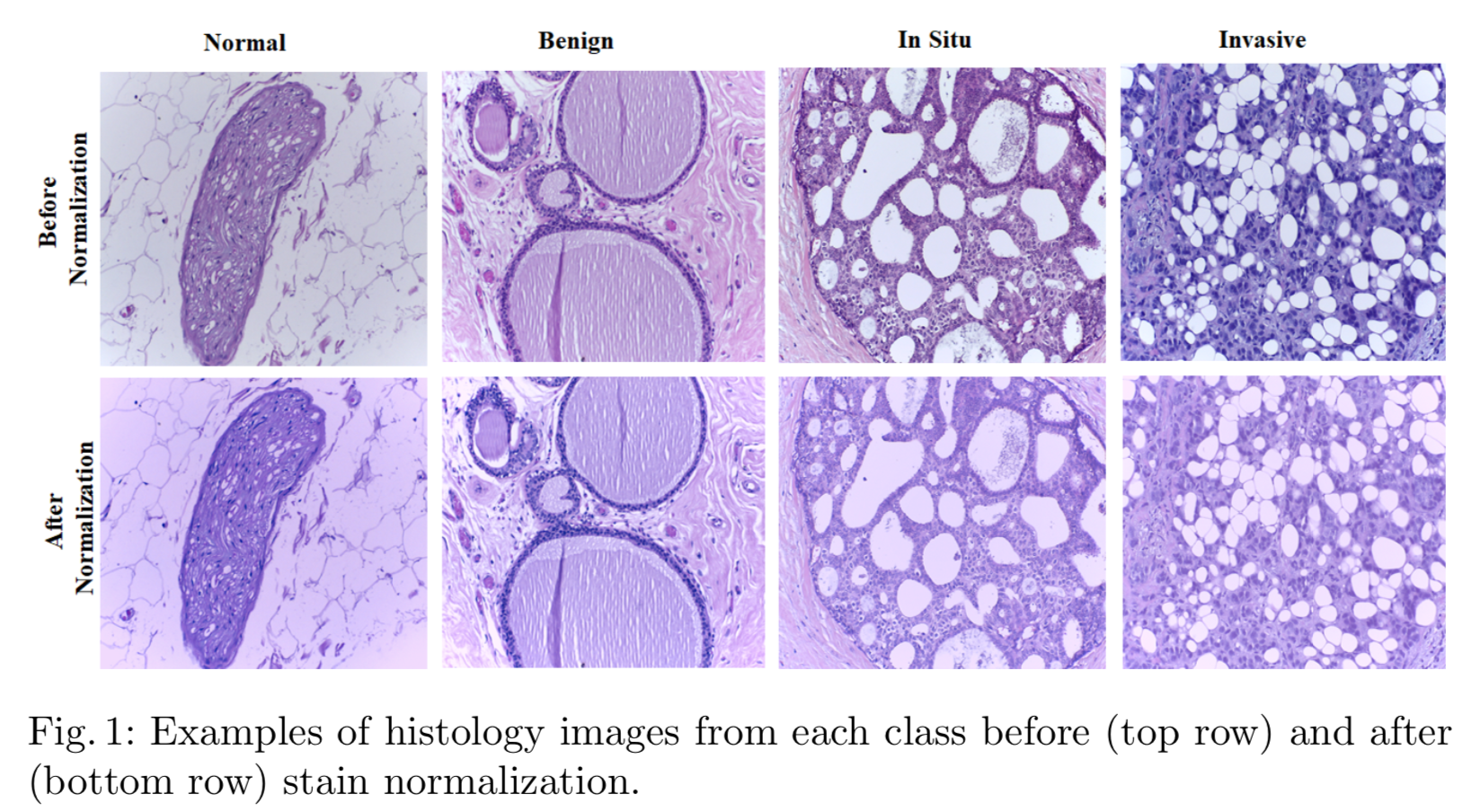
2. 其他处理:
由于2018*1536对于训练来说还是过大,而每张图片只有一个标签。裁剪过大没有减少网络的负载,而裁剪尺寸过小则有可能不包含标签所指定的这一类,通常我们只有看到较大范围的信息才能确定类别。作者选择了50%重叠(overlap)的512*512进行裁剪。也就是,每张图片35patches,共14000patches。标签(label)仍然使用原图的标签。
每个patch同时采用旋转(90、180、270)+ 镜像翻转(水平,垂直),相当于原来6倍的数据量。原来320训练数据,现在67200训练数据。测试集依然是80张(没有裁剪和变换),测试集的重叠部分采用投票机制。
三、网络结构
作者使用了Google的Inception V3 和 ResNet50,使用了他们在ImageNet预训练的参数进行fine-tune(这就是作者所说的迁移学习)。针对上述的两个网络,作者都做了些许的改动,比如V3,作者去掉了末尾五层,增加了average global pooling,fc,softmax。ResNet50也做相应的改变。据作者说,他是想获得全局的信息来判断。
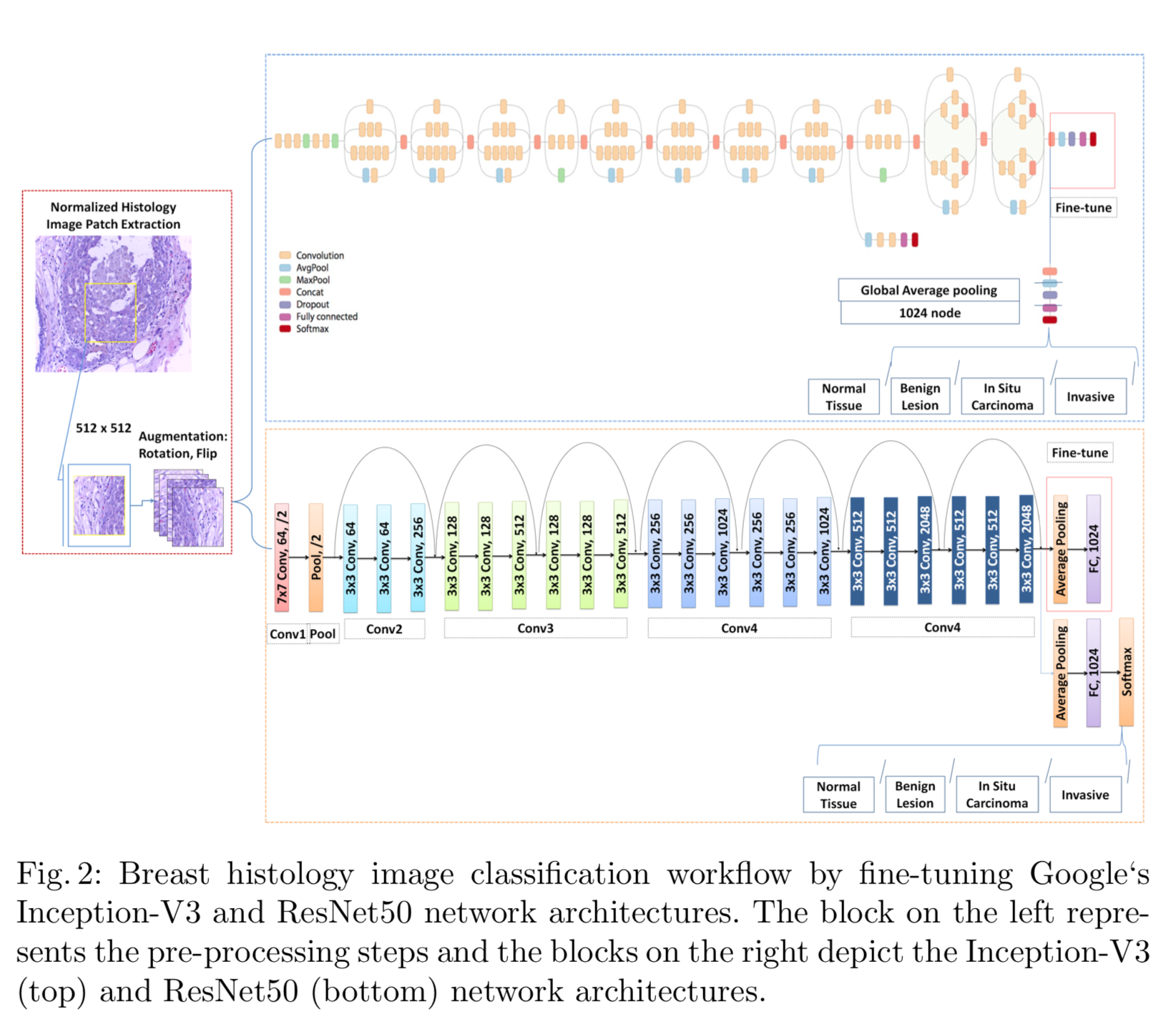
网络配置:SGD,batch_size=32, lr = 0.0001, momentum = 0.9
四、结果
评测指标:accuracy准确率=正确/总数 (作者分别计算了patch和全图的准确率)
官网对于该任务的评测指标也是accuracy

作者也给出了ROC曲线,官网并没有这个指标,但是ROC确实是医学图像分类常见的指标。
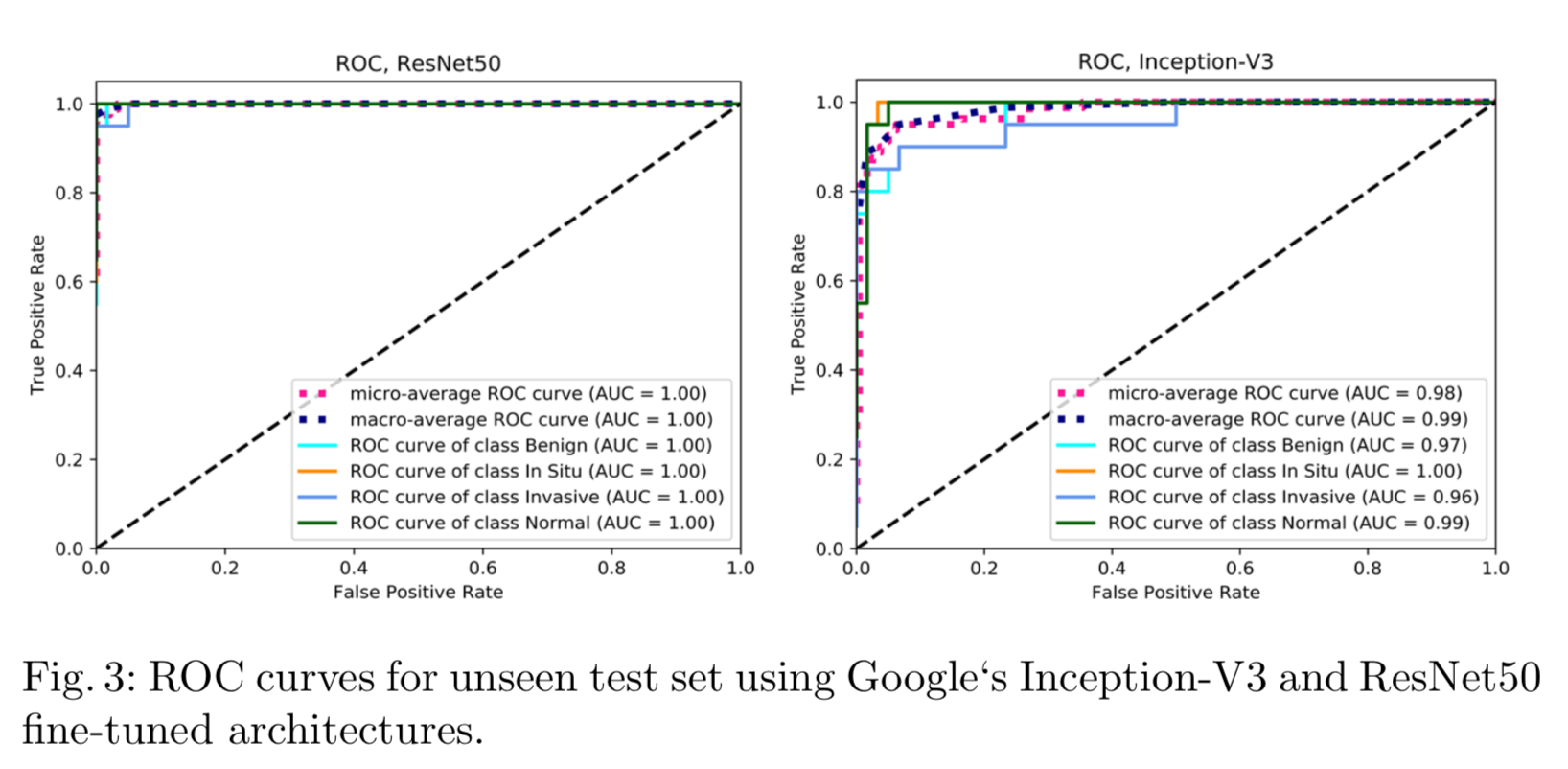
个人评价:
1、归一化的部分我还需要再仔细研究一下来验证其效果。
2、迁移学习,创新性不强,算得上baseline,同时缺乏比较。可能就是因为看起来思路过于简单了。
3、训练集结果过高,反观官网给出的测试结果最高只有0.87,作者这个是否有过拟合的嫌疑有待考证。
4、数据很新,又没有用测试集,仅仅在训练集用了一种划分产生了的结果很难有说服力。可能这种情况交叉验证会好一些。但是本文创新性不够,也没啥可比较的……如果用了交叉验证,别人也好和你比一比,现在这种随机划分一次,别人复现不出来也不好说什么……
5、改变了网络末尾的结构只给了作者自己的想法,并没有用实验证明修改的正确性(感觉是硬伤)。
6、以上纯属个人瞎唠叨,说错了别打我。还是去看看染色归一化吧(这数据看起来差异还真挺大的)