写在前面:
这篇文章的作者参加了BACH2018的竞赛,
在任务一51个队伍中取得第21名,测试集准确率0.76(冠军0.87),
在任务二13个队伍中取得第9名,测试集准确率0.46(冠军0.69)。
个人认为方法过于繁杂,一定有更简单效果更好的方法。
但也是可以借鉴的(尤其是里面有一些和常规方法背道而驰的做法,或许可以帮助我们另辟蹊径)。
阅读笔记:
Ensembling Neural Networks for Digital Pathology Images Classification and Segmentation

一、难点:
1、数字病理图片过大
2、有标签训练数据过少
二、数据集
BACH2018 这个数据集在之前的笔记中提到过。分为两个任务。
任务一:分类。400张2018*1536RGB图像,四类每类100张训练数据。
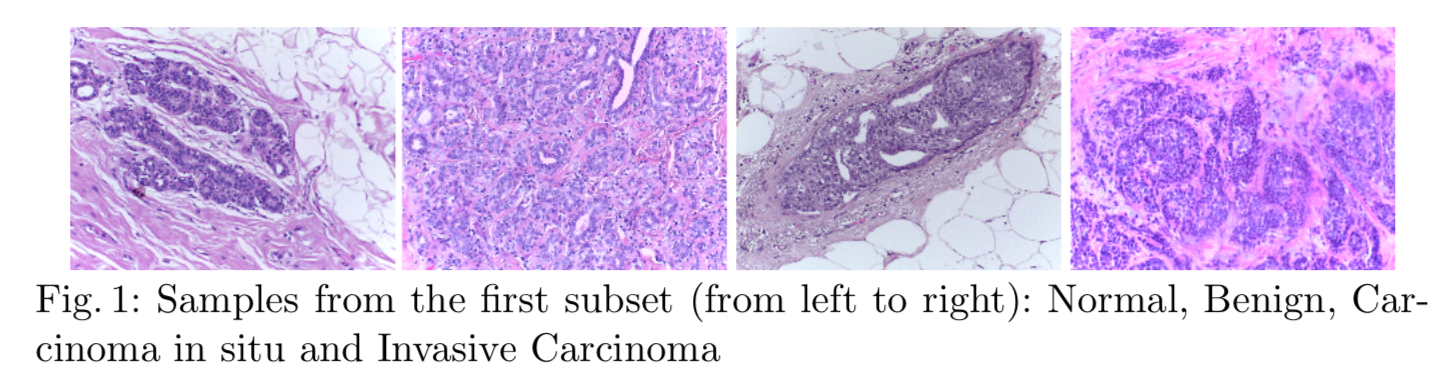
任务二:分割。WSI(20张,仅10张有标注)40000*60000RGB
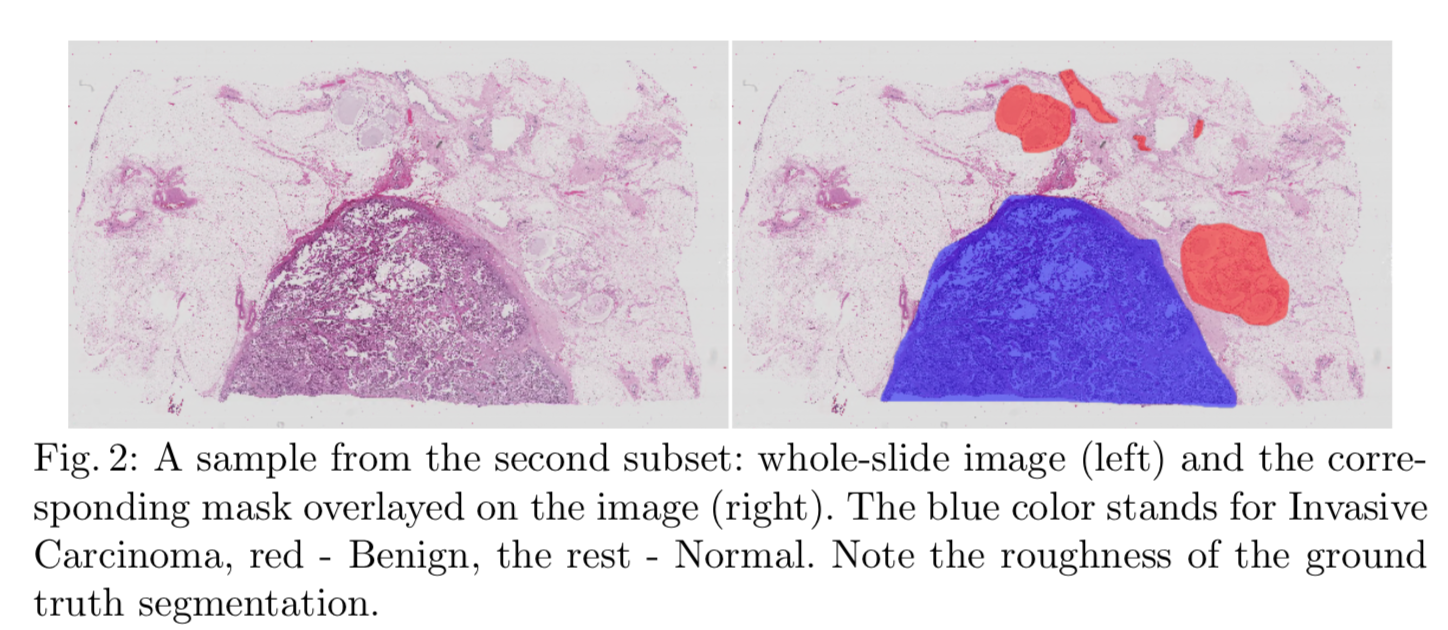
三、预处理
作者认为,病理图片的分类主要关注细胞的形态变化,因而采取了减去该图片该通道均值的方法进行归一化(作者认为针对patch归一化好于针对全图)。
四、网络结构
分类:
ResNet34:去掉了fc层前的pooling,换成了ASPP
DenseNet169和201也做类似处理
裁剪500*500(stride=100)每张图片可裁剪patch176张(每个图片会获得176*4个概率值)
Adam;lr初始0.01,每20epoch减半;batch_size=10( 模型不同而不同);有其他数据集的预训练
作者共训练了29个模型,也就意味着每类每个模型有176维的数据,根据这些数据设计特征
如 每一类的min max mean等;或有多少patch的最大概率是属于同一类的……【可以考虑采用这种特征的模式,应该会比简单的vote好一些】
作者采用XGBoost对特征分类,用贪婪算法移除不必要的模型(模型由29个缩减为12个)
分割:
同样采用减均值的归一化方法等预处理手段,网络训练策略基本与分类相同。
网络结构T-net类似于U-net,但是比U-net更浅,在跨层连接时增加了卷积层
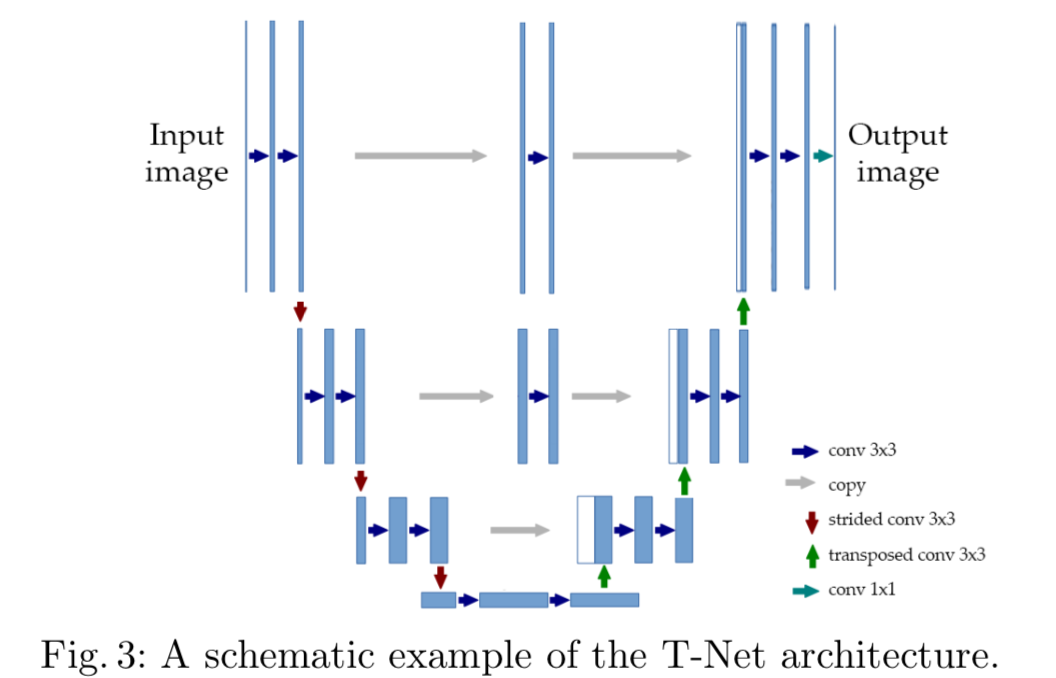
作者采用了三种方法训练网络。
1、二分类的分割。分为正常与不正常(实际上不正常有三类)
2、边缘加权
3、直接多分类分割
作者采用原图的40倍下采样进行分割
(真果敢啊,这样做大区域是不会错,但是肯定会漏检,如果精度很低是一种提升办法,如果对精度要求越来越高这肯定不太行)
作者发现生成的预测结果过了阈值之后有很多空洞,于是将原图进行了高斯模糊来减少空洞
(本来就下采样了,还要模糊?!不过作者说空洞少了,那就是少了吧……总觉得怪怪的)
作者对最后的结果进行了形态学闭运算
(这样做确实能填补空洞,但是噪声呢。。。噪点放大了真的好么,难道通常不是都恨不得开运算么……)
作者为了提升精度,采用了Normal vs All 的二分类分割模式,然后再对非正常区域二分类分成iv和b
(直接舍弃了一类,作者说这样虽然简单粗暴,但是能够显著提升精度。。。)
细节不赘述了,这篇文章真的是让人印象深刻,果敢……没准胆大有奇迹呢,说不定常规思路没人家做的好呢……反正任务二参赛的队伍做的都不算太好。
不过说实话名次不算高,尤其第一个任务,过程繁琐(主要是指模型训练了一大堆)但收效不佳,坐等更好的方法~