sklearn是Python重要的机器学习库,是scikit-learn的简称,支持包括分类、回归、降维和聚类四大机器学习算法。本文以Kmeans与DBSCAN为例,介绍其聚类函数的用法。
sklearn中聚类的具体用法,可在sklearn之cluster 中查看,实现了包括KMeans、DBSCAN在内的如下九种聚类:
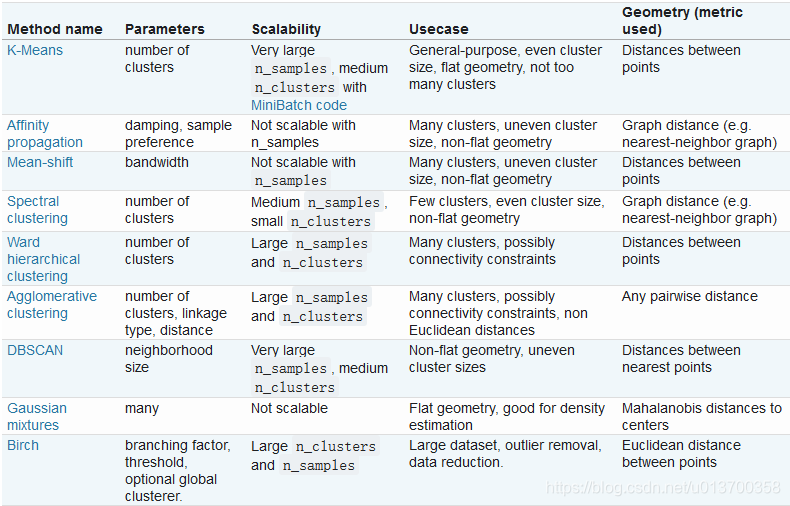
KMeans函数构造如下:
class sklearn.cluster.KMeans(n_clusters=8, init=’k-means++’, n_init=10, max_iter=300, tol=0.0001, precompute_distances=’auto’, verbose=0, random_state=None, copy_x=True, n_jobs=None, algorithm=’auto’)其中,我们平时主要会涉及的参数有如下几个:
1) n_clusters: 即k值,一般需要多试一些值以获得较好的聚类效果。默认为8个聚类簇
2)max_iter: 最大的迭代次数,一般如果是凸数据集的话可以不管这个值,如果数据集不是凸的,可能很难收敛,此时可以指定最大的迭代次数让算法可以及时退出循环。
3)n_init:用不同的初始化质心运行算法的次数。由于K-Means是结果受初始值影响的局部最优的迭代算法,因此需要多跑几次以选择一个较好的聚类效果,默认是10,一般不需要改。如果你的k值较大,则可以适当增大这个值。
4)init: 即初始值选择的方式,可以为完全随机选择'random',优化过的'k-means++'或者自己指定初始化的k个质心。一般建议使用默认的'k-means++'。
5)algorithm:有“auto”, “full” or “elkan”三种选择。"full"就是我们传统的K-Means算法, “elkan”是我们原理篇讲的elkan K-Means算法。默认的"auto"则会根据数据值是否是稀疏的,来决定如何选择"full"和“elkan”。一般数据是稠密的,那么就是 “elkan”,否则就是"full"。一般来说建议直接用默认的"auto"
DBSCAN的函数构造如下:
class sklearn.cluster.DBSCAN(eps=0.5, min_samples=5, metric=’euclidean’, metric_params=None, algorithm=’auto’, leaf_size=30, p=None, n_jobs=None)平时可能用到的参数解释如下:
1)eps: DBSCAN算法参数,即ϵ-邻域的距离阈值,和样本距离超过ϵ的样本点不在ϵ-邻域内。一般需要通过在多组值里面选择一个合适的阈值。eps过大,则更多的点会落在核心对象的ϵ-邻域,此时类别数可能会减少, 本来不应该是一类的样本也会被划为一类。反之则类别数可能会增大,本来是一类的样本却被划分开。默认为0.5
2)min_samples: DBSCAN算法参数,即样本点要成为核心对象所需要的ϵ-邻域的样本数阈值。 一般需要通过在多组值里面选择一个合适的阈值。通常和eps一起调参。在eps一定的情况下,min_samples过大,则核心对象会过少,此时簇内部分本来是一类的样本可能会被标为噪音点,类别数也会变多。反之min_samples过小的话,则会产生大量的核心对象,可能会导致类别数过少。默认值是5.
3)metric:最近邻距离度量参数。可以使用的距离度量较多,一般来说DBSCAN使用默认的欧式距离(即p=2的闵可夫斯基距离)就可以满足我们的需求。可以使用的距离度量参数有:
a) 欧式距离 “euclidean”:
b) 曼哈顿距离 “manhattan”:
c) 切比雪夫距离“chebyshev”:
d) 闵可夫斯基距离 “minkowski”: p=1为曼哈顿距离, p=2为欧式距离。
e) 带权重闵可夫斯基距离 “wminkowski”: 其中w为特征权重
f) 标准化欧式距离 “seuclidean”: 即对于各特征维度做了归一化以后的欧式距离。此时各样本特征维度的均值为0,方差为1.
g) 马氏距离“mahalanobis”:其中,
为样本协方差矩阵的逆矩阵。当样本分布独立时, S为单位矩阵,此时马氏距离等同于欧式距离。
还有一些其他不是实数的距离度量,一般在DBSCAN算法用不上,这里也就不列了。
4)algorithm:最近邻搜索算法参数,一共有4种可选输入,‘brute’对应第一种蛮力实现,‘kd_tree’对应第二种KD树实现,‘ball_tree’对应第三种的球树实现, ‘auto’则会在上面三种算法中做权衡,选择一个拟合最好的最优算法。需要注意的是,如果输入样本特征是稀疏的时候,无论我们选择哪种算法,最后scikit-learn都会去用蛮力实现‘brute’。个人的经验,一般情况使用默认的 ‘auto’就够了。 如果数据量很大或者特征也很多,用"auto"建树时间可能会很长,效率不高,建议选择KD树实现‘kd_tree’,此时如果发现‘kd_tree’速度比较慢或者已经知道样本分布不是很均匀时,可以尝试用‘ball_tree’。而如果输入样本是稀疏的,无论你选择哪个算法最后实际运行的都是‘brute’。
5)leaf_size:最近邻搜索算法参数,为使用KD树或者球树时, 停止建子树的叶子节点数量的阈值。这个值越小,则生成的KD树或者球树就越大,层数越深,建树时间越长,反之,则生成的KD树或者球树会小,层数较浅,建树时间较短。默认是30. 因为这个值一般只影响算法的运行速度和使用内存大小,因此一般情况下可以不管它。
6) p: 最近邻距离度量参数。只用于闵可夫斯基距离和带权重闵可夫斯基距离中p值的选择,p=1为曼哈顿距离, p=2为欧式距离。如果使用默认的欧式距离不需要管这个参数。
设定了参数后,即可调用进行聚类,具体用法如下:
import random
from sklearn.cluster import KMeans
from sklearn.cluster import DBSCAN
import matplotlib.pyplot as plt
all_points = []
colors = ['b', 'g', 'r', 'c', 'm', 'y', 'k', 'b']
markers = ['o', 's', 'D', 'v', '^', 'p', '*', '+']
# 随机生成100个点
for i in range(100):
generateddata=[random.randint(1,100)/100,random.randint(1,100)/100]
if not generateddata in all_points: # 去掉重复数据
all_points.append(generateddata)
# 调用KMeans方法, 聚类数为4个,fit()之后开始聚类
kmeans = KMeans(n_clusters=4).fit(all_points)
# 调用DBSCAN方法, eps为最小距离,min_samples 为一个簇中最少的个数,fit()之后开始聚类
dbscan = DBSCAN(eps = 0.132, min_samples = 2).fit(all_points)
# 开始画图
plt.subplot(1, 2, 1)
plt.title('kmeans')
for i, l in enumerate(kmeans.labels_):
plt.plot(all_points[i][0], all_points[i][1], color = colors[l], marker = markers[l])
plt.subplot(1, 2, 2)
plt.title('dbscan')
for i, l in enumerate(dbscan.labels_):
# if l == -1
plt.plot(all_points[i][0], all_points[i][1], color = colors[l], marker = markers[l])
plt.show()
要注意dbscan方法,会自动识别离群点,并将其标记为-1,结果如下:
