提示:文章写完后,目录可以自动生成,如何生成可参考右边的帮助文档
文章目录
一、聚类算法
聚类算法又叫做“无监督分类”,其目的是将数据划分成有意义或有用的组(或簇)。这种划分可以基于我们的业务需求或建模需求来完成,也可以单纯地帮助我们探索数据的自然结构和分布。比如在商业中,如果我们手头有大量的当前和潜在客户的信息,我们可以使用聚类将客户划分为若干组,以便进一步分析和开展营销活动,最有名的客户价值判断模型RFM,就常常和聚类分析共同使用。再比如,聚类可以用于降维和矢量量化(vectorquantization),可以将高维特征压缩到一列当中,常常用于图像,声音,视频等非结构化数据,可以大幅度压缩数据量。典型的聚类算法有DBSCAN、KMeans、EM聚类等。
二、KMeans
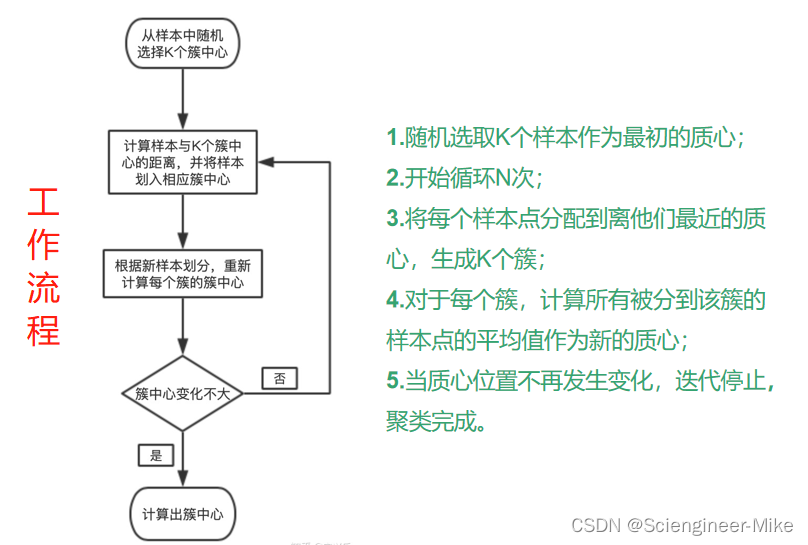
2.1 算法原理介绍
作为聚类算法的典型代表,KMeans是聚类算法中最简单的算法之一,那它是怎么完成聚类的呢?KMeans算法将一组N个样本的特征矩阵X划分为K个无交集的簇,直观上来看是簇是一组一组聚集在一起的数据,在一个簇中的数据就认为是同一类。簇就是聚类的结果表现。簇中所有数据的均值 通常被称为这个簇的“质心”(centroids)。在一个二维平面中,一簇数据点的质心的横坐标就是这一簇数据点的横坐标的均值,质心的纵坐标就是这一簇数据点的纵坐标的均值。同理可推广至高维空间。
在KMeans算法中,簇的个数k是一个超参数,需要我们根据业务需求进行确定。找出K个最优的质心,并将离这些质心最近的数据分别分配到这些质心代表的簇中去。具体过程可以总结如下:
分类的目标是为了达到"簇内差异小,簇外差异大",而这个差异是样本点到其所在簇的质心的距离来衡量。对于一个簇来说,所有样本点到质心的距离之和越小,我们就认为这个簇中的样本越相似,簇内差异就越小。而距离的衡量方法有多种,令 表示簇中的一个样本点,表示该簇中的质心,n表示每个样本点中的特征数目,i表示组成点的每个特征,则该样本点到质心的距离可以由以下距离来度量:

如我们采用欧几里得距离,则一个簇中所有样本点到质心的距离的平方和为:

其中,m为一个簇中样本的个数,j是每个样本的编号。这个公式被称为簇内平方和cluster Sum of Square,又叫做Inertia。而将一个数据集中的所有簇的簇内平方和相加,就得到了整体平方和Total Cluster Sum of Square,又叫做total inertia。Total Inertia越小,代表着每个簇内样本越相似,聚类的效果就越好。因此KMeans追求的是,求解能够让Inertia最小化的质心。
2.2 算法性能评估指标
轮廓系数是最常用的聚类算法的评价指标。我们假定:
(1)样本与其所在的簇中的其他样本的相似度a,等于样本与同一簇中所有其他点之间的平均距离。
(2)样本与其他簇中的样本的相似度b,等于样本与下一个最近的簇中的所有点之间的平均距离。
单个样本的轮廓系数计算为:

很容易理解轮廓系数范围是(-1,1),其中值越接近1表示样本与自己所在的簇中的样本很相似,并且与其他簇中的样本不相似,当样本点与簇外的样本更相似的时候,轮廓系数就为负。当轮廓系数为0时,则代表两个簇中的样本相似度一致,两个簇本应该是一个簇。
在sklearn中,我们使用模块metrics中的类silhouette_score来计算轮廓系数,它返回的是一个数据集中,所有样本的轮廓系数的均值。但我们还有同在metrics模块中的silhouette_sample,它的参数与轮廓系数一致,但返回的是数据集中每个样本自己的轮廓系数。除了轮廓系数是最常用的,我们还有卡林斯基-哈拉巴斯指数(Calinski-Harabaz Index,简称CHI,也被称为方差比标准)对应的API为:sklearn.metrics.calinski_harabaz_score (X, y_pred),戴维斯-布尔丁指数(Davies-Bouldin)对应的API为sklearn.metrics.davies_bouldin_score (X, y_pred),以及权变矩阵(Contingency Matrix)对应的API为sklearn.metrics.cluster.contingency_matrix (X, y_pred)可以使用。
三、代码实现
3.1 sklearn_api的介绍
class sklearn.cluster.KMeans (n_clusters=8, init=’k-means++’, n_init=10, max_iter=300, tol=0.0001,
precompute_distances=’auto’, verbose=0, random_state=None, copy_x=True, n_jobs=None, algorithm=’auto’)
重要参数:
n_clusters:分类簇数。
init:初始化质心的方法,”K-means++“,"random"或者一个n维数组。
max_iter:最大迭代次数。
tol:连续两次迭代inertia下降的量,小于该值迭代停止。
3.2 sklearn代码实现
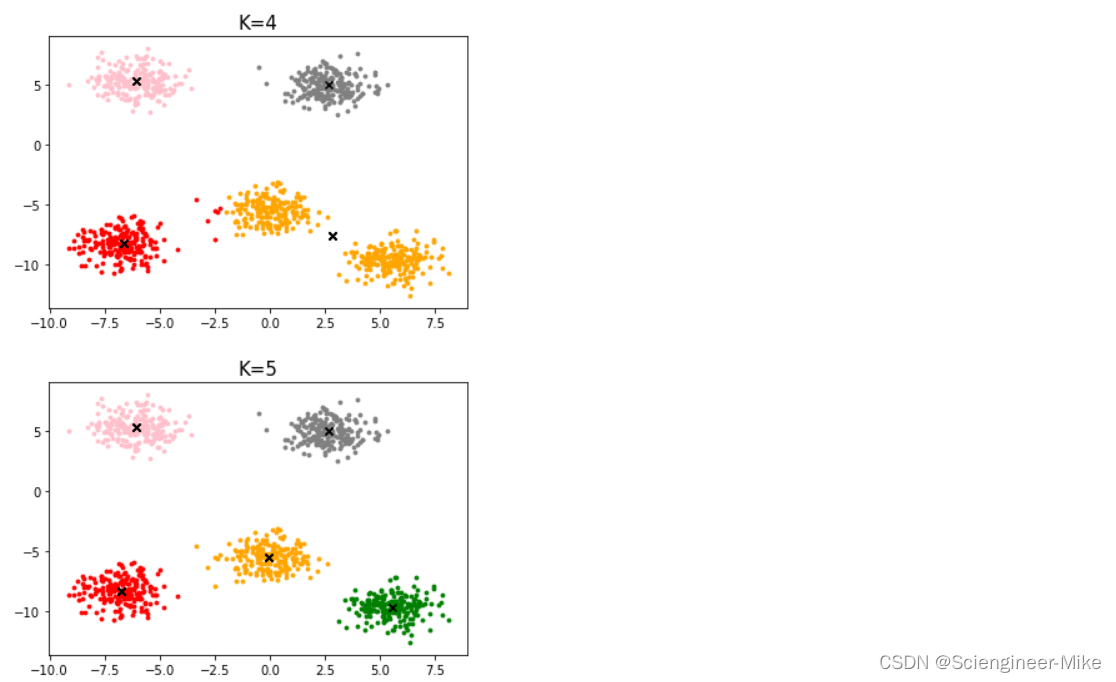
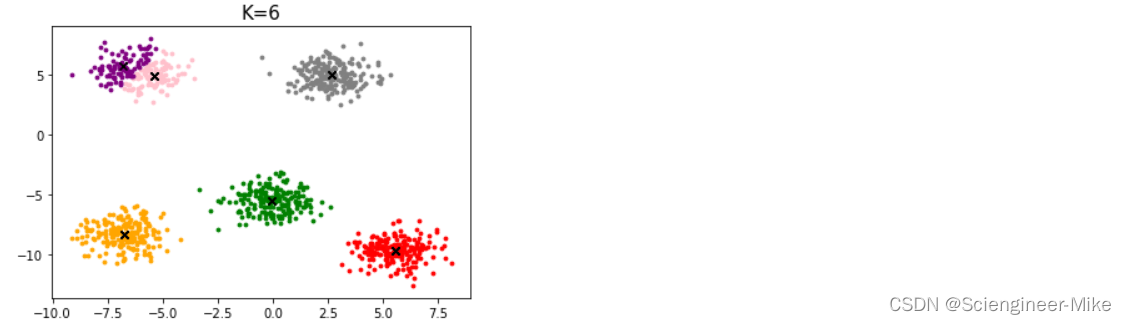
使用make_blobs随机生成1000个样本,五个blob,对其进行聚类,代码如下:
# 导入包
from sklearn.datasets import make_blobs
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_score
#自己创建数据集
X, y = make_blobs(n_samples=1000,n_features=2,centers=5,random_state=10)
# 聚类数据集并展示结果
for i in range(4,7):
n_clusters=i
cluster = KMeans(n_clusters=n_clusters, random_state=0).fit(X)
y_pred = cluster.predict(X)
centroid = cluster.cluster_centers_# 质心
# 输出整体平方和和轮廓系数
# print(centroid)
inertia = cluster.inertia_
print("K = %s 整体平方和:"%i,inertia)
s_score = silhouette_score(X,y_pred)
print("K = %s轮廓系数:"%i,s_score)
# 可视化结果
color = ["red","pink","orange","gray","green","purple"]
fig, ax1 = plt.subplots(1,sharey=True)
for i in range(n_clusters):
ax1.scatter(X[y_pred==i, 0], X[y_pred==i, 1]
,marker='o'
,s=8
,c=color[i]
)
ax1.set_title("K=%s"%i,fontsize=15)
ax1.scatter(centroid[:,0],centroid[:,1]
,marker="x"
,s=35
,c="black")
plt.show()
K值为4,5,6的结果,如下,

3.3 Python代码
代码如下:
import numpy as np
import matplotlib.pyplot as plt
# 欧氏距离计算
def distEclud(x,y):
return np.sqrt(np.sum((x-y)**2)) # 计算欧氏距离
# 为给定数据集构建一个包含K个随机质心的集合
def randCent(X,k):
m,n = X.shape
centroids = np.zeros((k,n))
for i in range(k):
index = int(np.random.uniform(0,m)) #
centroids[i,:] = X[index,:]
return centroids
# k均值聚类
def KMeans(X,k):
m = np.shape(X)[0] #行的数目
# 第一列存样本属于哪一簇
# 第二列存样本的到簇的中心点的误差
clusterAssment = np.mat(np.zeros((m,2)))
clusterChange = True
# 第1步 初始化centroids
centroids = randCent(X,k)
while clusterChange:
clusterChange = False
# 遍历所有的样本(行数)
for i in range(m):
minDist = 1000000.0
minIndex = -1
# 遍历所有的质心
#第2步 找出最近的质心
for j in range(k):
# 计算该样本到质心的欧式距离
distance = distEclud(centroids[j,:],X[i,:])
if distance < minDist:
minDist = distance
minIndex = j
# 第 3 步:更新每一行样本所属的簇
if clusterAssment[i,0] != minIndex:
clusterChange = True
clusterAssment[i,:] = minIndex,minDist**2
#第 4 步:更新质心
for j in range(k):
pointsInCluster = X[np.nonzero(clusterAssment[:,0].A == j)[0]] # 获取簇类所有的点
centroids[j,:] = np.mean(pointsInCluster,axis=0) # 对矩阵的行求均值
print("Congratulations,cluster complete!")
return centroids,clusterAssment
def showCluster(X,k,centroids,clusterAssment):
m,n = X.shape
if n != 2:
print("数据不是二维的")
return 1
mark = ['or', 'ob', 'og', 'ok', '^r', '+r', 'sr', 'dr', '<r', 'pr']
if k > len(mark):
print("k值太大了")
return 1
# 绘制质心
plt.scatter(centroids[:,0],centroids[:,1],c="black")
# 绘制所有的样本
for i in range(m):
markIndex = int(clusterAssment[i,0])
plt.plot(X[i,0],X[i,1],mark[markIndex])
plt.show()
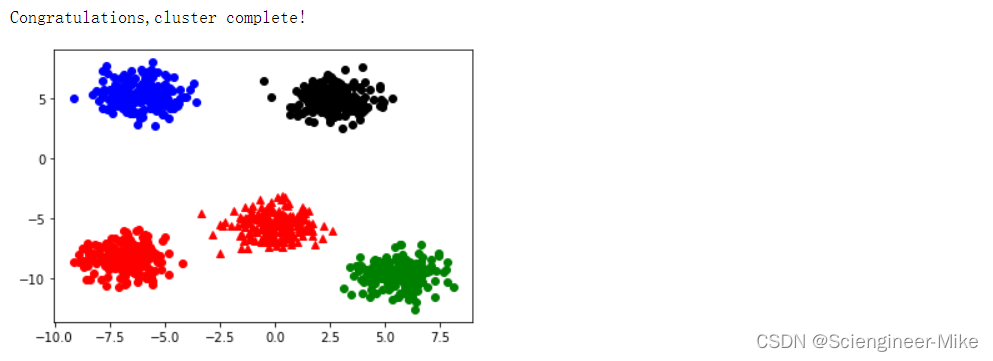
X, y = make_blobs(n_samples=1000,n_features=2,centers=5,random_state=10)
k = 5
centroids,clusterAssment = KMeans(X,k)
showCluster(X,k,centroids,clusterAssment)
聚类结果展示:
四、总结
通过以上的学习,发现KMeans是一个比较简单的算法,原理简单,收敛速度快,这个是业界用它最多的重要原因之一。调参的时候只需要改变k一个参数,可解释性好。但对于离群点和噪音点敏感。例如在距离中心很远的地方手动加一个噪音点,那么中心的位置就会被拉跑偏很远,并且,k值的选择很难确定。
链接: 【机器学习之集成算法】RandomForest和XGboost原理介绍与代码实现
链接: 【机器学习之特征工程】数据预处理、特征选择、降维及不平衡处理
链接: 【机器学习之逻辑回归】sklearn+python逻辑回归详解
链接: 【机器学习之线性回归】多元线性回归模型的搭建+Lasso回归的特征提取
链接: 【机器学习之决策树】决策树原理介绍及代码实现sklearn