小麦叶际细菌中存在大量活性、多样性和功能均未知的原噬菌体
Widespread and largely unknown prophage activity, diversity, and function in two genera of wheat phyllosphere bacteria

Article,2023-11-2,The ISME Journal, [IF 11]
DOI:https://doi.org/10.1038/s41396-023-01547-1
原文链接:https://doi.org/10.1038/s41396-023-01547-1
第一作者:Peter Erdmann Dougherty
通讯作者:Lars Hestbjerg Hansen
主要单位:
1.哥本哈根大学植物与环境科学系(Department of Plant and Environmental Sciences, University of Copenhagen)
2.荷兰生态研究所微生物生态学系(Department of Microbial Ecology, Netherlands Institute of Ecology)
- 摘要 -
环境细菌中寄生着大量原噬菌体,但它们的多样性和天然功能在很大程度上仍然难以捉摸。本文研究了从小麦旗叶中分离出的63株欧文氏菌和假单胞菌中的原噬菌体活性和多样性。通过引入和验证病毒诱导测序(VIP-Seq),科研人员识别并量化了12个自发诱导的原噬菌体的活性,发现一些叶际细菌在过夜培养中产生的病毒超过108 个/毫升,在植物体内也观察到显著的诱导作用。测序结果和噬菌斑测定表明,大部分种内遗传多样性由蚜虫欧文氏菌原噬菌体造成,并通过将细菌宿主分裂为不同的派系而广泛参与到微生物战争中揭示了原噬菌体介导的微生物多样性的重要性。当将自发活性的原噬菌体与预测的原噬菌体比较时,我们还发现插入序列与非活性原噬菌体密切相关。总之,研究发现了植物叶际细菌中存在广泛且很大程度上功能和多样性均未知的原噬菌体。
- 引言 -
尽管噬菌体是地球上最丰富的实体已成为一种经常被引用的说法,但许多噬菌体并不以我们通常想象的自由漂浮的病毒颗粒的形式存在,而是作为原噬菌体整合在大多数细菌的基因组中。从这个角度来看,“大多数”微生物学家都在研究噬菌体,尽管是间接的并且常常是在不知不觉中进行的。因此,更好地了解原噬菌体的活性和功能可能会深入了解其细菌宿主的行为。
原噬菌体代表温和噬菌体感染的休眠阶段,是与其宿主细菌垂直复制的噬菌体基因组。原噬菌体可以无限期地保持在这种状态,直到被诱导产生噬菌体病毒颗粒并从细胞中爆发出来,或者被突变驯化,使其无法诱导。尽管噬菌体在细菌类群中几乎普遍存在,但其分布并不均匀。最近对10,370个细菌和古细菌基因组的研究发现,75%的基因组中存在预测的原噬菌体,平均每个基因组有3.24个原噬菌体。与烈性(非整合)噬菌体的严格捕食性质相反,这些温和的噬菌体对其细菌宿主而言是一把双刃剑。绝大多数温和噬菌体感染会导致病毒立即复制和细菌死亡,但当整合时,原噬菌体可能通过毒力因子有条件地提高宿主适应性。有益的是,原噬菌体还可以为其宿主提供对相关噬菌体感染的抵抗力,尽管这种抵抗力的广度各不相同。两种微生物系统的建模还表明,原噬菌体诱导可以作为宿主的有效的自我复制武器。然而,与烈性噬菌体相反,由于毒力因子的潜在传播,温和噬菌体通常被禁止作为生物防治剂。
存在许多可能的原噬菌体诱导触发因素,包括DNA损伤、细菌毒素和噬菌体编码的通讯系统。许多原噬菌体还表现出所谓的自发诱导,即溶原菌在常规细菌培养物中产生可检测水平的游离噬菌体,尽管有证据表明“自发”诱导是对某些触发因素的反应,例如在细菌群落的子集中观察到的DNA损伤。更复杂的是,原噬菌体诱导率受到培养基选择的影响,而群体感应等间接效应也可能以不可预测的方式影响诱导率。尽管存在这些有趣的动态,但关于原噬菌体在微生物生态学中的作用,尤其是在叶际中的作用,人们仍知之甚少。原噬菌体诱导可在肠道细菌中被广泛观察到,有证据表明它们可能在调节微生物群落中发挥重要作用。尽管我们越来越了解它们的重要性,但在对植物有益的合成菌群(SynComs) 领域中,噬菌体并未得到普遍研究。然而,有迹象表明原噬菌体是叶际中的重要参与者。尽管未对原噬菌体进行研究,但噬菌体耗竭已被证明会改变细菌组成。最近一项针对小麦叶际的宏基因组学研究还发现,预计24%的噬菌体是温和噬菌体,而数量最多的噬菌体是温和噬菌体哈米顿氏菌病毒APSE,它可以保护蚜虫免受寄生黄蜂的侵害。
识别原噬菌体可能很困难。尽管PHASTER和VIBRANT等生物信息学工具可以通过细菌基因组组装进行计算机原噬菌体预测,但基于序列的原噬菌体鉴定具有局限性,例如确定原噬菌体是否可行,并且无法预测原噬菌体的相对诱导率或它们是在什么条件下被诱导的。为了量化诱导的原噬菌体,广泛使用传统的噬菌斑测定。然而,由于这需要易感宿主,许多研究使用了免培养技术(TEM、落射荧光显微镜、qPCR)。最近,一些工具通过将全基因组鸟枪法(WGS)读序序列比对到细菌组装体来识别原噬菌体活性。PropagAtE和hafeZ都直接搜索具有高读序覆盖率的区域(计算原噬菌体/宿主读序覆盖率),而Prophage Tracer搜索不一致的读序以估计原噬菌体切除率(尽管它不能在具有多个活性原噬菌体的宿主中准确量化切除)。然而,高水平的背景染色体覆盖限制了WGS数据的检测限。为了解决这个问题,Tranductomics流程仅对诱导噬菌体的封装 DNA 进行测序,从而提高了检测限并能够研究转导模式(无需噬菌体定量)。基于这些工具的优点,我们引入并验证了Virion诱导分析测序(VIP-Seq)。通过量化封装DNA中的DNA浓度,结合读序比对和不一致读序检查,VIP-Seq能够以高灵敏度对所有诱导的原噬菌体滴度进行鉴定和绝对定量。
我们将VIP-Seq和其他技术应用于从单一环境中分离出来的菌株集合,首次对叶际中的原噬菌体进行了研究。从单田小麦旗叶中分离出的63株新测序的欧文氏菌和假单胞菌中,我们发现了来自23个新属的120个自发诱导的原噬菌体,量化了它们的滴度,发现许多在过夜培养物中高度诱导。我们还利用小麦幼苗进一步证明,广泛的噬菌体诱导也可以在植物体内发生。通过调查传统宏基因组研究未发现微多样性的重要性,我们发现原噬菌体是种内多样性和战争的主要促进者,将其宿主分裂成不相容的“吞噬型”。最后,与生物信息学原噬菌体预测的比较也揭示了IS介导的原噬菌体失活的主要差异和建议。我们的研究结果首次发现,原噬菌体在小麦叶际细菌中普遍存在、活跃、有遗传多样性且具有影响力。
- 方法 -
用病毒粒子诱导分析测序(VIP-Seq)鉴定和定量活性原噬菌体
Identification and quantification of active prophages using
Virion Induction Profiling Sequencing (VIP-Seq)
我们使用VIP-Seq来鉴定和定量过夜细菌培养物上清液中的原噬菌体。简而言之,将细菌上清液浓缩并进行DNA酶消化,然后对包封的DNA进行定量,并将读序比对回细菌宿主(图1)。
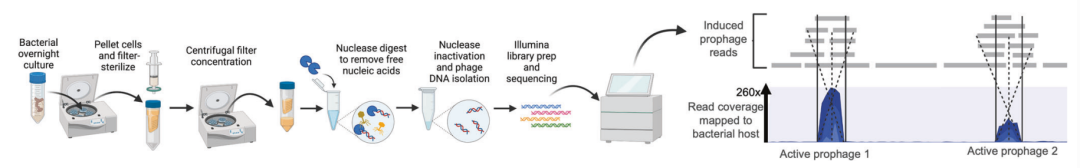
图1 用于鉴定和定量细菌培养物中活性原噬菌体的VIP-Seq工作流程。
由BioRender.com创建。
- 结果 -
来自叶际的欧文氏菌和假单胞菌菌株含有高滴度自发诱导的多种原噬菌体
Erwinia and Pseudomonas strains from the phyllosphere
harbour diverse prophages spontaneously induced at
high titres
许多自发诱导的噬菌体(Spontaneously Induced Prophages, SIPs)是在隔夜培养中高度诱导的,滴度高达3*108/毫升的病毒粒子(图2A-B)。在一些菌株中,病毒粒子滴度甚至与菌落形成单位(CFU)计数相媲美;在蚜虫Z9_1株中,聚集的病毒粒子/CFU比率为0.32(图2C)。
欧文氏菌和假单胞菌的SIP含量有显著差异(图2,A-C)。在中位数中,菌株含有2个SIP,而aphidicola菌株Z9_3最多,含有4个SIP。相比之下,六种假单胞菌菌株在过夜培养中似乎缺乏SIPs。欧文氏杆菌的病毒粒子聚集滴度(中位数(IQR)6.5*107(2.4-12)*107个病毒粒子/毫升)高于假单胞菌(中位数(IQR)5.0*105(2.9-12)*105个病毒粒子/毫升)。尽管在调整CFU浓度后这些差异有所减小(图2C),但它们仍然显著(p=8.9*10-4,Kruskal-Wallis秩和检验)
研究SIP基因组发现了许多共享的基因组片段,其中散布着未对齐的区域(图2D)。
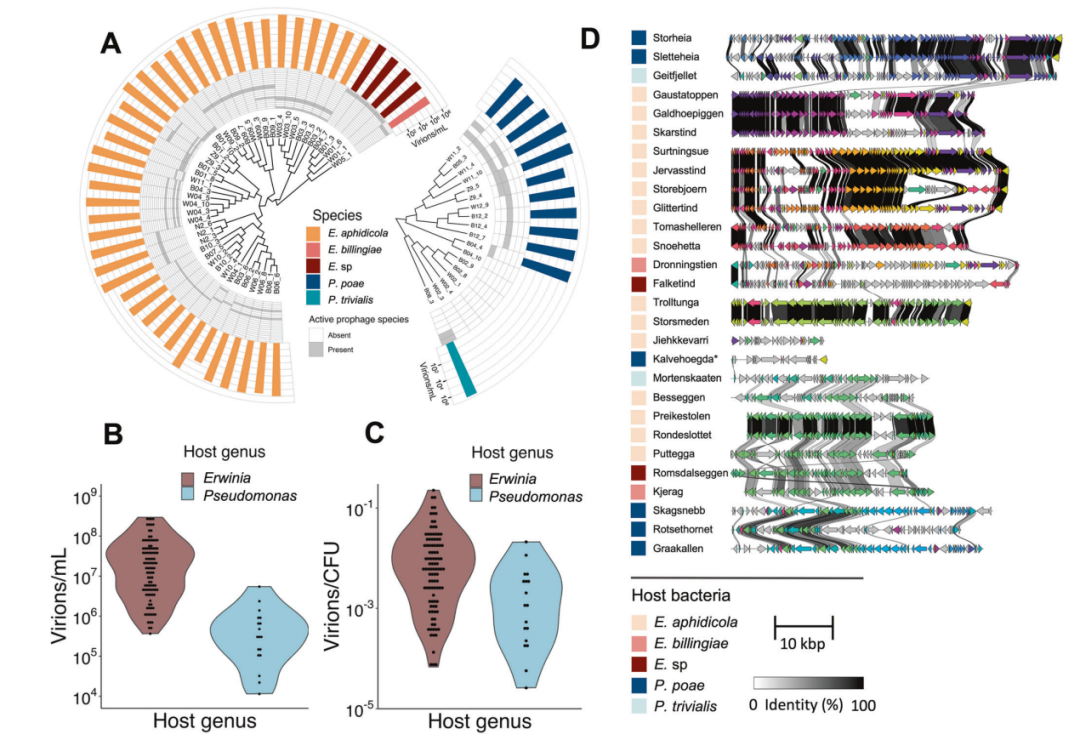
图2 细菌菌株及其SIP含量概述。
A 基于全基因组相似性(UPGMA聚类)的45种欧文氏菌和18种假单胞菌菌株的分支图。图中还显示了每个菌株中物种水平的活性噬菌体簇。最后,每个菌株过夜培养中的聚合病毒颗粒滴度以条形图显示。
B 所有120种欧文氏菌和假单胞菌活性原噬菌体的估计病毒数/毫升的小提琴图。
C 所有120种欧文氏菌和假单胞菌活性原噬菌体的估计病毒数/CFU的小提琴图。
D 来自28个已识别的原噬菌体物种级簇的代表,并标有每个簇的名称。Kalvehoegda簇标记为“*”,注释显示该区域可能是噬菌体卫星。接下来, clinker图显示了物种代表之间编码区的氨基酸排列。
VIP-Seq验证
Validation of VIP-Seq
除W02_4上清液外,VIP-Seq滴度与EPI计数符合良好,T4、B01_5和W01_1的EPI计数分别占EPI计数的54%、65%和19%(图3)。相比之下,PFU的可变性更大;当T4的斑块计数超过EPI计数时,B01_5和W01_1的PFU/EPI比率分别为4*10-2和2.8*10-5。由于这两个菌株都含有可能形成PFU的多个SIPs,我们对这两个指示菌株上的斑块进行了测序,以确定哪个SIP正在形成斑块(图3B,C)。在调整了过夜培养中噬菌斑的相对滴度后,调整后的PFU/EPI比率分别为1.05和1.3*10-4,表明PFU只能有条件地用于量化诱导的原噬菌体滴度。
特别的是来自Vibrant的非常长的原噬菌体基因组长度,部分原因是几对紧密排列的原噬菌体(即B01.5原噬菌体Glittertind_A和Skarstind_A,图3B)合并为单个原噬菌体预测。
在W02_4上清液中,VIP-Seq和空斑试验均为阴性,而EPI计数为2.5*104VLP/mL(图3A,D)。
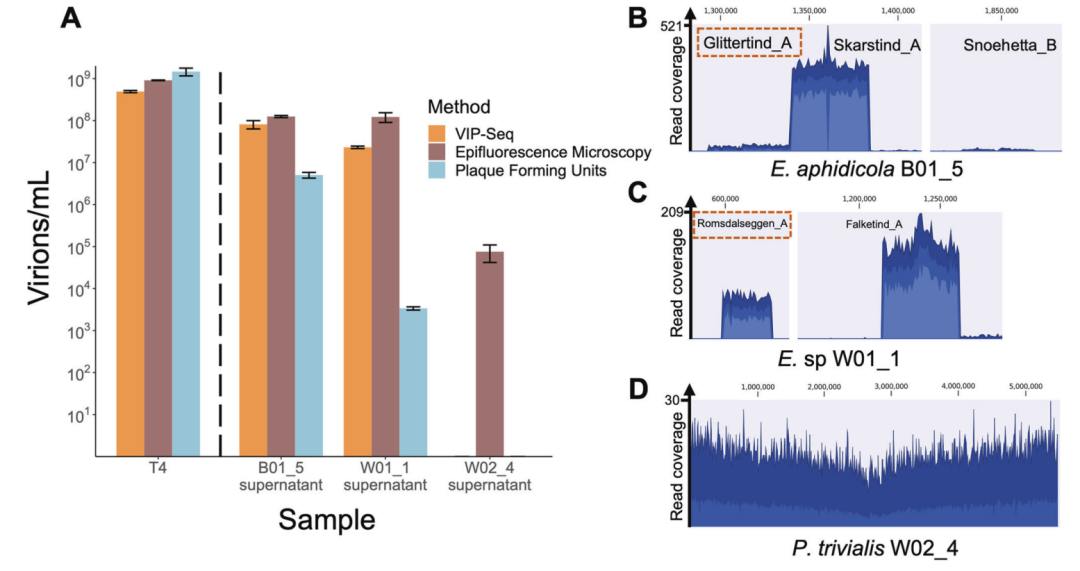
图3 VIP-Seq定量与斑块和荧光显微镜计数的比较。
A 用噬菌体滴度测定方法比较了T4强毒噬菌体、B01_5噬菌体、W01_1噬菌体和W02_4噬菌体四个菌株的噬菌体滴度。用VIP-Seq、荧光显微镜和空斑计数测定了B01.10(B01.5 SIPs)和B01.5(W01.1 SIPs)敏感菌株的噬菌体滴度。W02_4上清液对任何受试菌株都没有空斑。对于VIP-Seq,B01_5、W01_1和W02_4的上清在提取DNA之前用Amcon过滤器进行浓缩,与高滴度的T4不同。每个误差条代表三个技术重复的标准偏差。
B−D 分别对B01_5、W01_1和W02_4未处理的过夜培养上构建的VIP-seq文库reads覆盖图。B01_5和W01_1的噬菌体区域被放大和整理(保持相对比例),而全基因组显示在W02_4中。
能够进行细菌战的欧文氏菌宿主噬菌体
Erwinia host prophages capable of bacterial warfare
虽然许多欧文氏菌上清液中的SIPs在从相同环境中分离的竞争菌株上显示出广泛的宿主范围(图4A),但假单胞菌上清液未能产生单个可见的斑块,尽管观察到一些混浊清除区。寄主范围存在显著差异,欧文氏菌上清液在0个(蚜虫 B07.5)和27个(蚜虫 Z9.1和Z9.3)竞争菌株上留下印迹(图4B)。原噬菌体的敏感性也有相似的变化,从0(多个菌株)到30(蚜虫 E. N2.3)。还有至少两个广泛感染的例子,都是E.billingiae W05.1和新的E. sp菌株在蚜虫菌株上留下斑块。一些上清液在同一宿主上也显示出多种斑块形态(如图4B中最左边的三个点分别对应于W11.1、Z9.1和Z9.3的上清液,分别含有2、3和4个已鉴定的SIPs),表明可能有多个SIPs形成斑块。
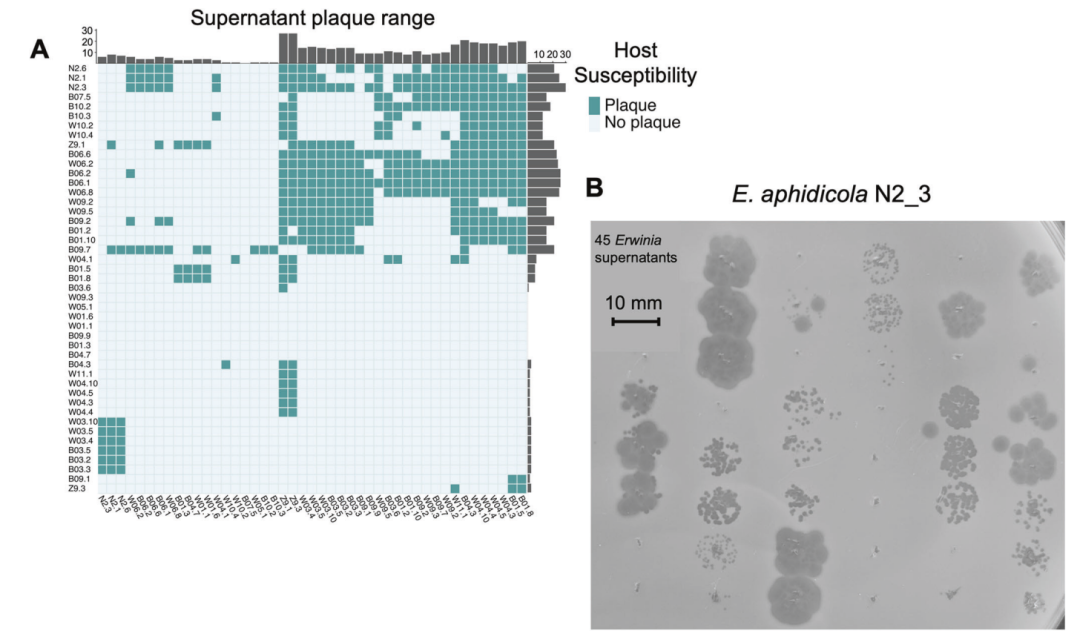
图4 欧文氏菌上清菌斑试验
A 热图显示了45 × 45欧文氏菌上清菌斑测定结果,上清宿主在垂直方向上显示,而宿主敏感性在水平方向上显示。显示累积宿主范围/敏感性的条形图与各自的轴相邻。
B 在蚜虫N2.3平板上对45个欧文氏菌上清进行菌斑分析,显示出广泛的易感性和多种菌斑形态。图像裁剪清晰。
在植物中观察到高水平的原噬菌体诱导
High levels of prophage induction observed in planta
接下来,对实验组和对照组的CFU和PFU(利用B01.10上的B01_5 SIP Glittertind_A斑块)进行了五天的监测(图5,补充表S7)。当无细胞对照的PFU在第二天降至检测下限以下时,B01_5处理的PFU在所有五天中保持相对稳定(图5B)。事实上,在第0天和第3天之间,PFU甚至在统计学上显著增加(P=0.04,Kruskal-Wallis秩和检验),在第5天下降。PFU的这种相对稳定性与B01_5计数的CFU计数形成对比,B01_5计数在第0天到第5天之间下降了近三个数量级。由于这些不同的趋势,B01_5处理的PFU/CFU比率在时间序列的过程中变化很大。尽管PFU/CFU开始时远低于体外过夜培养的PFU/CFU比率(4.7×10-3),但到了第三天,它已经攀升到体外比率的500倍以上(图5C),显示出非常高的植物诱导率。
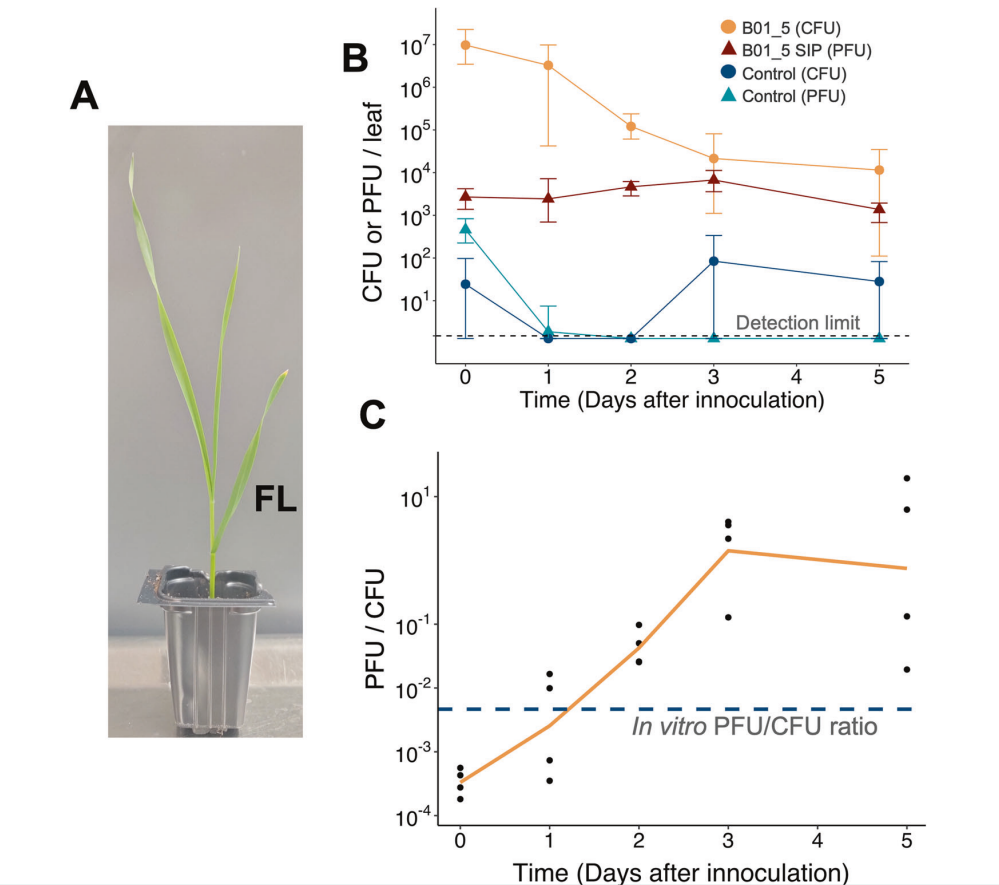
图5 E. aphidicola菌株在植物体内产生原噬菌体。
A 以12日龄第一片叶片标记为FL。
B CFU和PFU的小麦幼苗接种在12日龄Heerup幼苗第一片叶片上。第一个处理(B01_5)是在PBS缓冲液中洗涤蚜虫菌株 B01_5 两次,而第二个处理(对照)是洗涤的B01_5的无细胞上清液。将叶子在SM缓冲液中洗涤,并在假单胞菌选择琼脂上对菌落进行计数,同时使用以敏感菌株 B01_10作为宿主的软琼脂覆盖层对噬菌斑进行计数。每个数据点是四个生物重复的平均值,误差线代表最大/最小值。四个生物重复的平均值的检测限为1.5PFU或CFU/叶(用黑色虚线表示),为了清晰起见,低于此值的数据点人为设置为1.3。
C B01_5 细胞处理的PFU/CFU比率,以及蓝色虚线表示的体外记录的PFU/CFU比率。橙色线连接每个数据点的平均值,而所有数据点均显示为黑点。
将活性原噬菌体与生物信息学预测进行比较表明可能存在IS介导的原噬菌体失活
Comparing active prophages to bioinformatic predictions suggests possible IS-mediated prophage inactivation
检查工具是否正确预测了VIP-Seq识别的SIP,我们发现Vibrant预测117/120(0.98)和PHASTER预测109/120(0.91)(图6A)。PHASTER和VERIFIANT都没有预测到可能的噬菌体卫星Kalvehoegda_A。这两种工具的核苷酸精确度也相对较低,因为它们经常在VIP-Seq验证的前驱体内添加无关的宿主DNA。即使只考虑高置信度的原噬菌体预测,这些差异也是明显的,因为基因组大小和相对GC含量的分布与实验上活跃的原噬菌体不同(图6B,C)。除了预测>90%的SIPs外,PHASTER和Vibrant还预测了许多没有被观察到的SIPs的原噬菌体(图6D)。
令人惊讶的是,一半(53%)的PHASTER预测完全缺乏结构基因注释,因此不太可能是可信的原噬菌体;事实上,这些预测都不是SIP(图6E,补充表S9)。
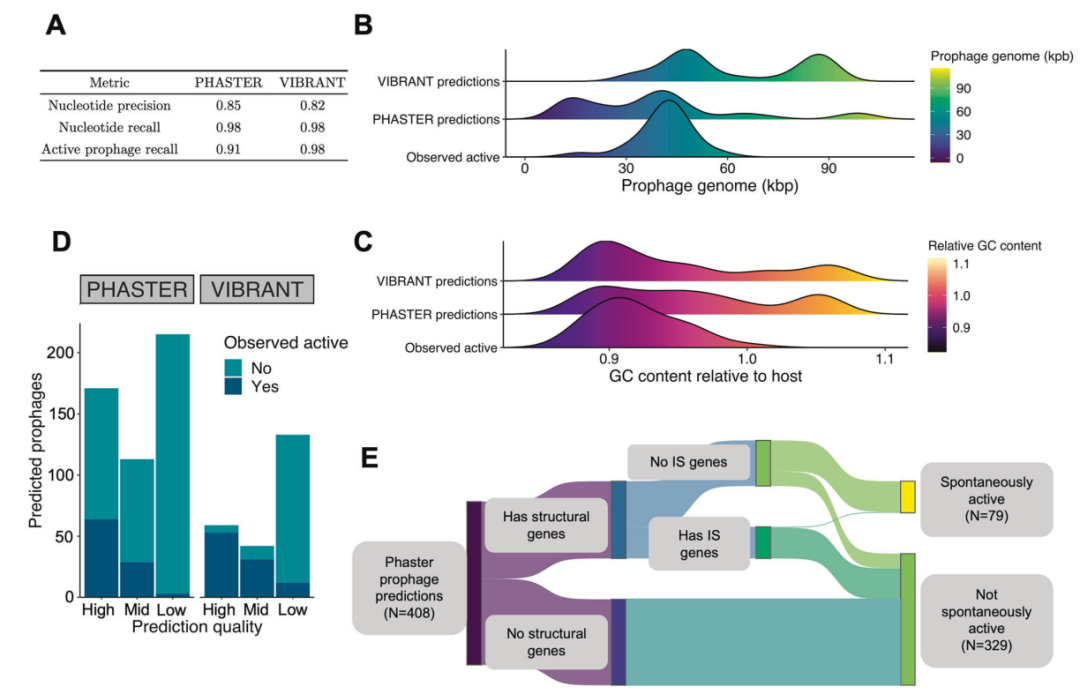
图6 SIPs的生物信息学分析
A 与使用VIP-Seq发现的120个SIPs相比,PHASTER和VIBRANT噬菌体预测的性能。第2.6节中详细介绍了性能指标。
B 用于高质量原噬菌体预测的原噬菌体区域大小与所有SIPs的比较分布。
C 与(B)相同,但原噬菌区GC含量与宿主菌相对。
D 柱状图显示PHASTER和VIBRANT的每种置信度预测中观察到自发活跃的噬菌体预测数量(按预测置信度依次标为 "高"、"中 "和 "低")。
E Sankey图表说明了来自40个蚜虫基因组的PHASTER原噬菌体预测的组成。
- 讨论 -
原噬菌体活动在农业中的影响可能是巨大的。尽管温和噬菌体通常被认为不适合生防,因为它们倾向于整合为原噬菌体,但我们发现许多叶际原噬菌体已经参与了细菌战。这些结果也强调了原噬菌体在设计有益微生物-合成菌群方面的重要性。尽管很少在这种情况下考虑,但许多合成菌群成员很可能含有活性的原噬菌体。其中一些可能会杀死致病菌株并促进有益群落的建立,而另一些可能会消除有益于植物的细菌或传播有害基因。无论如何,它们都不应被忽视。原噬菌体将传统的基于噬菌体的生物防治与传统的基于细菌的合成菌群相结合,为下一代植物微生物组工程和可持续农业提供了一个潜在的工具箱。
参考文献
Dougherty, P.E., Nielsen, T.K., Riber, L. et al. Widespread and largely unknown prophage activity, diversity, and function in two genera of wheat phyllosphere bacteria. ISME J 17, 2415–2425 (2023). https://doi.org/10.1038/s41396-023-01547-1
- 作者简介 -
通讯作者

哥本哈根大学
Lars Hestbjerg Hansen
教授
Lars Hestbjerg Hansens 环境微生物基因组学小组拥有丰富的专业知识,能够利用内部高通量和全基因组测序(Illumina 和 Nanopore 测序仪)对原核生物和真核生物进行高度定制化的基因组和宏基因组研究。该小组利用基于序列分析的信息来描述关键物种的基因组特征及其在自然和人工微生物群落和宏基因组中的流行情况,包括噬菌体信息以及其他移动遗传因子如何影响微生物进化和性状。小组活动涵盖自然、农业和健康相关系统中的微生物。
研究方向:
作为农业生物控制剂的叶球枢纽物种和噬菌体
噬菌体基因组中改变碱基的使用
通过FMT或噬菌体疗法操纵肠道微生物组
抗生素耐药性的基因起源
基因寄生虫的流动性及其在细菌进化中的作用
微生物组学、移动组学和病毒组学
细菌和噬菌体的表观遗传学
实验室官网:https://plen.ku.dk/forskning/mikrobiel-oekologi-og-bioteknologi/environmental-microbial-genomics/
猜你喜欢
iMeta简介 高引文章 高颜值绘图imageGP 网络分析iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文,跳转最新文章目录阅读