文章目录
前言
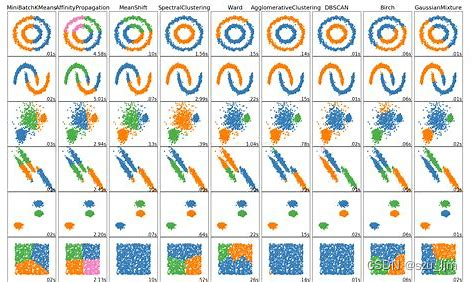
机器学习中有大量的分类任务,除了常见的分类算法能解决这些问题,还有经典的聚类算法来添砖加瓦,聚类和分类其实差不多,本质上都是为了将不同的数据分成不同的类别。不同的是,分类算法都是有监督学习或半监督学习,而聚类算法基本上都是无监督学习,即在没有标签的情况下进行数据分类,所以很多聚类的效果都不尽如人意。
一、常见的聚类算法简介
聚类是一种常见的无监督学习方法,能够按照某些特定的标准、特征将一个数据集分割成不同的类或者聚簇,在同一个类或者聚簇里的数据差异性尽可能小,不在同一个类或者聚簇的数据差异性尽可能的大。
a、划分聚类
划分聚类最大的特点就是聚出来的类的数量是已知的,它通过事先指定聚类中心点,通过反复迭代,最终达到聚簇内数据点足够近,聚簇间数据点足够远的目标。常见的划分聚类有:k-means、k-means++、bi-kmeans等
b、层次聚类
层次聚类是一种聚类分析方法,它将数据集中的对象按照相似性逐步合并到不同的层次结构中。在层次聚类中,每个数据点开始被视为单个聚类,然后它们被合并成更大的聚类,直到所有数据点都被分配到一个聚类中。
层次聚类有两种不同的方法:凝聚聚类(Agglomerative)和分裂聚类(Divisive)。在凝聚聚类中,每个数据点开始被视为单独的聚类,然后通过计算它们之间的相似性来合并聚类。最终形成一个大的聚类。在分裂聚类中,所有数据点被视为一个大的聚类,然后通过计算它们之间的不相似性来分割聚类,最终形成一组小的聚类
c、密度聚类
密度聚类是一种基于数据点之间密度的聚类方法,其目标是将具有高密度的数据点分为一组,并将它们与低密度区域中的其他数据点分开。在密度聚类中,数据点的密度是通过计算每个数据点周围的邻居数量来确定的。具有足够高密度的点被认为是“核心点”,而那些周围没有足够多邻居的点则被认为是“噪声点”。密度聚类通过寻找核心点及其邻居来形成簇。簇的数量和形状是基于密度和距离的阈值设置来控制的。密度聚类是一种非参数聚类方法,可以在没有事先确定簇数的情况下对数据进行聚类,适用于具有复杂结构和噪声的数据集。常见的密度聚类算法有DBSCAN算法
二、两种聚类的数学原理
1. K-MEANS聚类

a、样本点分类
假设我们手上有很多样本点数据,但我们不知道它们的标签。首先我们要先确定聚类之后的簇的个数 k k k,然后在相同维度的平面上随机生成 k k k 个质心,或者我们也可以指定质心的初始向量。接着我们要度量每个样本点到不同质心的距离,用该距离来衡量样本点与哪个质心相似度更高,并将该点分到离他最近的质心的簇中,常见的距离衡量方法有:
欧式距离
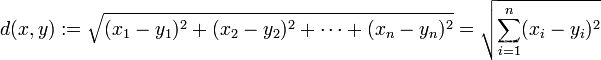
曼哈顿距离
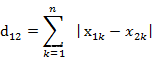
余弦相似度(将一个样本数据看成一个向量)
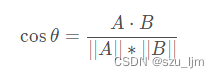
b、质心更新迭代
当所有的数据样本点被聚类到不同的簇后,质心要进行一次更新,对该簇中的所有样本点向量进行不同维度的均值计算,每个维度的均值就是更新后质心的向量 c i c_{i} ci,其中 i i i 是第 i i i 个簇, n n n表示向量的不同维度
c i ⃗ = ( ∑ j = 1 m i x i j 1 m i , ∑ j = 1 m i x i j 2 m i , ∑ j = 1 m i x i j 3 m i , ⋯ , ∑ j = 1 m i x i j n m i ) , i = 1 , 2 , 3 , ⋯ , k \vec{c_{i}} = (\frac{\sum_{j=1}^{m_i}x_{ij}^{1}}{m_{i}},\frac{\sum_{j=1}^{m_i}x_{ij}^{2}}{m_{i}},\frac{\sum_{j=1}^{m_i}x_{ij}^{3}}{m_{i}},\cdots,\frac{\sum_{j=1}^{m_i}x_{ij}^{n}}{m_{i}}), i=1,2,3,\cdots,k ci=(mi∑j=1mixij1,mi∑j=1mixij2,mi∑j=1mixij3,⋯,mi∑j=1mixijn),i=1,2,3,⋯,k
当质心更新完后,就继续迭代这两步,先重新对样本点进行分类,再对质心进行更新,最后迭代到损失函数值基本趋于平稳就可以停止。这就是K-MEANS聚类实现的数学原理。
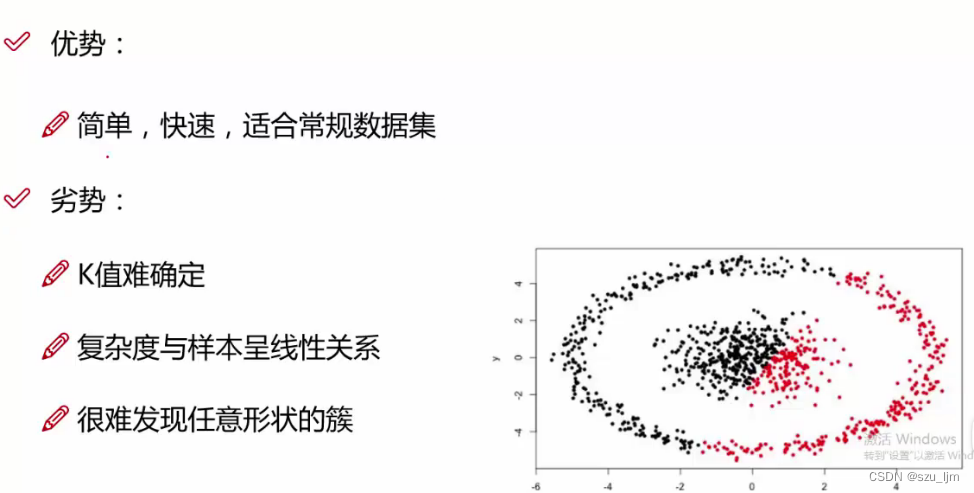
K-MEANS聚类算法的好处是实现起来快捷简单那个,比较适合常规基础的数据集,但一旦数据集分布比较复杂,这时候K-MEANS聚类的效果则远远不如DBSCAN聚类。而且K-MEANS聚类的结果与初始K值选择和初始质心位置选择有很大相关性,选不好效果差强人意
2. DBSCAN聚类
DBSCAN聚类是基于密度可达性进行聚类,在数学上如果某个点 p p p 在点 q q q 的 r r r 邻域内,即两个点的形成向量的在每个维度的分量都小于等于 r r r,这就是直接密度可达

假设我们手上有 m m m 个样本点,首先先随机取一个样本点作为一个簇出发点,然后以给定阈值 r r r 和特征维度 n n n 进行扫描,将进入扫描范围的样本点囊括进来,被扫描到的样本点会作为新的出发点进行下一次扫描,以此类推,直到该簇的样本点数量不再增加 ,然后进行下一个簇的聚类


DBSCAN聚类相较于K-MEANS聚类有很多优势,比如可以处理分布比较复杂的数据集,擅长找到离群点噪音点,因为不需要指定簇的个数,所以DBSCAN聚类的效果只会受到聚类半径的影响。但如果聚类半径选择不当,也会影响DBSCAN聚类的结果,最好多实验几次,画出不同半径下的的损失函数取最优解

3. 两种评估指标
inertia指标可用于衡量K-MEANS算法的聚类效果,它表示的是每个样本点到其所在簇质心的距离之和。 按照inertia的定义来说inertia是越小越好,但是实际上我们可以发现当分簇的数量越来越多的时候inertia的值自然就会越来越小。 甚至再极端的情况下,簇的数量等于样本数的时候,每一个样本就是一个簇,那么inertia就为0。 所以说inertia虽然是一个不错的衡量标准,但却很难界定最优的聚类效果,毕竟在无监督的情况下我们很难具体衡量聚类效果的好坏。
s ( i ) = b ( i ) − a ( i ) max ( ( a ( i ) , b ( i ) ) ) s(i) = \frac{b(i)-a(i)}{\max((a(i),b(i)))} s(i)=max((a(i),b(i)))b(i)−a(i)
轮廓系数 s ( i ) s(i) s(i),是用于评价聚类效果好坏的一种指标。可以理解为描述聚类后各个类别的轮廓清晰度的指标。其包含有两种因素——内聚度和分离度,内聚度可以理解为反映一个样本点与类内元素的紧密程度,分离度可以理解为反映一个样本点与类外元素的紧密程度,其中 a ( i ) a(i) a(i) 表示样本 i i i 到同一个簇的其他样本的平均距离,其中 b ( i ) b(i) b(i) 表示样本 i i i 到另一个簇的其他样本的平均距离。当 s ( i ) s(i) s(i) 越趋近于1,表示该样本所在簇的密集度越高,聚类效果较好;当 s ( i ) s(i) s(i) 越趋近于0,表示该样本位于簇的边缘;当 s ( i ) s(i) s(i) 越趋近于-1,表示该样本所在簇的密集度越低,聚类效果较差,很有可能该样本点被聚错簇了
三、Python实现聚类算法
1. K-MEANS聚类和评估
Python实现K-MEANS聚类首先导入几个常用库和生成几个块状数据群,块的中心、数据量、分布离散程度和块的个数可以自己指定,接着定义质心绘制函数和数据样本点绘制函数,然后定义决策边界绘制函数,其中包括绘制棋盘、分类边界和质心绘制函数的调用,然后实例化K-MEANS并对模型进行训练,实例化要制定簇的个数、质心初始化状态和迭代次数,最后我们导入轮廓系数作为指标来衡量在给定的不同簇的个数中K-MEANS的聚类效果
import numpy as np
import matplotlib.pyplot as plt
from sklearn.datasets import make_blobs
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_score
k = 5
kmeans_per_k = []
blob_centers = np.array([[0.3, 1.1], [-0.2, -0.4], [0.6, -1.8], [1.3, 0.2], [-1.0, 0.8]])
blob_std = np.array([0.32, 0.25, 0.18, 0.12, 0.12])
x, y = make_blobs(n_samples=3000, centers=blob_centers, cluster_std=blob_std, random_state=6)
def plot_clusters(x, y=None):
plt.plot(x[:, 0], x[:, 1], 'k.', markersize=2)
plt.xlabel("x_1", fontsize=12)
plt.xlabel("x_2", fontsize=12)
def plot_centroids(centroids, circle_color='w', cross_color='w'):
plt.scatter(centroids[:, 0], centroids[:, 1], marker='o', s=10, linewidths=6, color=circle_color, zorder=10, alpha=0.8)
plt.scatter(centroids[:, 0], centroids[:, 1], marker='x', s=18, linewidths=20, color=cross_color, zorder=10, alpha=1)
def plot_decision_boundaries(clusterer, X, resolution=2000, show_centroids=True):
mins = X.min(axis=0) - 0.1
maxs = X.max(axis=0) + 0.1
xx, yy = np.meshgrid(np.linspace(mins[0], maxs[0], resolution), np.linspace(mins[1], maxs[1], resolution))
Z = clusterer.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contourf(Z, extent=(mins[0], maxs[0], mins[1], maxs[1]), camp="Pastel2")
plt.contour(Z, extent=(mins[0], maxs[0], mins[1], maxs[1]), linewidths=1, color='k')
plot_clusters(X)
if show_centroids:
plot_centroids(clusterer.cluster_centers_)
kmeans = KMeans(n_clusters=k, init='random', n_init=5, random_state=2000)
kmeans_per_k = [KMeans(n_clusters=i).fit(x) for i in range(2, 8)]
y_pred = kmeans.fit(x)
silhouette_score(x, kmeans.labels_)
silhouette_scores = [silhouette_score(x, model.labels_) for model in kmeans_per_k[1:]]
plt.figure(figsize=(12, 8))
plt.subplot(121)
plot_decision_boundaries(kmeans, x)
plt.subplot(122)
plt.plot(range(2, 7), silhouette_scores, 'go--')
plt.show()
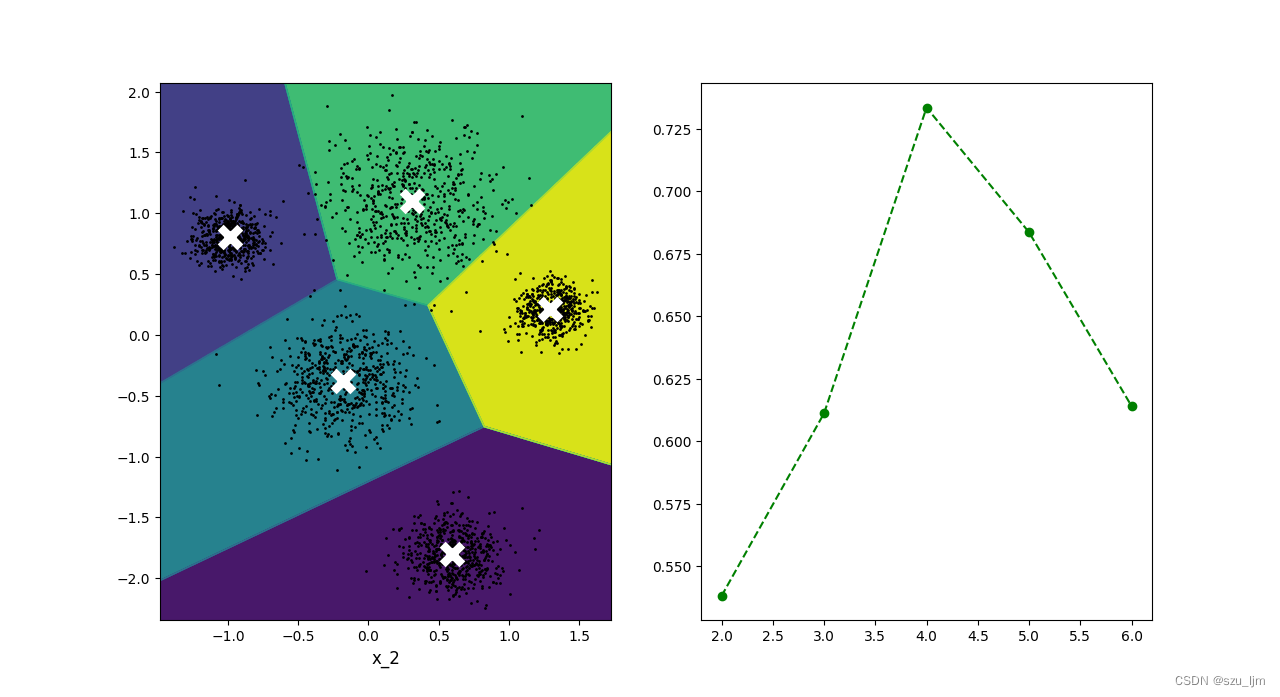
从轮廓系数的折线图可以看到最优的簇个数是在4附近,但我们人为设定的数据块个数有5个,所以轮廓系数只能作为一个借鉴指标来帮助你估量最优的K值
2. K-MEANS聚类对比DBSCAN聚类
Python实现DBSCAN聚类和K-MEANS聚类差不多,这里我们拿类似月亮的分布比较复杂数据集来做对比,DBSCAN聚类绘图需要绘制样本点分布、噪音点和聚类扫描边界,实例化时需要传入聚类半径和簇的最小样本点数量,实例化完后进行模型训练
import numpy as np
import matplotlib.pyplot as plt
from sklearn.datasets import make_moons
from sklearn.cluster import KMeans
from sklearn.cluster import DBSCAN
k = 2
blob_centers = np.array([[0.3, 1.1], [-0.2, -0.4], [0.6, -1.8], [1.3, 0.2], [-1.0, 0.8]])
blob_std = np.array([0.32, 0.25, 0.18, 0.12, 0.12])
x, y = make_moons(n_samples=3000, noise=0.1, random_state=56)
def plot_clusters(x, y=None):
plt.plot(x[:, 0], x[:, 1], 'b.', markersize=2)
plt.xlabel("x_1", fontsize=12)
plt.ylabel("x_2", fontsize=12)
def plot_centroids(centroids, circle_color='w', cross_color='w'):
plt.scatter(centroids[:, 0], centroids[:, 1], marker='o', s=10, linewidths=6, color=circle_color, zorder=10, alpha=0.8)
plt.scatter(centroids[:, 0], centroids[:, 1], marker='x', s=18, linewidths=20, color=cross_color, zorder=10, alpha=1)
def plot_decision_boundaries(clusterer, X, resolution=2000, show_centroids=True):
mins = X.min(axis=0) - 0.1
maxs = X.max(axis=0) + 0.1
xx, yy = np.meshgrid(np.linspace(mins[0], maxs[0], resolution), np.linspace(mins[1], maxs[1], resolution))
Z = clusterer.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contourf(Z, extent=(mins[0], maxs[0], mins[1], maxs[1]), camp="Pastel2")
plt.contour(Z, extent=(mins[0], maxs[0], mins[1], maxs[1]), linewidths=1, color='k')
plot_clusters(X)
plt.title("K-MEANS")
if show_centroids:
plot_centroids(clusterer.cluster_centers_)
def plot_dbscan(dbscan, X, size):
core_mask = np.zeros_like(dbscan.labels_, dtype=bool)
core_mask[dbscan.core_sample_indices_] = True
anomalies_mask = dbscan.labels_ == -1
non_core_mask = ~(core_mask | anomalies_mask)
cores = dbscan.components_
anomalies = X[anomalies_mask]
non_cores = X[non_core_mask]
plt.scatter(cores[:, 0], cores[:, 1], c=dbscan.labels_[core_mask], marker='o', s=size, cmap="Paired")
plt.scatter(cores[:, 0], cores[:, 1], c=dbscan.labels_[core_mask], marker='*', s=15)
plt.scatter(anomalies[:, 0], anomalies[:, 1], c='r', marker='x', s=100)
plt.scatter(non_cores[:, 0], non_cores[:, 1], c=dbscan.labels_[non_core_mask], marker='.')
plt.xlabel("x_1", fontsize=12)
plt.ylabel("x_2", fontsize=12)
plt.title("DBSCAN")
kmeans = KMeans(n_clusters=k, init='random', n_init=5, random_state=2000)
dbscans = DBSCAN(eps=0.1, min_samples=10)
y_kmeans_pred = kmeans.fit(x)
y_dbscan_pred = dbscans.fit(x)
plt.figure(figsize=(12, 8))
plt.subplot(121)
plot_decision_boundaries(kmeans, x)
plt.subplot(122)
plot_dbscan(dbscans, x, size=200)
plt.show()
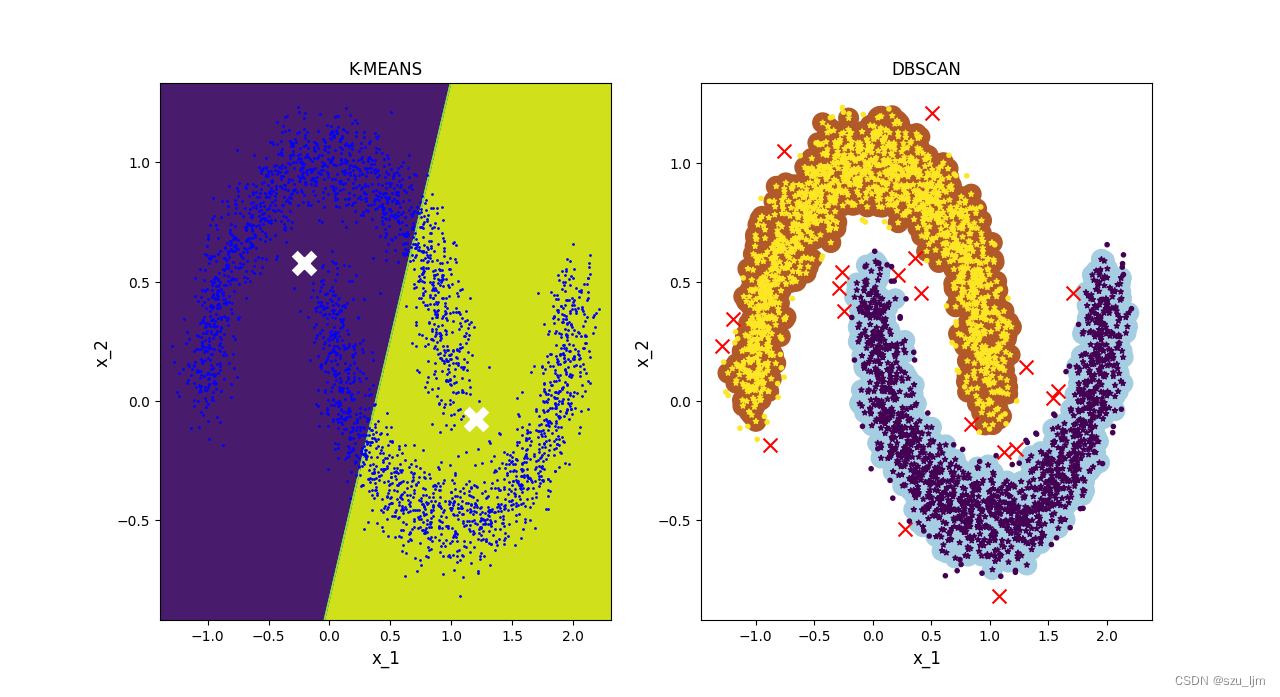
从上面两幅图可以明显看出来,在处理分布比较复杂的数据集时DBSCAN聚类的效果比K-MEANS聚类的效果好的多,而且DBSCAN聚类擅长找到离群点,在做检测任务的时候比较有用
3. K-MEANS图像分割
K-MEANS图像分割的核心原理就是基于距离的聚类,一个图像数据是由长度、宽度和颜色通道数组成的,每个对应的像素点都会存储RGB三个分量,RGB矩阵的每个元素取值范围为 [ 0 , 255 ] [0,255] [0,255],不同的RGB排列组合后可以构成 2 24 2^{24} 224 种颜色,这就还原我们人眼在现实生活中看到的颜色。K-MEANS图像分割是将每个像素点的颜色通道数按照给定簇的个数进行聚类,将同一簇的像素点填充为该簇质心的RGB三个分量值
import matplotlib.pyplot as plt
from matplotlib.image import imread
from sklearn.cluster import KMeans
image = imread("flower.jpg")
image = image / 255
division_images = []
x = image.reshape(-1, 3)
n_colors = (24, 18, 12, 6, 2)
for n_cluster in n_colors:
kmean = KMeans(n_clusters=n_cluster, random_state=42).fit(x)
division_image = kmean.cluster_centers_[kmean.labels_]
division_images.append(division_image.reshape(image.shape))
plt.figure(figsize=(12, 8))
plt.subplot(231)
plt.imshow(image)
plt.title('Original image')
for index, n_clusters in enumerate(n_colors):
plt.subplot(232+index)
plt.imshow(division_images[index])
plt.title('{}colors'.format(n_clusters))
plt.show()
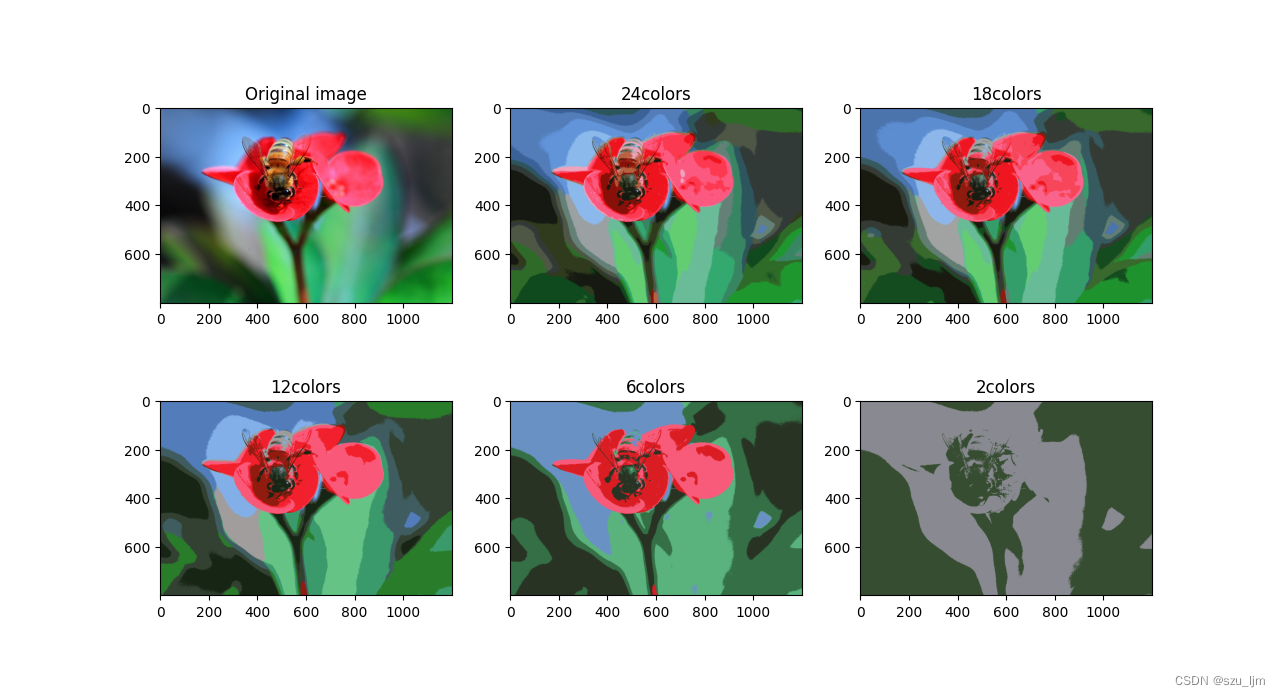
总结
以上就是机器学习聚类算法的学习笔记,本篇笔记简单的记录了聚类算法的数学原理和Python实现思路,虽然大多数情况下聚类算法的实现效果都不如分类算法好,毕竟聚类是无监督学习而分类是有监督学习,但是聚类算法在特定的应用案例中仍然能发挥优势,不如K-MEANS聚类在图像分割中实现的结果比较理想,总之我们需要掌握不同算法自身的优势,扬长避短,相得益彰!