
期刊:Plant Science
影响因子:5.363
发表时间:2022
样本类型:果皮
客户单位:南京农业大学
凌恩生物客户南京农业大学吴俊团队发表在《Plant Science》上的文章“DNA methylation reprogramming provides insights into light-induced anthocyanin biosynthesis in red pear”,本研究阐述了光诱导下梨果皮中DNA甲基化和基因表达的变化以及两者之间的相关性,这些结果为光诱导花青素生物合成中DNA的甲基化机制提供了新的见解。
一、研究背景
DNA甲基化是一种表观遗传标记,认为能够调节植物花青素的生物合成。众所周知,光诱导花青素积累,套袋处理通常用于研究光控制的花青素生物合成。目前关于光诱导花青苷积累的研究主要集中在转录因子对结构基因的调控,而基因组水平的DNA甲基化的动态景观尚未得到很好的表征。
二、实验设计
本研究对中国沙梨品种“云红一号”采用套袋(取样前果实在连续黑暗下生长)和除袋(取袋后果实再暴露在阳光下)处理,并进行了甲基组分析和RNA测序。基于此研究的结果和前人的研究结果,作者提出了光诱导花青苷积累的调控模型(如下图)。
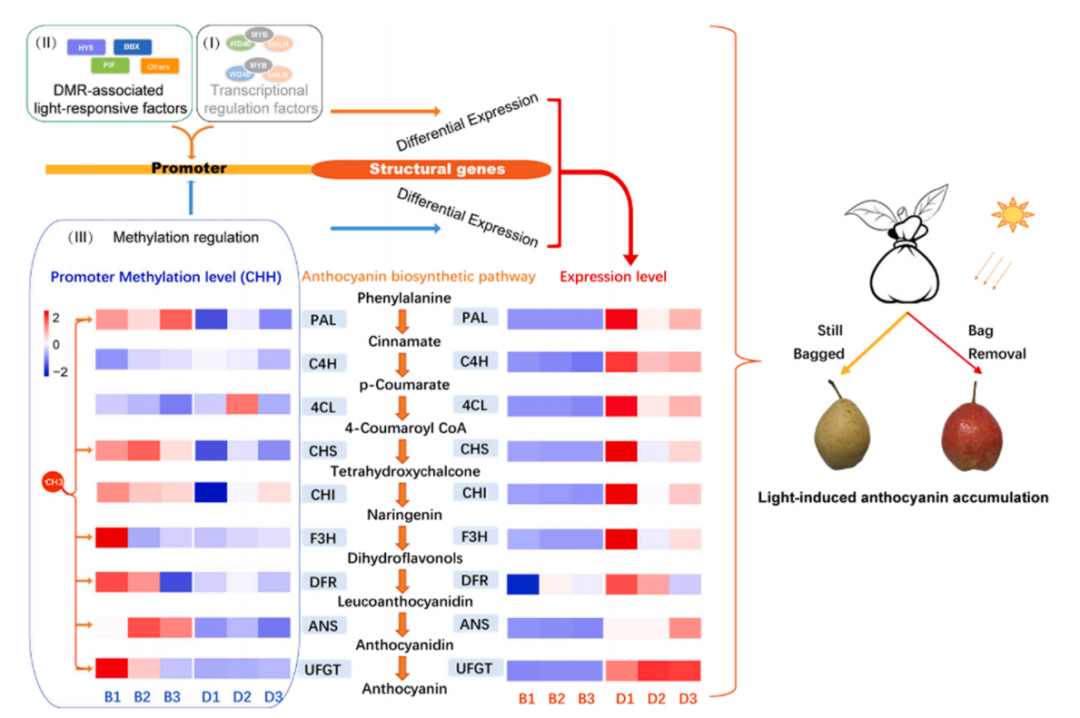
图1 光诱导花青素积累的调控模型
三、实验结果
1、套袋和除袋处理梨果实颜色变化及整体甲基组分析
梨品种“云红一号”的果实在开花后35天(DAFB)套袋,直到200 DAFB(收获前10天)除袋,除袋后果实皮颜色变红(图2A)。随着光照时间的延长,除袋后果实的花青素含量逐渐增加(图2B)。分别对除袋后不同时间下样本(D1、D2、D3)和相应时间下(B1、B2、B3)仍套袋处理的样本,进行全基因组甲基化测序(WGBS)。
研究发现,上下游区CHG和CHH甲基化水平均高于基因体(gene body)(图2C)。CG甲基化水平在转录起始位点和转录终止位点(TSS和TTS)附近较基因体以及上游和下游区域降低。DNA甲基化被认为是转座因子(TE)活性的最典型的调节因子,因此,本研究进一步分析了它们在光诱导的花青素积累过程中的甲基化状态,结果表明,在这三种情况下,TE区域的甲基化水平都高于它们的侧翼区域(图2C)。

图2 DNA甲基化分析结果。A.“云红一号”套袋及除袋后果实的表型。B.“云红一号”套袋及除袋后果实的花青素含量。C. mCG、mCHG和mCHH邻近基因和TEs的DNA甲基化分析。D. DMR、TEs和基因的密度图。E. 三种情况下DMRs的DNA甲基化状态。F. 不同比较中DMRs的数量。G. 在不同的光照条件下,重叠或唯一的DMRs的数量。
2、光再暴露诱导花青素生物合成过程中甲基化的再分布模式
甲基化水平和低甲基化和高甲基化DMRs (hypod - DMRs和hyperd - DMRs)的数量表明,光再暴露诱导花青素生物合成过程中甲基化状态的重新分布,尤其是在mCHH(图2E)。染色体上甲基化水平的分布表明,套袋组CHH明显发生高甲基化。进一步的研究表明,在套袋组中,CG和CHG环境中的甲基化水平在商业成熟时下降。三个着色阶段的CG和CHG没有重叠。只有少数CHH-DMRs在不同的阶段中共享,分别为59个和84个(图2G)。
3、DMR介导的DEGs参与了光诱导的花青素生物合成
对来自套袋和除袋处理的果皮样本进行转录组测序。相对于套袋组,除袋组样本(D1、D2和D3)共鉴定出2957个、3529个和3596个上调基因,1818个、3141个和3340个下调基因(图3A)。三组共有DEGs 2568个,其中上调表达基因1714个,下调表达基因805个(图3B)。通过对DEGs的热图聚类分析,2545个DEGs根据其表达模式分为三个聚类,显示了整体转录组的变化(图3C),以上结果表明,这些基因的差异转录水平与甲基化水平的波动密切相关。

图3 DEGs的表达模式和甲基化水平特征。
采用Pearson相关系数的计算结果来评价甲基化水平动力学与转录本水平变化之间的关系。在三次比较中,共鉴定出310个DMR介导的基因在共同的DEGs中重叠,其中包括217个含有低甲基化启动子的上调DEGs和93个含有高甲基化启动子的下调DEGs。310个DMR介导的DEGs的甲基化组和转录组水平之间的相关性热图显示,DMR介导的DEGs的甲基化水平与其表达水平呈负相关。
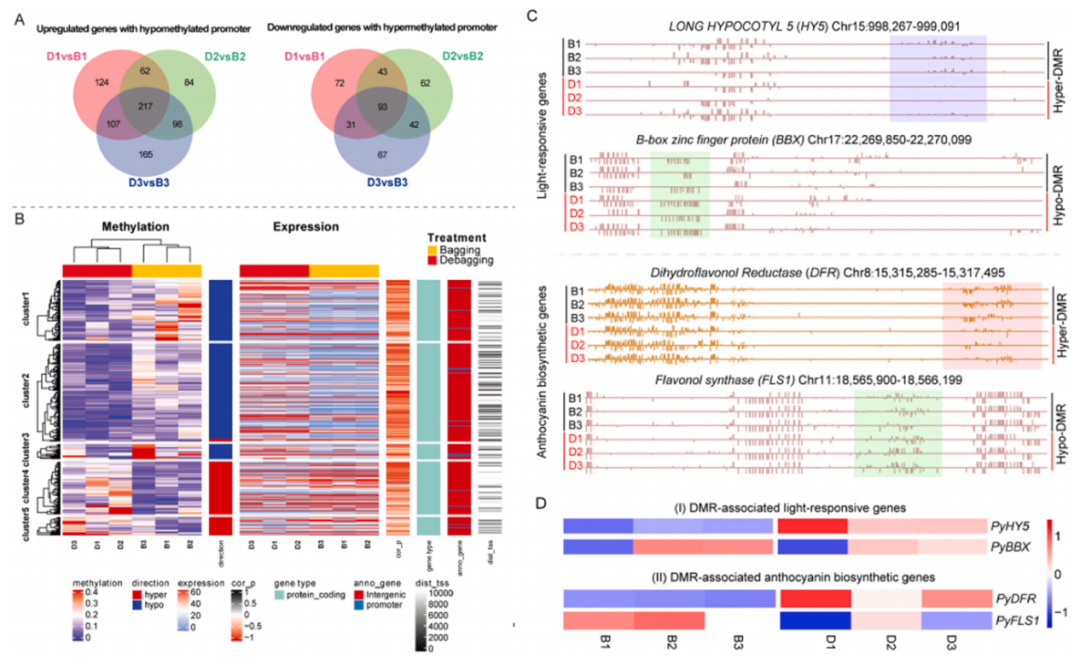
图4 DMR介导的基因的甲基化和表达水平。
4、PyUFGT在除袋果实中表达水平的增加是由于启动子区域的低甲基化
PyUFGT酶是一种重要的糖基转移酶,有报道称它可以催化不稳定的花色素转化为花青素,这对于梨颜色的稳定性至关重要。PyUFGT(Pbr001279.1)在310个DMR介导的DEGs中被发现。PyUFGT属于一组具有低甲基化启动子的上调基因(图4A),在除袋水果中表达增加(D1、D2、D3)。根据WGBS的分析结果,在PyUFGT启动子区域发现了一个DMR(图5A)。以该基因为代表,对PyUFGT启动子进行BS-PCR,并计算甲基化率。除袋后,PyUFGT启动子在−2689~−2190 bp区域的甲基化水平较低,甲基化环境为“mCHH”(图5B,C)。同时,与套袋水果相比,脱袋果皮中PyUFGT的表达明显增加(图5C)。因此,光诱导花青苷生物合成过程中的糖基化可能受到甲基化的影响。
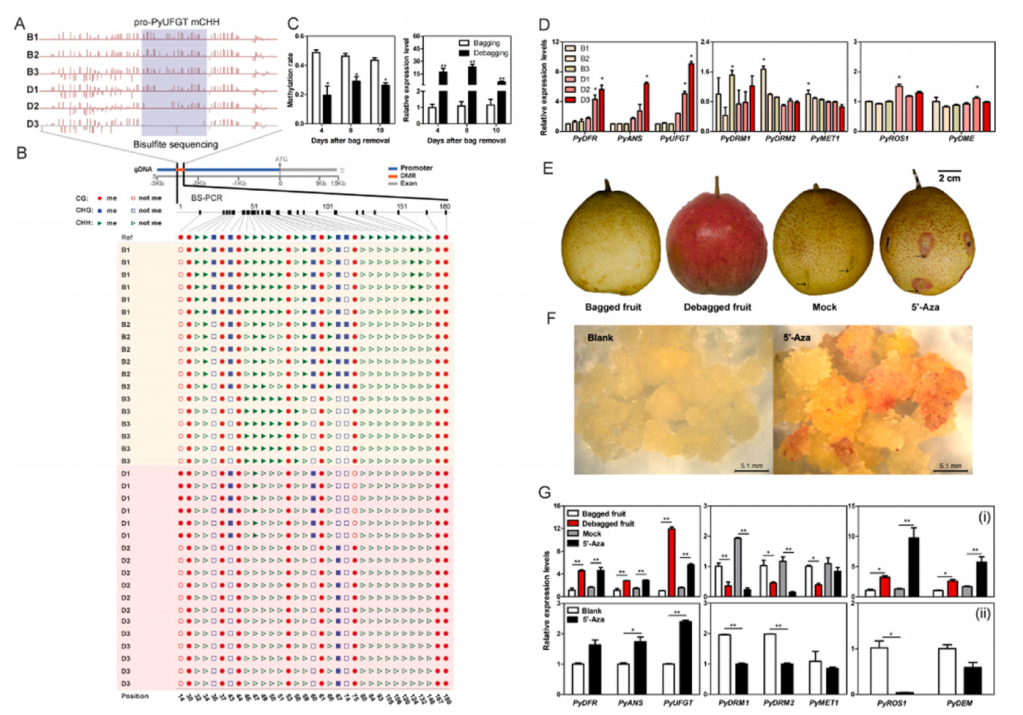
图5 PyUFGT启动子的甲基化分析及5ʹ-Aza处理对花青素生物合成和甲基化修饰相关基因表达模式的影响。
5、甲基化抑制剂5ʹ-Aza诱导梨的红色色素沉积
为了研究套袋和除袋果皮中DNA甲基化水平的差异是由DNA甲基转移酶还是去甲基化酶引起的,对这些酶的基因表达谱进行了分析。通过基因组注释和转录组分析,在梨中分别鉴定出拟南芥的同源基因,包括2个PyDRM1、1个PyDRM2、1个PyMET1、2个PyROS1和7个PyDMEs。PyDRM1、PyDRM2和PyMET1在袋装果实中的表达量普遍较高。此外,PyDRM1在B3中表达量最高,而PyDRM2和PyMET1在B1中表达量最高(图5D)。DNA去甲基化酶同源物PyROS1和PyDME在除袋早期(D1或D2)的表达水平显著升高。
5ʹ-Aza是一种核苷抑制剂(胞苷的核苷类似物),有助于表观遗传变化,将“云红一号”套袋果实注射5ʹ-Aza,作为平行实验,梨愈伤组织在5ʹ-Aza固体培养基上处理梨愈伤组织。处理结果表明,5ʹ-Aza诱导了梨皮和愈伤组织的红色色素沉积,而模拟对照和空白对照均没有诱导(图5E、F)。
四、研究结论
本研究分析了不同条件下梨果皮着色时的DNA甲基化变化,在除袋处理和套袋处理之间,基因体/TE及其侧翼序列的DNA甲基化水平大致相似,但在光照后,甲基化差异区域(DMRs)重新建立。DNA去甲基化酶同源物和RNA定向的DNA甲基化(RdDM)途径都促成了这种重新分布。在套袋处理和除袋处理的光诱导花青素生物合成过程中,共有310个DEGs与DMR相关。在PyUFGT的启动子中可以看到低甲基化的mCHH环境,以及其他花青素生物合成基因(PyPAL、PyDFR和PyANS)。这增强了转录激活,促进了再次光照后花青素的积累。本研究观察了甲基化模式的全基因组再分布,表明芽突变或环境变化引起的花青素甲基化模式的不同机制。

参考文献
DNA methylation reprogramming provides insights into light-induced anthocyanin biosynthesis in red pear. Plant Science, 2022.