Uni-Mol: 一个通用的三维分子表示学习框架
ICLR 2023
Uni-Mol 论文:Uni-Mol: A Universal 3D Molecular Representation Learning Framework | OpenReview
Uni-Mol 代码::GitHub - dptech-corp/Uni-Mol: Official Repository for the Uni-Mol Series Methods
官方视频解析:Uni-Mol 分子3D表示学习框架和预训练模型 | 郑行(深势科技)| 青年科学半月谈_哔哩哔哩_bilibili
预训练模型正在席卷 AI 领域。从大规模无标注数据中提取表征信息,再在小范围标注的下游任务上进行监督学习,正在成为很多领域的事实解决方案。NLP 中有 BERT、GPT-3,CV 中有 ViT,而这样的模式如何助力药物设计,也一直都是人们密切关注的问题。药物分子与图片、语言文字的不同之处在于,“什么是最好的分子表征”依旧是一个人们未能形成共识的问题。主流分子预训练模型均从一维序列或二维图结构出发,但分子结构本身是在三维空间中表示的。能否直接从三维信息出发构建预训练模型、获得更好的分子表征,是一个重要而有意义的问题。
深势科技团队又发布了首个三维分子预训练模型 Uni-Mol。Uni-Mol 直接将分子三维结构作为模型输入,而非采用一维序列或二维图结构。从三维信息出发的表征学习让 Uni-Mol 在几乎所有与药物分子和蛋白口袋相关的下游任务上都超越了 SOTA(state of the art),也让 Uni-Mol 得以能够直接完成分子构象生成、蛋白-配体结合构象预测等三维构象生成相关的任务,并超越现有解决方案。
01 Uni-Mol框架
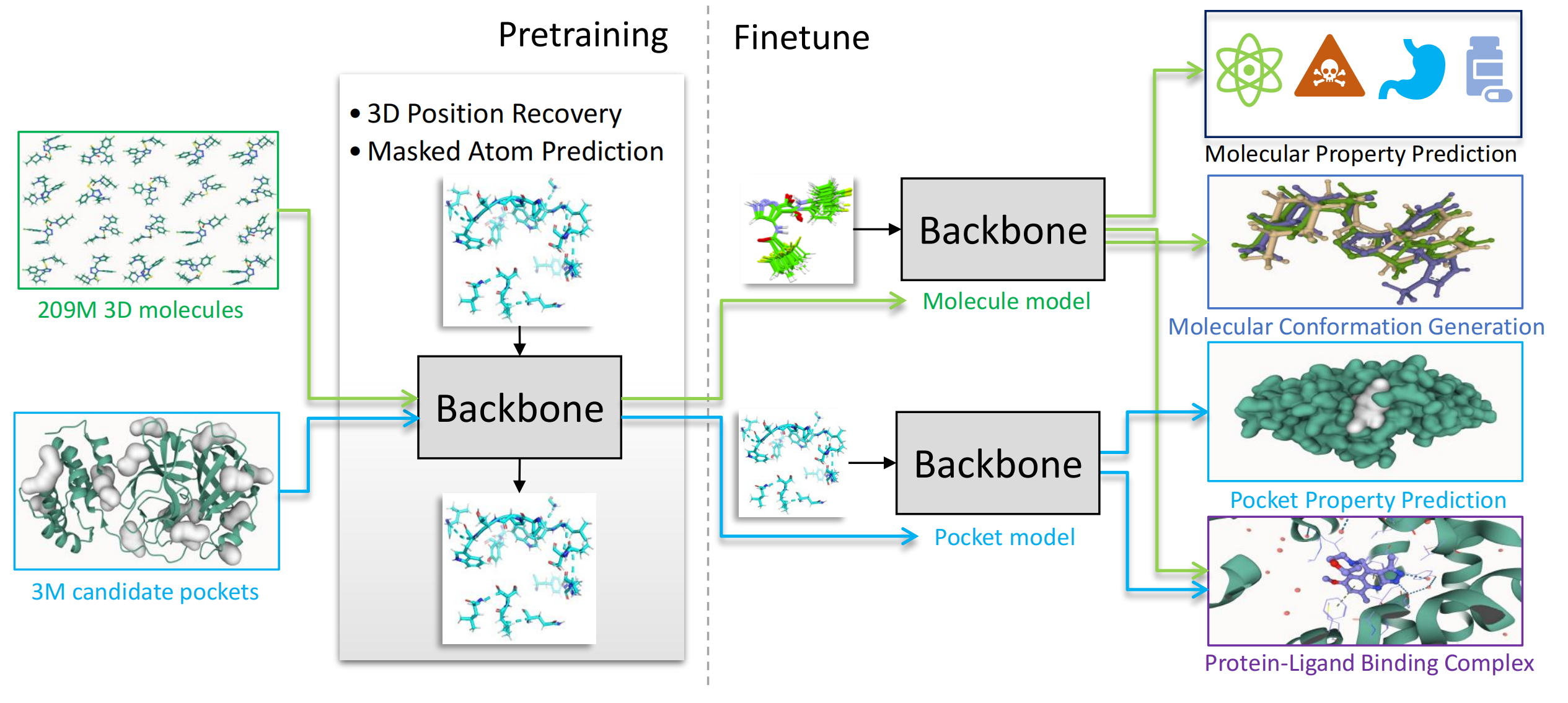
图1. Uni-Mol框架示意图。 ▲ Uni-Mol由两个模型组成:一个是由2.09亿分子三维构象训练的分子预训练模型;一个是由3百万候选蛋白口袋数据训练的口袋预训练模型。这两个模型独立用于不同的任务,在蛋白质-配体结合任务上两个都用。

图2. Uni-Mol模型架构。 左图:整体预训练架构,包括输入和预训练设计的任务;
中间:模型结构,包括模块连接和3D结构编码;
右图:模型基本单元,包括表征以及更新过程
1.1 处理3D空间信息的Transformer
1.1.1 旋转平移不变的空间位置编码
由于 Transformer 有置换不变性,它在没有位置编码的情况下无法区分输入的具体位置,而且位置编码需要在全局旋转和平移的情况下保持不变。不同于基于离散值的位置编码,分子的 3D 信息,即三维空间中的坐标是连续值。要保证它对旋转和平移的不变性,类似相对位置编码,深势科技团队简单地使用所有原子对的欧氏距离,融合分子图中边的类型,之后经过高斯核函数得到位置编码,形式上可以表达成如下公式:
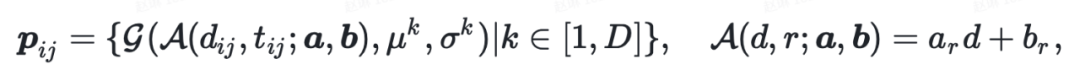
其中为通道数,为原子对,为原子对之间的欧氏距离,为边类型,为高斯核函数,其参数为。注意这里边类型不是化学键类型,它只与原子对的原子类型有关。是一个仿射变换,参数为和,它在原子对的欧氏距离和其对应的边类型之间建立联系。
1.1.2 原子对表征
通常 Transformer 只维护 Token(原子)级别的表征,在微调时的下游任务中也会调用它。然而由于分子的空间位置信息是在原子对级别上编码的,Uni-Mol 模型中也维护原子对表征,以便更好地学习分子的 3D 表征。具体实现中,原子对表征的初始化是上面提到的空间位置编码。之后为了更新原子对表征,深势科技团队通过自注意力机制中多头的 Query-Key 的乘积,进行原子到原子对的通信。形式上,原子对的更新可以表达成如下公式:
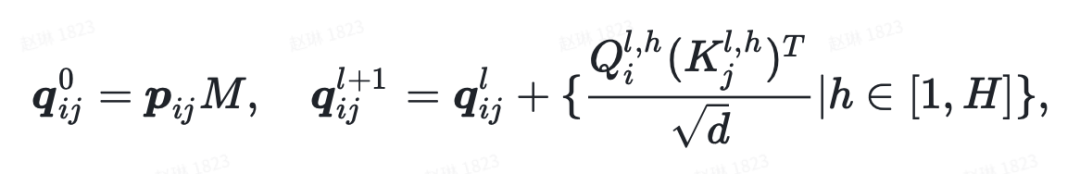
其中 是原子对表征, 是 attention heads 的数目, 是隐藏层维度, 是投影矩阵。
此外,为了利用原子表征中的三维信息,作者还引入了原子对到原子的通信,将原子对表征作为自注意力机制的 bias 项,可表示成如下公式:

1.1.3 具有SE(3)等变性的coordinate head
通过 3D 空间位置编码和原子对表征,Uni-Mol 可以学习到一个不错的分子 3D 表征。但它仍然缺乏直接输出坐标的能力,而这在 3D 空间任务中是必不可少的。为此,模型中加入了一个简单的 SE(3)-等变 head。按照 EGNN 的思路,SE(3)- 等变 head 的设计可以表示为:

其中为分子中的原子数目,是模型层数,是输入坐标,是投影矩阵。
1.2 预训练任务设计
1.2.1 预训练数据集
为了利用大规模无标签数据进行预训练,深势团队自己构造了两个大规模的有机小分子和蛋白口袋 3D 结构数据集。通过统一的预训练模型框架,结合有效的预训练任务策略,在大规模分布式集群上进行了预训练模型的训练。
分子预训练数据集是基于多个可购买分子数据集构造。经过归一化和去重,数据集包含大约 1900 万个分子,共 2.1 亿的 3D 分子构象。通过使用 rdkit 结合分子力场优化,高效生成分子构象。每个分子随机生成 10 个构象。由于某些分子 rdkit 生成 3D 构象失败,因此还额外对每一个分子生成了一个 2D 构象(基于分子图)帮助预训练。
蛋白质口袋预训练数据集来自于蛋白质数据库(RCSB PDB [http://www.rcsb.org])。库中有 180K 的结晶真实蛋白 3D 结构。为了构造合理的用于预训练的候选口袋数据,深势团队首先通过补全蛋白侧链和极性氢来进行蛋白准备,然后使用口袋检测工具 Fpocket 检测蛋白质上的潜在药效性口袋,同时保留了其中的水分子,通过上述方法,深势团队构造一个由 320 万个候选蛋白口袋组成的 3D 构象数据集。
1.2.2 自监督策略
与 BERT 类似,Uni-Mol 中也使用了对原子掩码的预测任务。对于每个分子/口袋,通过添加了一个特殊的原子 [CLS],其坐标是所有原子的中心,用 [CLS] 的表征代表整个分子/口袋的表征。然而,由于 3D 空间位置编码是有化学键信息泄露的,模型很容易依据相互间的距离推测出被掩盖的原子类型。因此单独对于原子掩码进行预测并不能帮助模型学习有用的信息。
为了解决这个问题同时又能从 3D 信息中学习,Uni-Mol 设计了一个基于 3D 坐标的去噪任务。具体实现中,对于被掩盖的 15% 的原子,给坐标同时加入 [-1Å , 1Å] 的均匀分布噪声,之后模型根据被污染的坐标计算出来空间位置编码。这样一来,对于原子掩码的预测任务就不再可有可无。此外,这里还加入了两个额外的任务单元来直接对于原子坐标进行预测:
1)还原被掩盖的原子间欧氏距离 基于原子对表征,预测被掩盖的原子对的欧氏距离。
2)直接预测被掩盖的原子坐标 通过设计合理的结构,从而保证模型更新对于平移、旋转具有等变性(SE(3) 等变性),去直接预测被掩盖的原子的正确坐标。
两个预训练模型都使用了上述的自监督任务帮助训练,由于蛋白口袋与许多药物设计任务直接相关,作者认为在候选蛋白质口袋数据上的预训练可以提高与蛋白质-配体结构及相互作用有关的任务的表现。图2是整个预训练框架的说明。
02 实验
2.1 分子性质预测
首先在备受 AI 从业者关注的分子性质预测任务上进行了实验。实验使用的 15 个数据集均来自于 MoleculeNet,划分方式上对齐了之前的工作,采用了骨架划分。从结果上来看,Uni-Mol 在 14/15 个数据集上取得 SOTA,尤其是在 3D 结构强相关的回归任务上,例如水化自由能(ESOL,FreeSolv),亲脂性( Lipo),物化性质(QM 系列)上面相对于之前的 SOTA 平均有 21% 的效果提升!


2.2 分子构象生成
分子构象生成是指使用计算机程序预测和模拟分子的空间构造和排列方式。它基于分子力学和量子化学原理,通过对分子的电子结构、力学和热学性质的计算,以预测一定的分子构象。
这些预测有助于研究分子之间的相互作用,从而帮助合成新的物质,设计新的药物以及理解分子之间的化学反应。分子构象生成也可以应用于研究蛋白质结构和蛋白质与药物的相互作用。
区别于以往的分子构象生成 baseline,Uni-Mol 是对 RDKit 生成的构象进行优化,在评价 AI 模型生成构象多样性的指标 Coverage 和精度指标 Matching 上,Uni-Mol 基本上全面超越现有的 baseline。
同时深势科技团队也提出对于该领域,目前使用的公开数据集主要关注低能的真空、水相模拟构象,而缺少真实的和蛋白结合的药效构象,因此分子构象生成的数据标准也是未来一个很重要的研究方向。

2.3 口袋性质预测
可药性,即候选蛋白质口袋与特定分子配体产生稳定结合的能力,是候选蛋白质口袋最关键的性质之一。由于有标签的数据很有限,这项任务非常具有挑战性。例如常用的 NRDLD 数据集,只包含 113 条数据。因此,除了 NRDLD 之外,作者还构建了一个回归数据集,用于模型性能测试。在表 4 中可以看到,Uni-Mol 表现卓越。

2.4 蛋白质-配体结合位点预测

图3. 蛋白质-配体结合位点预测模型框架, ▲ 编码器使用两路预训练Uni-Mol分表表征分子和口袋,解码器使用同样结构的随机初始化的Uni-Mol
蛋白质-配体结合的预测是药物设计中最重要的任务之一。Uni-Mol 结合了分子和口袋预训练模型来学习基于距离矩阵的评分函数,之后对复杂的构象进行采样和优化。在基准数据集上,作者使用 CASF-2016 作为测试集,使用 PDBbind General set 作为训练集,并且和测试集进行了去重,确保结果的可泛化性。
在 docking power 和 binding pose 两项评估结合最关键的指标上,Uni-Mol 均表现非常出色,在打分函数 docking power 测评上超越了一系列主流的 docking 工具以及 AI based 打分函数模型,更令行业兴奋的是,在最为直接的 binding pose 预测能力上面,对于 CASF-2016 基准数据集(RMSD<2.0 一般认为是可接受的 pose 预测结果)预测的准确结合构象的比例超过目前主流的 docking 工具约 35%,这无疑是巨大的飞跃。

03 总结
深势科技的研究员们希望通过建立统一的分子预训练框架,能够方便药物研发相关的从业人员高效精准的对于关注的具体下游任务能做到统一的建模,此外对于蛋白分子结合预测这一核心问题的探索,也预期着 Uni-Mol 能发挥巨大的潜力。作者同时也提到多个潜在的研究方向:
1. 更好的交互机制,如何将两个预训练模型放在一起进行微调。在当前版本的 Uni-Mol 中,预训练口袋模型和预训练分子模型之间的交互很基础,这块认为有较大的改进空间;
2. 更大的 Uni-Mol 模型。增大预训练模型往往能带来可观的提高,因此,用更多的数据来训练一个更大的 Uni-Mol,也是很值得探索的;
3. 更多高质量的 benchmark。尽管在 AI 模型在药物设计领域已经有很多应用,但高质量的公开数据集一直比较少,许多公开数据集并不能满足现实世界的需求。相信高质量的 benchmark 将成为整个领域的灯塔,并大大加速药物设计的发展。
Automatic Screen-out of Ir(III) Complex Emitters by Combined Machine Learning and Computational Analysis
结合机器学习和计算分析,自动筛选出Ir(III)复合发射体
Uni-Mol 性能优越、模型泛化能力强,在小分子性质预测、蛋白靶点预测和蛋白-配体复合物构象预测等任务上都超越之前方法。同时,我们成功把Uni-Mol应用在了例如材料设计等更多领域,并取得了优异成果。

Uni-Mol 在各类任务上都表现优异,超越之前的最好方法。图中内部灰色区域为之前的最好方法的效果,外部多种颜色区域描述的是是Uni-Mol在多种任务上超出之前最好方法的百分比。
二、Uni-Mol Universe: 更多的应用场景
1. Uni-Mol for QSAR
QSAR是定量构效关系(Quantitative Structure-Activity Relationship)的缩写,是一种基于化合物结构预测化合物的生物活性和生化性质的计算方法。该方法通过将分子结构与其物理、化学性质及生物活性相关联,然后构建一个模型,以期该模型可以预测新的化合物的生物活性。QSAR在药物设计、环境毒理学和农药研究等领域中得到广泛应用。
Uni-Mol based Auto-QSAR (Uni-QSAR) 是一套基于Uni-Mol模型开发的自动化分子属性预测工具,可供专业领域相关人员使用。我们对目前主流的QSAR工具在TDC ADMET Group Benchmark上的测评结果进行了比较。TDC(Therapeutics Data Commons)是哈佛医学院主导开发的一个基准平台,其中ADMET包含了药物小分子的吸收、分布、代谢、排泄和毒性五个方面的指标。这些因素对药物的疗效和安全性有着至关重要的影响。在药物研发过程中,需要对药物的ADMET特性进行评估和优化,以提高药物的成功率,减少不良反应的发生。Uni-QSAR在这些任务上表现出了非常优异的效果。通过结合Uni-Mol和高效的自动化工作流,用户不需要关注模型细节,无需调参,即可自动化地进行特征构造和筛选。同时,Uni-QSAR也考虑到了样本不平衡性和预测任务类型的多样化(分类、回归、多任务学习、缺失值训练等等),用户只需要关注自己的任务本身。
Uni-QSAR的内测版本已经成功地应用于国际知名的快速消费品牌Top3之一,表现出了优秀的预测能力。在多个数据库中,预测的准确性都有明显提升,超过了其他方案。该项目已经完成了首轮交付,并且正在探索未来的合作空间。同时,Uni-QSAR也即将上线Hermite®药物计算设计平台,敬请期待。

2. Uni-Mol for Materials
Uni-Mol在材料领域也积累了不少的应用和案例,以下我们会选取MOF和OLED两个经典案例分别阐释Uni-Mol的通用性和预测能力的扩展。
MOF材料是一种由金属离子或者簇合物和有机配体组成的多孔晶体材料,对MOF材料的气体吸附研究具有重要的理论和实际意义,例如,可以用于环境污染控制、能源储存和转换、化学催化等领域。
基于Uni-Mol,我们设计训练了一个跨体系的模型Uni-MOF,可以对不同的气体(甚至未知的气体)、在不同的环境下面(温度、压强等)进行预测,其结果也大幅超越了之前单体系模型。这种建模思路也非常契合目前大火的ChatGPT,可以认为我们是在MOF吸附领域实现了大一统模型,具体细节可以关注我们即将发布的论文。

我们也把Uni-Mol成功拓展到OLED Ir(III) 体系的大规模虚拟筛选上面,用于搜索性能更好的OLED发光材料。
OLED Ir(III)体系是一种基于有机发光二极管(OLED)技术的发光材料体系,其中使用了含铱(Ir)的荧光材料。这种体系具有高效、低功率消耗、高亮度和高稳定性等优点,因此在电子显示领域有广泛的应用。其中,Ir(III)配合物材料具有较高的荧光效率和发光寿命,可以用于制备高效的红、绿、蓝光发射器件。OLED Ir(III)体系在智能手机、平板电脑、电视、汽车仪表盘等领域都有着广泛的应用前景。
通过利用Uni-Mol强大的预测能力,我们可以极大地降低额外的计算成本,同时高通量的筛选迭代也能够进一步提高模型的预测效果(如下图左图所示)。这种大模型训练和QM小规模计算相互迭代的思路也将成为材料研发的一种新型范式。从下图右图所示的结果可以看出,Uni-Mol 也满足了OLED材料的筛选基本要求,例如需要光色尽可能纯和plqy尽可能大。
更多细节,请参阅我们在 ChemRxiv 上的预印本文章:Automatic Screen-out of Ir(III) Complex Emitters by Combined Machine Learning and Computational Analysis | Materials Chemistry | ChemRxiv | Cambridge Open Engage

除了MOF和OLED,Uni-Mol还可应用于更多的材料设计任务。由于篇幅所限,无法一一描述,期待不同背景的研究者与我们一起探索Uni-Mol的潜力。
三、Uni-Mol讲解教程及在线Notebook 体验
关于Uni-Mol详细的原理讲解,可以关注青年科学论坛上的报告(报告详见:Uni-Mol 分子3D表示学习框架和预训练模型 | 郑行(深势科技)| 青年科学半月谈_哔哩哔哩_bilibili),报告中使用深势科技推出的科学计算平台上的Bohrium Notebook展示了如何将Uni-Mol快速地应用在分子性质预测的任务上。
在Bohrium Notebook 上,我们准备了一系列基于Uni-Mol的封装好的软件库,与开源版本不同的是,这些小工具和软件包更加适配于应用层,环境和软件包都是内置安装好的,同时接口也进行了二次开发,可扩展性更高。用户只需要关注其具体的数据和应用。同时我们也持续收集用户的反馈,进行开发迭代。大家可以点击下面的链接直接进行体验测试:
3.1 分子属性预测案例
Bohrium,Bohrium Notebook可以自动地加载运行环境,通过几行代码即可对于自己的数据任务进行训练、预测,生成自己的属性预测工作流。
3.2 Uni-Mol Docking案例
Bohrium,Bohrium Notebook目前仅展示了对于CASF-2016的docking结果,大家可以自由选取靶点和对应的配体分子,然后进行docking,后续会开放更多的功能。