autoReg | 自动线性和逻辑回归和生存分析
autoReg可以制作描述性统计汇总表,在各种回归模型中自动选择解释变量。支持线性模型、广义线性模型和环比例风险模型。生成可发表的回归分析结果汇总表和图表。
#install.packages("devtools")
devtools::install_github("cardiomoon/autoReg")
library(autoReg)基线特征描述统计
## 基线特征描述统计
library(moonBook) # 示例数据
data(acs)
library(dplyr)
ft=gaze(sex~.,data=acs) %>% myft()
ft
# library(rrtable)
# table2pptx(ft) #Exported table as Report.pptx
# table2docx(ft) #Exported table as Report.docx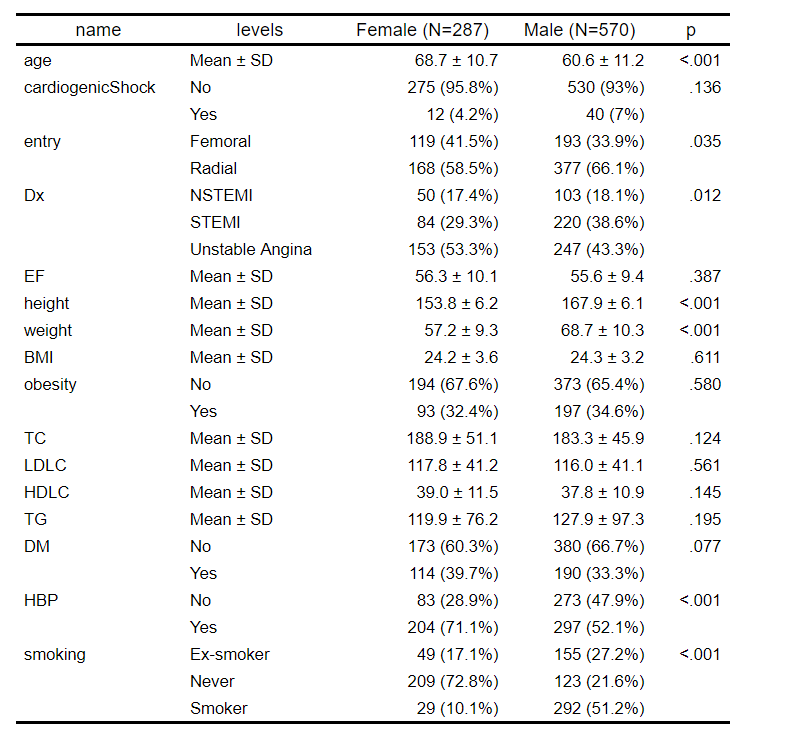
用两个或多个分组变量总结基线特征
gaze(sex+Dx~.,data=acs) %>% myft()
gaze(sex+DM+HBP~age,data=acs) %>% myft()自动选择解释变量
可以做一个表格总结回归分析的结果。例如,对结肠癌数据进行逻辑回归分析。
library(survival) # For use of data colon
data(cancer)
fit=glm(status~rx+sex+age+obstruct+perfor+nodes,data=colon,family="binomial")
summary(fit)
autoReg(fit) %>% myft()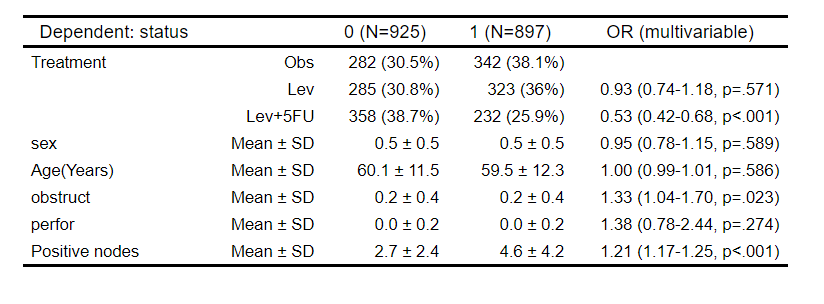 如果想要一个有更多解释的表,你可以用数字变量来做分类变量。
如果想要一个有更多解释的表,你可以用数字变量来做分类变量。
colon$status.factor=factor(colon$status,labels=c("Alive","Died"))
colon$obstruct.factor=factor(colon$obstruct,labels=c("No","Yes"))
colon$perfor.factor=factor(colon$perfor,labels=c("No","Yes"))
colon$sex.factor=factor(colon$sex,labels=c("Female","Male"))
fit=glm(status.factor~rx+sex.factor+age+obstruct.factor+perfor.factor+nodes,data=colon,family="binomial")
result=autoReg(fit)
result %>% myft()
## setLabel()函数给变量名添加标签
colon$status.factor=setLabel(colon$status.factor,"Mortality")
colon$rx=setLabel(colon$rx,"Treatment")
colon$age=setLabel(colon$age,"Age(Years)")
colon$sex.factor=setLabel(colon$sex.factor,"Sex")
colon$obstruct.factor=setLabel(colon$obstruct.factor,"Obstruction")
colon$perfor.factor=setLabel(colon$perfor.factor,"Perforation")
colon$nodes=setLabel(colon$nodes,"Positive nodes")
fit=glm(status.factor~rx+sex.factor+age+obstruct.factor+perfor.factor+nodes,data=colon,family="binomial")
result=autoReg(fit)
result %>% myft()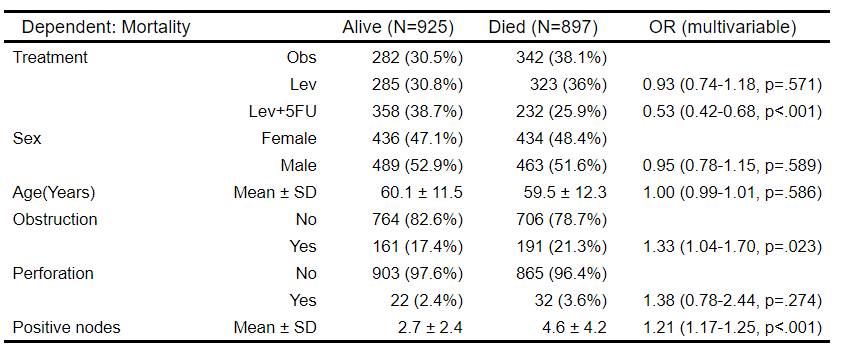
# 如果不想在表中显示参考值,可以缩短表。
shorten(result) %>% myft()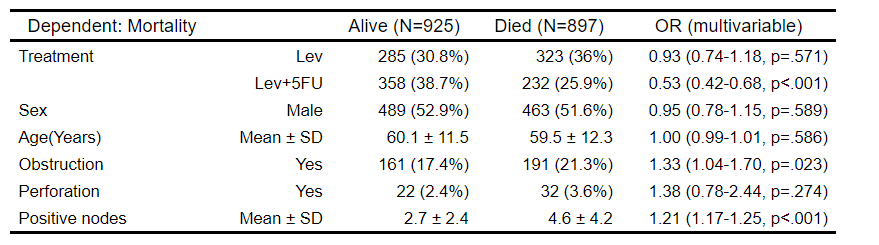
添加单变量模型表和自动选择解释变量
可以将单变量分析的结果添加到表中。此时,autoReg()函数自动选择阈值(默认值0.2)以下的解释变量,并执行多变量分析。在本表中,解释变量的p值为性别。因子和年龄均高于缺省阈值(0.2),在多元模型中被排除。
autoReg(fit, uni=TRUE) %>% myft()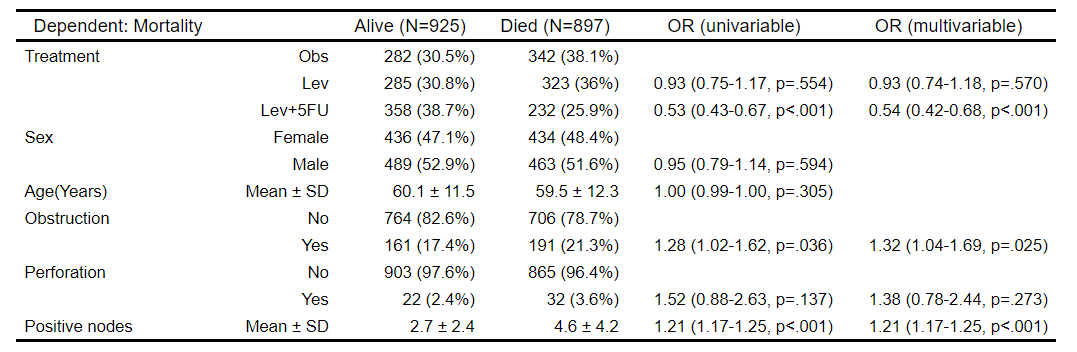
如果想在多元模型中包含所有解释变量,只需将阈值设置为1。
autoReg(fit, uni=TRUE,threshold=1) %>% myft()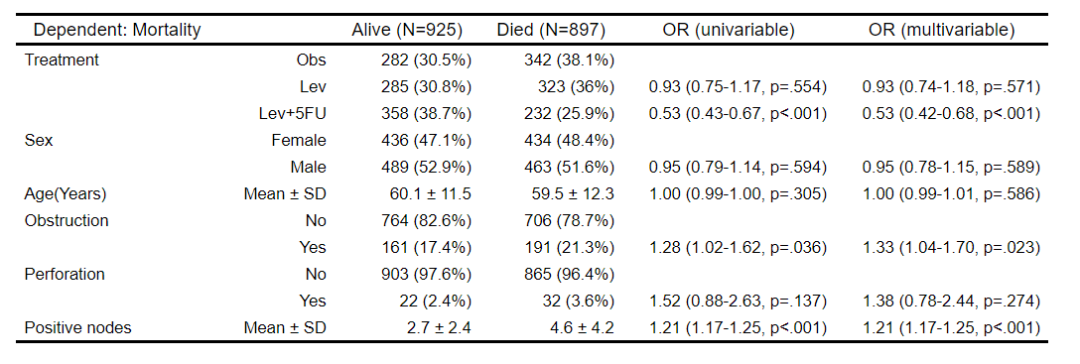
可以通过逐步的向后消除来选择变量并做出最终的模型。
autoReg(fit, uni=TRUE,threshold=1, final=TRUE) %>% myft()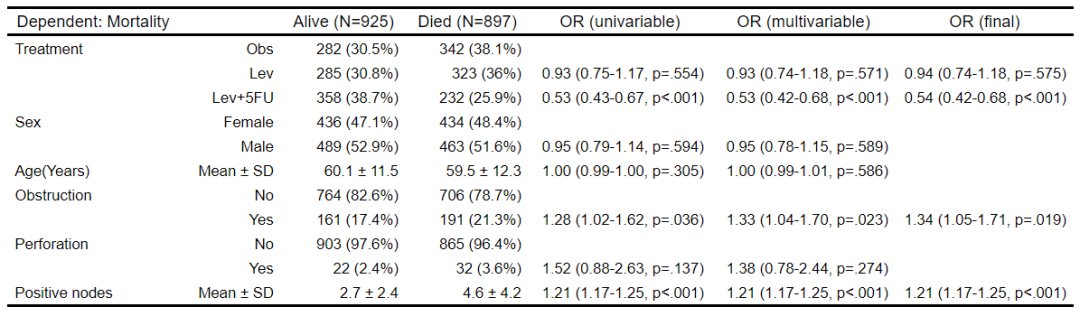
在图表中总结回归模型的结果
x=modelPlot(fit)
x
# plot2pptx(print(x)) ##Exported plot as Report.pptx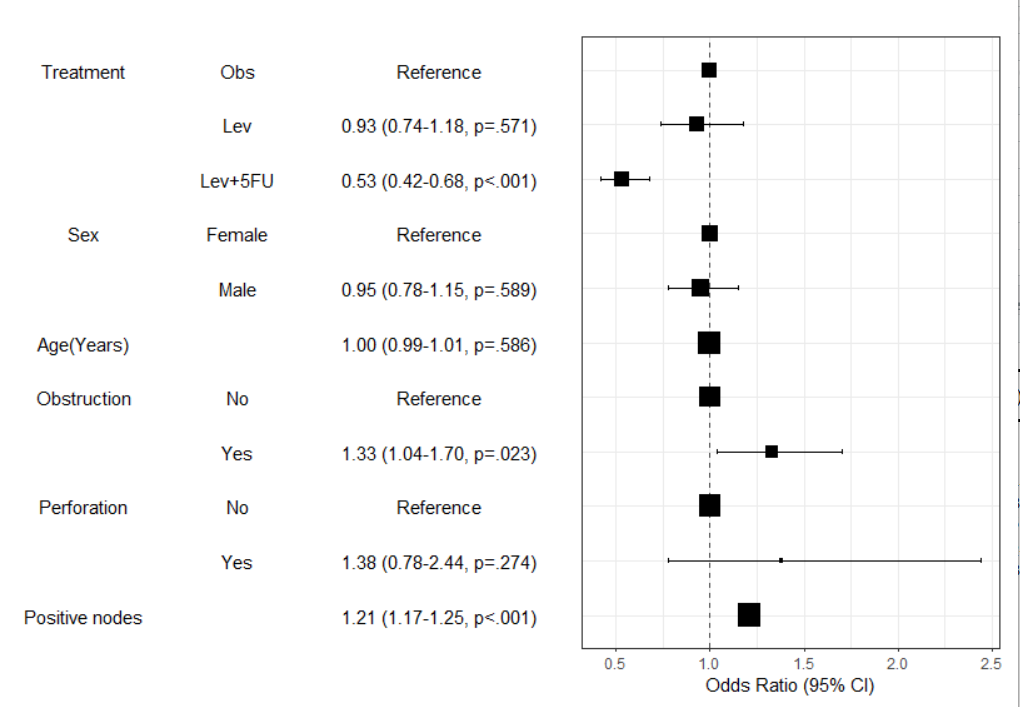
modelPlot(fit,uni=TRUE,threshold=1,show.ref=FALSE)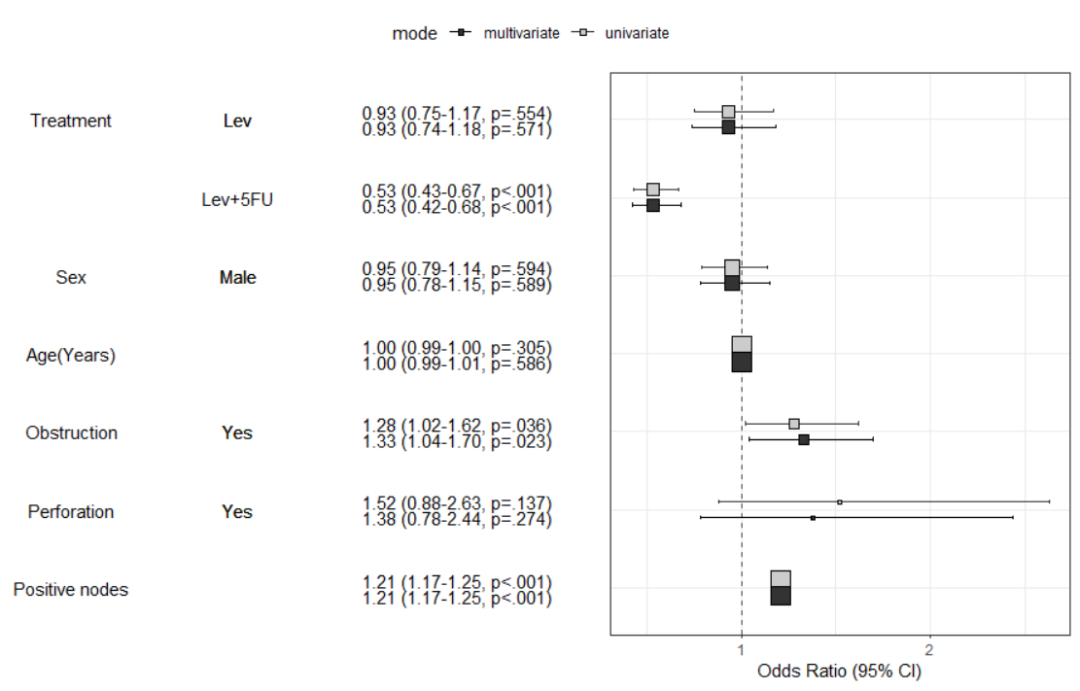
参考
GitHub - cardiomoon/autoReg: R package autoReg
木舟笔记2022年度VIP企划
关于木舟笔记2022年度VIP会员企划
权益:
2022年度木舟笔记所有推文示例数据及代码(含大部分2021年)。
木舟笔记科研交流群。
半价购买
跟着Cell学作图系列合集(免费教程+代码领取)|跟着Cell学作图系列合集。
收费:
99¥/人。可添加微信:mzbj0002 转账,或直接在文末打赏。

往期内容
