在前文中,我们已经详细介绍了决策树算法中的ID3/C4.5/CART算法的原理,以及决策树的剪枝问题。
ID3算法戳我
C4.5算法戳我
CART算法戳我
决策树剪枝问题戳我
本文将详细介绍如何用R语言实现决策树算法。
| 算法 | 区分要点 | R包 |
|---|---|---|
| ID3 | 使用信息增益 | rpart包中rpart函数 |
| C4.5 | 使用信息增益 | RWeka包中J48() |
| CART | 使用gini | rpart包中rpart函数 |
| C5.0 | C4.5的改进,比较适合于大规模数据 | c50包 |
数据简介
本文数据选择了红酒质量分类数据集,这是一个很经典的数据集,原数据集中“质量”这一变量取值有{3,4,5,6,7,8}。为了实现二分类问题,我们添加一个变量“等级”,并将“质量”为{3,4,5}的观测划分在等级0中,“质量”为{6,7,8}的观测划分在等级1中。
因变量:等级
自变量:非挥发性酸性、挥发性酸性、柠檬酸、剩余糖分、氯化物、游离二氧化硫、二氧化硫总量、浓度、pH、硫酸盐、酒精
library(openxlsx)
wine = read.xlsx(".../winequality-red.xlsx") #读入数据
#将数据集分为训练集和测试集,比例为7:3
train_sub = sample(nrow(wine),7/10*nrow(wine))
train_data = wine[train_sub,]
test_data = wine[-train_sub,]
R语言实现
ID3算法
ID3算法和CART算法都是用rpart包实现的,rpart包常用参数如下:
| 参数 | 意义 |
|---|---|
| formula | ~ + +…+ 或者 ~ . 指明了因变量和自变量 |
| data | 使用的数据集 |
| na.action | 缺失值处理,默认na.rpart,表示删掉因变量 缺失的观测,但是保留自变量缺失的观测 |
| method | 决策树的类型,“exp”用于生存分析,“poisson”用于二分类变量,“class”用于分类变量(使用居多),”anova”对应回归树 |
| parms | 只用于分类树,parms=list(split,prior,loss),其中split的选项默认"gini"(对应CART),和"information"(对应ID3算法) |
| control | 控制决策树形状的参数 |
rpart包的参数详解
rpart.control的参数详解
library(rpart)
#决策树的生成
wine_decisiontree_ID3 <- rpart(等级 ~ 非挥发性酸性 + 挥发性酸性 + 柠檬酸 + 剩余糖分 + 氯化物
+ 游离二氧化硫+二氧化硫总量+ 浓度+pH+硫酸盐+酒精,
data = train_data,
method="class",
parms=list(split="information"))
###决策树的剪枝
#树的剪枝判断
printcp(wine_decisiontree_ID3)
###结果
CP nsplit rel error xerror xstd
1 0.315068 0 1.00000 1.00000 0.032608
2 0.037182 1 0.68493 0.70450 0.030580
3 0.015656 4 0.57339 0.65362 0.029955
4 0.013699 5 0.55773 0.65166 0.029929
5 0.010437 6 0.54403 0.65753 0.030006
6 0.010000 9 0.51272 0.64384 0.029825
#树的剪枝,选择使得xerror最小的cp值
wine_decisiontree_ID3_prune<-prune(wine_decisiontree_ID3,cp= 0.010000)
至此,我们已经将决策树生成好,之后我们将使用rpart.plot绘制决策树
rpart.plot主要的参数有:
| 参数 | 意义 |
|---|---|
| tree | 所绘制的模型 |
| type | 可选1,2,3,4,控制节点的形状 |
| fallen.leaves | 默认为T |
| branch | 控制树的外观,若branch=1则获得垂直的决策树 |
| cex | 图中符号的大小 |
library(rpart.plot)
rpart.plot(wine_decisiontree_ID3_prune,branch=1, fallen.leaves=T,cex=0.6)
ID3算法所绘树图:
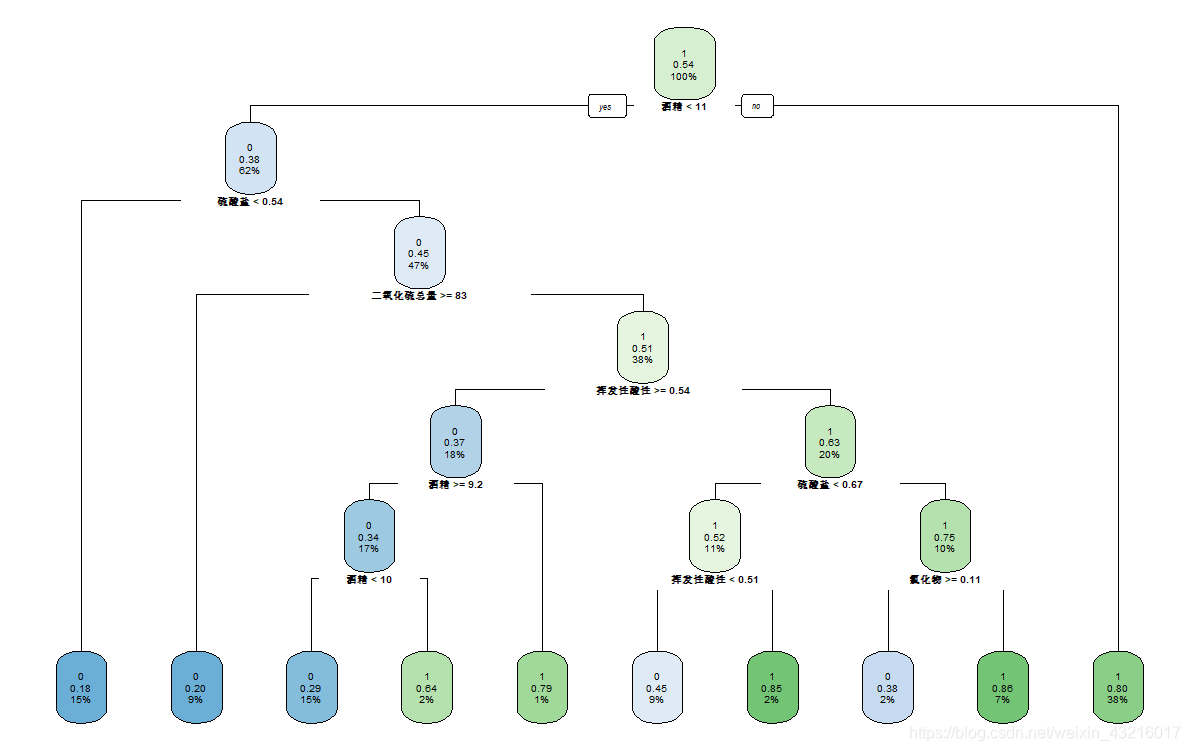
用所得的ID3模型在测试集上预测
#在测试集上预测
pre_decisiontree_ID3<-predict(wine_decisiontree_ID3_prune,newdata=test_data,type="class")
#将测试集计算所得概率与观测本身取值整合到一起
obs_p_decision_ID3 = data.frame(prob=pre_decisiontree_ID3,obs=test_data$等级)
#输出混淆矩阵
table(test_data$等级,pre_decisiontree_ID3,dnn=c("真实值","预测值"))
#绘制ROC曲线
library(pROC)
decisiontree_roc_ID3 <- roc(test_data$等级,as.numeric(pre_decisiontree_ID3))
plot(decisiontree_roc_ID3, print.auc=TRUE, auc.polygon=TRUE, grid=c(0.1, 0.2),grid.col=c("green", "red"), max.auc.polygon=TRUE,auc.polygon.col="skyblue", print.thres=TRUE,main='ID3算法ROC曲线')
ID3算法所得AUC值为0.746
CART算法
CART算法的实现也是使用rpart包
library(rpart)
library(rpart.plot)
#决策树的生成
wine_decisiontree_cart <- rpart(等级 ~ 非挥发性酸性 + 挥发性酸性 + 柠檬酸 + 剩余糖分 + 氯化物 + 游离二氧化硫 +
二氧化硫总量 + 浓度 + pH + 硫酸盐 + 酒精,
data = train_data,
method="class",
parms=list(split="gini"))
###决策树的剪枝
#树的剪枝判断
printcp(wine_decisiontree_cart)
CP nsplit rel error xerror xstd
1 0.350294 0 1.00000 1.00000 0.032608
2 0.022179 1 0.64971 0.69667 0.030490
3 0.019569 4 0.58317 0.64775 0.029877
4 0.011742 7 0.52446 0.64579 0.029851
5 0.010000 10 0.48924 0.63796 0.029745
#树的剪枝,选择使得xerror最小的cp值
wine_decisiontree_cart_prune<-prune(wine_decisiontree_cart,cp= 0.010000 )
#画出决策树
rpart.plot(wine_decisiontree_cart_prune,branch=1, fallen.leaves=T,cex=0.6)
CART算法所绘树图:
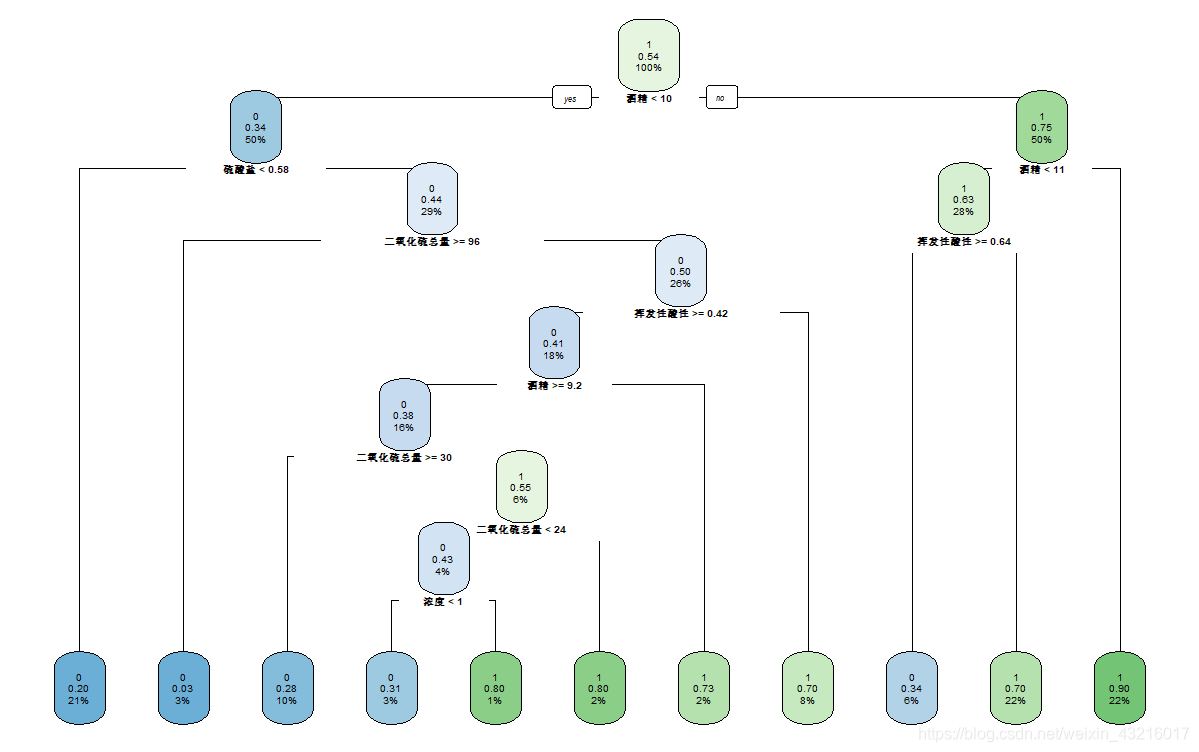
用所得的CART模型在测试集上预测
#在测试集上预测
pre_decisiontree_cart<-predict(wine_decisiontree_cart_prune,newdata=test_data,type="class")
#将测试集计算所得概率与观测本身取值整合到一起
obs_p_decision_cart = data.frame(prob=pre_decisiontree_cart,obs=test_data$等级)
#输出混淆矩阵
table(test_data$等级,pre_decisiontree_cart,dnn=c("真实值","预测值"))
#绘制ROC图像
library(pROC)
decisiontree_roc_cart <- roc(test_data$等级,as.numeric(pre_decisiontree_cart))
plot(decisiontree_roc_cart, print.auc=TRUE, auc.polygon=TRUE, grid=c(0.1, 0.2),grid.col=c("green", "red"), max.auc.polygon=TRUE,auc.polygon.col="skyblue", print.thres=TRUE,main='Cart算法ROC曲线')
CART算法所得AUC值为0.718
C4.5算法
C4.5算法使用RWeka包,RWeka包的安装需要依赖于rJava包。
在安装好rJava包后,使用如下代码即可安装RWeka包
install.packages("RWeka")
library(RWeka)
我们使用RWeka包中的J48()来调用C4.5算法。
J48(formula,data,subset,na.action,control=Weka_control(u=T,c=0.25,M=2,R=T,N=3,B=T),options=NULL)
| 参数 | 意义 |
|---|---|
| U | 默认位TRUE,表示不剪枝 |
| C | 对剪枝过程设置置信区间 |
| M | 表示叶节点的最小样本量,默认为2 |
| R | 按错误率降低剪枝法剪枝 |
| N | 当R=TRUE时,交叉验证的折叠次数,默认为3 |
| B | 表示是否建立二叉树,默认为TRUE |
#Sys.setenv(JAVA_HOME='C:\\Program Files\\Java\\jdk1.8.0_191\\jre') #看是否有必要更改环境变量
library(rJava)
library(RWeka)
library(pROC) #绘制ROC曲线
wine_decisiontree_C4_5 <- J48(factor(等级) ~ 非挥发性酸性
+ 挥发性酸性 + 柠檬酸 + 剩余糖分
+ 氯化物 + 游离二氧化硫+二氧化硫总量+浓度
+pH+硫酸盐+酒精, data = train_data,
control = Weka_control(U = FALSE , R= TRUE, M=2,B=FALSE))
#这样可以得到一个比较简陋的树图
print(wine_decisiontree_C4_5)
###library(partykit)
###plot(wine_decisiontree_C4_5) #可以得到一个相对好看的树图
#在测试集上预测
pre_decisiontree_C4_5<-predict(wine_decisiontree_C4_5,newdata=test_data)
#将测试集计算所得概率与观测本身取值整合到一起
obs_p_decision_c4_5 = data.frame(prob=pre_decisiontree_C4_5,obs=test_data$等级)
#输出混淆矩阵
table(test_data$等级,pre_decisiontree_C4_5,dnn=c("真实值","预测值"))
#绘制ROC图像
decisiontree_roc_C4_5 <- roc(test_data$等级,as.numeric(pre_decisiontree_C4_5))
plot(decisiontree_roc_C4_5, print.auc=TRUE, auc.polygon=TRUE, max.auc.polygon=TRUE,auc.polygon.col="skyblue", print.thres=TRUE,main='C4.5算法ROC图像')
C4.5算法所得AUC值为0.731
C5.0算法
C5.0算法是C4.5算法的改进,使用的是c50包
library(C50)
#c50算法
#c5.0(x,y,trials=1) x表示自变量,y表示因变量,
#c5.0是一个boosting算法,trails控制循环次数
wine_decisiontree_C50 <- C5.0(train_data[1:11], factor(train_data$等级))
用所得的C5.0模型在测试集上预测
#在测试集上预测
pre_decisiontree_C50 <- predict(wine_decisiontree_C50, test_data)
#将测试集计算所得概率与观测本身取值整合到一起
obs_p_decision_c50 = data.frame(prob=pre_decisiontree_C50,obs=test_data$等级)
#输出混淆矩阵
table(test_data$等级,pre_decisiontree_C50,dnn=c("真实值","预测值"))
#绘制ROC图像
decisiontree_roc_c50 <- roc(test_data$等级,as.numeric(pre_decisiontree_C50))
plot(decisiontree_roc_c50, print.auc=TRUE, auc.polygon=TRUE, grid=c(0.1, 0.2),grid.col=c("green", "red"), max.auc.polygon=TRUE,auc.polygon.col="skyblue", print.thres=TRUE,main='C5.0算法ROC曲线')
C5.0算法所得AUC值为0.735