据降维是机器学习领域经常使用到的数据处理方法,所以在本专栏在正式开始深度学习专题之前,先介绍几种常用的降维方法(PCA、KPCA、t-SNE、MDS等)和MATLAB实现。
关于PCA的原理讲解在知乎上已经有了很好的文章,所以在这里我不再详细解释了,想了解更多的同学们可以看这里:
在这里想做的,主要是从很多人没太提到的实际应用的角度出发,给大家举两个形象一些的案例,以更直观的视角介绍PCA降维什么时候用、好不好用、以及怎么用。
一、PCA的基本概念
主成分分析(Principal Component Analysis, PCA)在数据降维中的应用特别广泛[1]。
PCA通过一系列的方差最大化的投影,可以得到特征值从大到小排列的特征向量,再根据需要选取维度截取对应的矩阵即可。
下边我来举两个例子,说明一下PCA常见的应用场合和使用方法。
二、案例1:降维、聚类与分类
这里介绍一下鸢尾花数据集,鸢尾花在机器学习里是常客之一。数据集由具有150个实例组成,其特征数据包括四个:萼片长、萼片宽、花瓣长、花瓣宽。数据集中一共包括三种鸢尾花,分别叫做Setosa、Versicolor、Virginica,就像下图:

也就是说这组数据的维度是150*4,数据是有标签的。(有标签是指每个实例我们都知道它对应的类别)
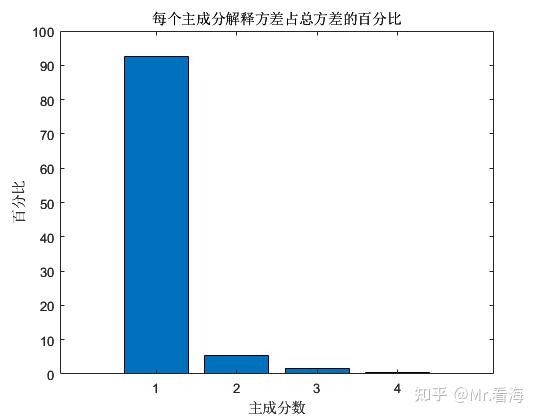
此时我们进行PCA降维,可以得到每个主成分解释方差占总方差的百分比,这个数值可以用以表示每个主成分中包含的信息量,从计算结果上来看,第1个主成分和第2个主成分的百分比之和已经超过97%,前三个主成分百分比之和更是超过了99%。
我们可以绘制一下数据降到二维和三维时,降维数据的分布情况:
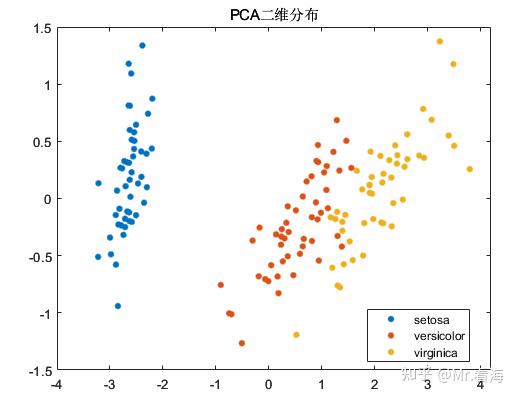
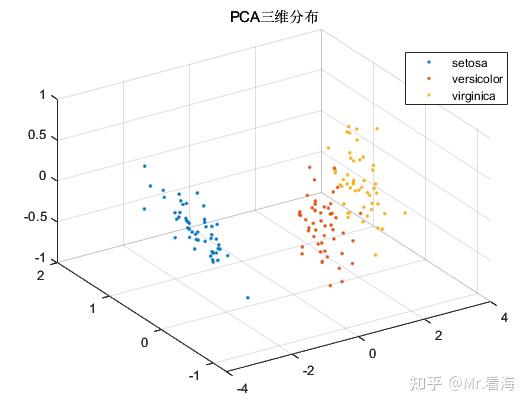
尽管PCA算法的初衷是降维而非聚类,不过由于PCA降维后的数据常常会用做机器学习的输入数据,在数据降维的同时查看降维后数据的分布情况,对于模式识别/分类任务的中间状态确定还是十分有益的,再直白些说,这些图片放在论文里丰富一下内容也是极好的。
在这种应用场景下,数据降维的最主要目的其实还是解决数据特征过于庞大的问题,这个例子中特征只有4个,所以还不太明显。很多时候我们面对的是几十上百乃至更多的特征维度,这些特征中包含着大量冗余信息,使得计算任务变得非常繁重,调参的难度和会大大增加。此时加入一步数据降维就是十分有必要的了。
三、案例2:寿命预测中的健康指标
在之前的文章中,曾经使用PCA做过寿命预测健康指标的表征:
Mr.看海:三岁看大,七岁看老——基于退化模型进行剩余有效寿命预测的案例讲解
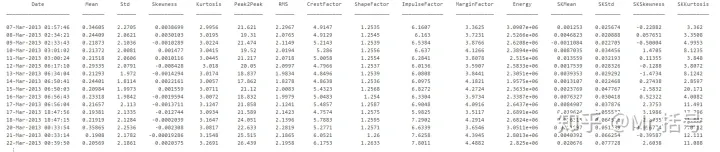
在该案例中,使用到的特征包括原始信号的:"Mean"; "Std"; "Skewness"; "Kurtosis"; "Peak2Peak","RMS"; "CrestFactor"; "ShapeFactor"; "ImpulseFactor"; "MarginFactor"; "Energy"。
以及谱峭度的:"Mean"; "Std"; "Skewness"; "Kurtosis"。
共计15中特征值,在50天的时间内,可以得到一张大小为50*15的特征表。表格中的部分数据如下:
但是做寿命预测与做分类不同,在寿命预测任务中,需要将上述特征融合成单一指标,作为表征当前设备健康状态的量化数值,也就是所谓的健康指标。此时就需要将15维特征降为1维,并用这1维数据代表退化特征。此时画出来的退化过程如下:
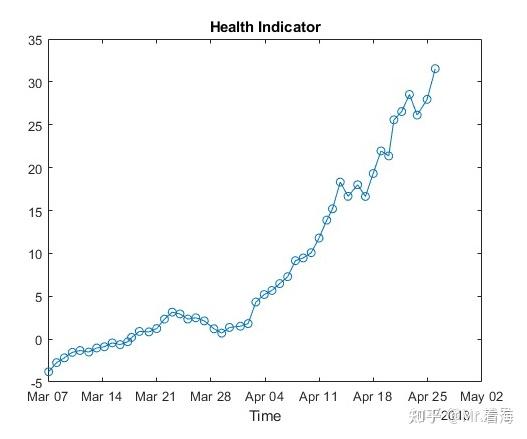
有了这张图,后续的寿命预测就可以以此为基准,将复杂的问题转换为时间序列预测了。
四、MATLAB的PCA降维快速实现
PCA算法在MATLAB中有官方函数,名字就叫做pca,熟悉编程的同学可以直接调用。
对于不熟悉MATLAB编程,或者希望更简洁的方法实现PCA降维,并同时绘制出相关图片的同学,则可以考虑使用本专栏封装的函数,它可以实现:
1.输入数据的行列方向纠正。是的,MATLAB的pca函数对特征矩阵的输入方向是有要求的,如果搞不清,程序可以帮你自动纠正。
options.autoDir='on';%是否进行自动纠错,'on'为是,否则为否。开启自动纠错后会智能调整数据的行列方向。2.指定输出的维度。也就是降维之后的维度,当然这个数不能大于输入数据的特征维度。
options.NumDimensions=3;%降维后的数据维度3.数据归一化。你可以选择在PCA之前,对特征数据进行归一化,这也只需要设置一个参数。
options.Standardize=false;%输入数据是否进行标准化,false (默认) | true 4.绘制特征分布图和成分百分比图。在降维维度为2或者3时,可以绘制特征分布图,当然你也可以选择设置不画图,图个清静。
figflag='on';%是否画图,'on'为画图,'off'为不画,只有NumDimensions为2或者3时起作用,3以上无法画图设置好这些配置参数后,只需要调用下边这行代码:
[pcaVal,explained,coeff]=khPCA(data,options,species,figflag);%pcaVal为降维后的数据矩阵就可以绘制出这样两张图:

如果要绘制二维图,把options.NumDimensions设置成2就好了。
不过上述是知道标签值species的情况,如果不知道标签值,设置species=[]就行了,此时画出来的分布图是单一颜色的。
上述代码秉承了本专栏一向的易用属性,功能全部集中在khPCA函数里了,这个函数更详细的介绍如下:
function [pcaVal,explained,coeff,PS] = khPCA(data,options,species,figflag)
% 对数据进行PCA降维并且画图
% 输入:
% data:拟进行降维的数据,data维度为m*n,其中m为特征值种类数,n为每个特征值数据长度
% options:一些与pca降维有关的设置,使用结构体方式赋值,比如 options.autoDir = 'on',具体包括:
% -autoDir:是否进行自动纠错,'on'为是,否则为否。开启自动纠错后会智能调整数据的行列方向。
% -NumDimensions:降维后的数据维度,默认为2,注意NumDimensions不能大于data原本维度
% -Standardize:输入数据是否进行标准化,false (默认) | true
%
% species:分组变量,可以是数组、数值向量、字符数组、字符串数组等,但是需要注意此变量维度需要与Fea的组数一致。该变量可以不赋值,调用时对应位置写为[]即可
% 例如species可以是[1,1,1,2,2,2,3,3,3]这样的数组,代表了Fea前3行数据为第1组,4-6行数据为第2组,7-9行数据为第三组。
% 关于此species变量更多信息,可以查看下述链接中的"Grouping variable":
% https://ww2.mathworks.cn/help/stats/gscatter.html?s_tid=doc_ta#d124e492252
%
% figflag:是否画图,'on'为画图,'off'为不画,只有NumDimensions为2或者3时起作用,3以上无法画图
% 输出:
% pcaVal:主成分分数,即经过pca分析计算得到的主元,每一列是一个主元
% explained:每个主成分解释方差占总方差的百分比,以列向量形式返回。
% coeff:主成分系数,由于data*coeff=score,所以当有一组新的数据data2想要以同样的主元坐标系进行降维时,可以使用data2*coeff得到,然后截取相应的列
% 但如果options.Standardize设置为true,则需要采用下述指令实施同步归一化及降维: mapminmax('apply',data2,PS)*coeff
% PS:数据归一化的相关参数,只有在options.Standardize设置为true时会返回该参数需要上边这个函数文件以及测试代码的同学,可以在下边链接获取:
参考
^跟着迪哥学 Python数据分析与机器学习实战