PCA,是一种降维算法,在我们处理高维数据的时候,他能够降低后序计算的复杂度,在“预处理”阶段先对原始数据进行降维。PCA就是通过将高维的数据通过线性变换投影到低维空间。
降维当然意味着信息的丢失,不过鉴于实际数据本身常常存在的相关性,我们可以想办法在降维的同时将信息的损失尽量降低。
以下就是PCA算法的基本步骤,我们假设有m条n维数据:
- 将原始数据按列组成n行m列矩阵X
- 将X的每一行(代表一个属性字段)进行零均值化,即减去这一行的均值
- 求出协方差矩阵
- 求出协方差矩阵的特征值及对应的特征向量
- 将特征向量按对应特征值大小从上到下按行排列成矩阵,取前k行组成矩阵P
- 即为降维到k维后的数据
以下是PCA实现的Python源码:
#coding=utf-8
from numpy import *
'''通过方差的百分比来计算将数据降到多少维是比较合适的,
函数传入的参数是特征值和百分比percentage,返回需要降到的维度数num'''
def eigValPct(eigVals,percentage):
sortArray=sort(eigVals) #使用numpy中的sort()对特征值按照从小到大排序
sortArray=sortArray[-1::-1] #特征值从大到小排序
arraySum=sum(sortArray) #数据全部的方差arraySum
tempSum=0
num=0
for i in sortArray:
tempSum+=i
num+=1
if tempsum>=arraySum*percentage:
return num
'''pca函数有两个参数,其中dataMat是已经转换成矩阵matrix形式的数据集,列表示特征;
其中的percentage表示取前多少个特征需要达到的方差占比,默认为0.9'''
def pca(dataMat,percentage=0.9):
meanVals=mean(dataMat,axis=0) #对每一列求平均值,因为协方差的计算中需要减去均值
meanRemoved=dataMat-meanVals
covMat=cov(meanRemoved,rowvar=0) #cov()计算方差
eigVals,eigVects=linalg.eig(mat(covMat)) #利用numpy中寻找特征值和特征向量的模块linalg中的eig()方法
k=eigValPct(eigVals,percentage) #要达到方差的百分比percentage,需要前k个向量
eigValInd=argsort(eigVals) #对特征值eigVals从小到大排序
eigValInd=eigValInd[:-(k+1):-1] #从排好序的特征值,从后往前取k个,这样就实现了特征值的从大到小排列
redEigVects=eigVects[:,eigValInd] #返回排序后特征值对应的特征向量redEigVects(主成分)
lowDDataMat=meanRemoved*redEigVects #将原始数据投影到主成分上得到新的低维数据lowDDataMat
reconMat=(lowDDataMat*redEigVects.T)+meanVals #得到重构数据reconMat
return lowDDataMat,reconMat对PCA降维后的数据最直接的应用是聚类,比如使用Kmeans。或者是使用在数据可视化这一个方向上,降维到二维或者三维数据。
以下为PCA的一个实际应用:
假设一组数据,其中包括n个feature(客户年龄、收入,每个月消费额度等等),每一个feature有一系列的观测点。而这n个feature中有一些存在线性相关,比如对于某些群体而言,收入和消费是线性相关的。此时我们进行多维度数据分析时只需要考虑其中一个参数就可以足够了,这样就能减少一个feature(维度),PCA方法就是用严谨的数据方法实现了这一过程。我们下面介绍了用sklearn中的PCA的简单实现和降维后的快速聚类工作。
以下为输入数据:
源码如下:
from sklearn.decomposition import PCA
from sklearn.cluster import KMeans
import pandas as pd
import numpy as np
#pca.txt是一个没有表头的多维数据,一共有7列,利用pandas读取
df = pd.read_table('d:/PCA.txt')
#将df转换成矩阵
dataMat = np.array(df)
#调用sklearn中的PCA,其中主成分有5列
pca_sk = PCA(n_components=5)
#利用PCA进行降维,数据存在newMat中
newMat = pca_sk.fit_transform(dataMat)
#利用KMeans进行聚类,分为3类
kmeans = KMeans(n_clusters=3,random_state=0).fit(newMat)
#labels为分类的标签
labels = kmeans.labels_
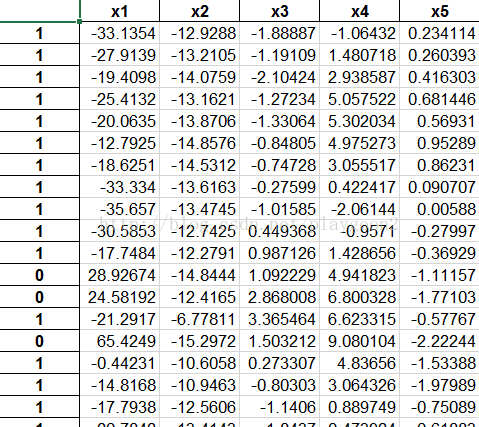
#把标签加入到矩阵中用DataFrame生成新的df,index为类别的编号,这里是0,1,2
dataDf = pd.DataFrame(newMat,index=labels,columns=['x1','x2','x3','x4','x5'])
#数据保存在excel文件中
dataDf.to_excel('d:/pca_cluster.xls')
print(pca_sk.explained_variance_ratio_) 输出为:
[ 0.85847673 0.09840701 0.0223092 0.01740787 0.00304186]结果为每一列所占特征方差的百分比,总和为0.9996说明降维保持了原数据的特征。
Excel的输出结果为: