近日,华中科技大学宁康教授团队针对野生和栽培甘草根的基因表达,次生代谢产物含量及根系微生物群落分布展开了深入研究,提出了野生和栽培甘草基因表达-微生物群落-代谢产物调控模式。相关研究成果以“Multi-omics profiling reveals comprehensive microbe-plant-metabolite regulation patterns for medicinal plant Glycyrrhiza uralensis Fisch”作为标题,6月在线发表于植物学知名期刊《Plant Biotechnology Journal》。
甘草是一种广泛使用的中药材,其药用部位为根及根茎,具有补脾益气、清热解毒、祛痰止咳等功效,其主要成分为甘草酸和甘草苷。由于野生资源的缺乏,栽培甘草逐渐成为解决甘草需求的重要途径。通过多组学技术研究野生及栽培甘草的遗传信息及其代谢调控网络,有助于揭示甘草重要次生代谢产物的积累机制。
该研究选用乌拉尔甘草(Glycyrrhiza uralensis Fisch)作为研究对象,重新定义了更为精准的乌拉尔甘草基因结构,鉴定的40,091个基因中包括数千个此前未见报道的基因,并完善了基因表达谱。利用代谢组学技术测定野生(WT),栽培一年(C1)和栽培三年(C3)乌拉尔甘草根中甘草酸和甘草苷的含量,发现野生甘草较栽培甘草积累了更多的甘草酸和甘草苷。结合转录组数据分析鉴定甘草酸和甘草苷合成途径的关键基因,进一步发现BAS、CYP72A154、CYP88D6等关键基因在野生甘草中的表达量显著高于栽培甘草(图1)。此外,通过甘草转录谱、代谢产物、根际微生物群落的网络联合分析,发现甘草酸和甘草苷合成关键基因的表达与微生物群落中细菌的多样性及丰度谱高度相关(图2)。进一步分析表明,根系微生物Lysobacter的丰度与甘草酸和甘草苷合成途径中的关键基因(如CYP72A154)的表达显著相关。最后,该研究提出了甘草生长过程的整体多组学调控模型,确认了根际微生物群落结构在甘草酸和甘草苷积累中的重要性。
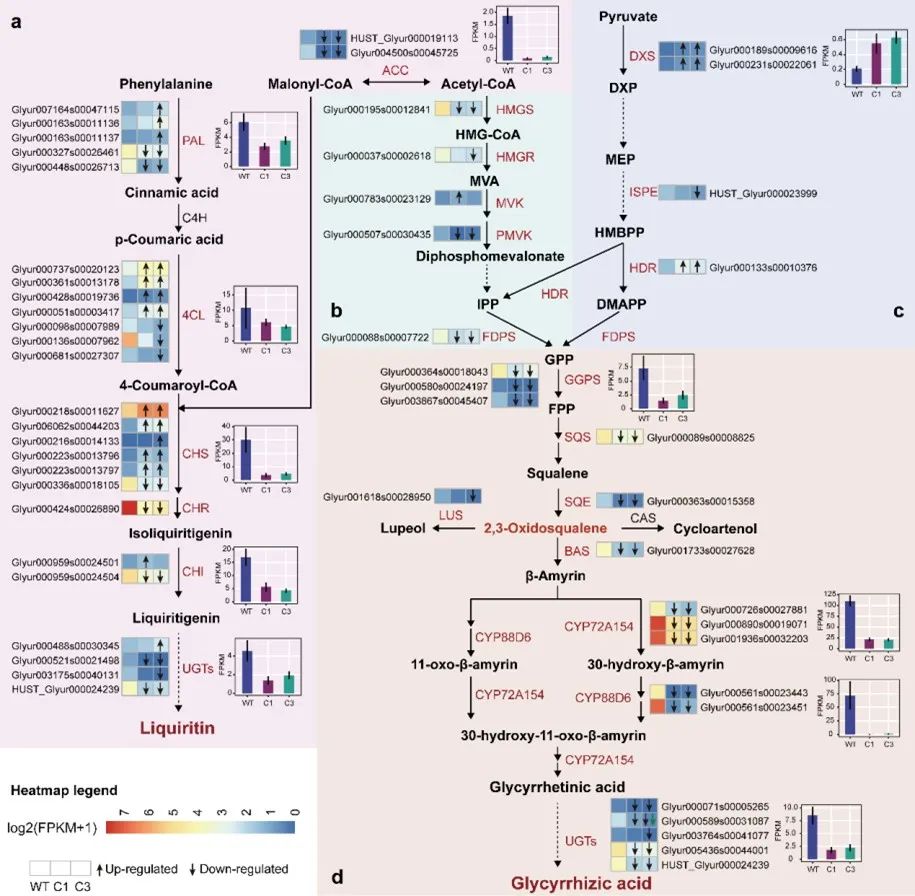
图1 野生(WT)及栽培(C1和C3)甘草中甘草酸和甘草苷合成途径的差异表达基因通路
该研究通过对野生和栽培甘草的转录组、代谢产物、根际微生物群落进行分析和比较,探究甘草酸和甘草苷在野生和栽培甘草根中的积累与基因表达的相关性,阐明了野生和栽培甘草主要成分含量差异的原因,同时揭示了根际微生物群落结构对甘草酸和甘草苷合成的潜在影响模式。该工作的研究结果,对于优化栽培甘草生长环境、强化甘草遗传改良、提升甘草品质具有重要意义。
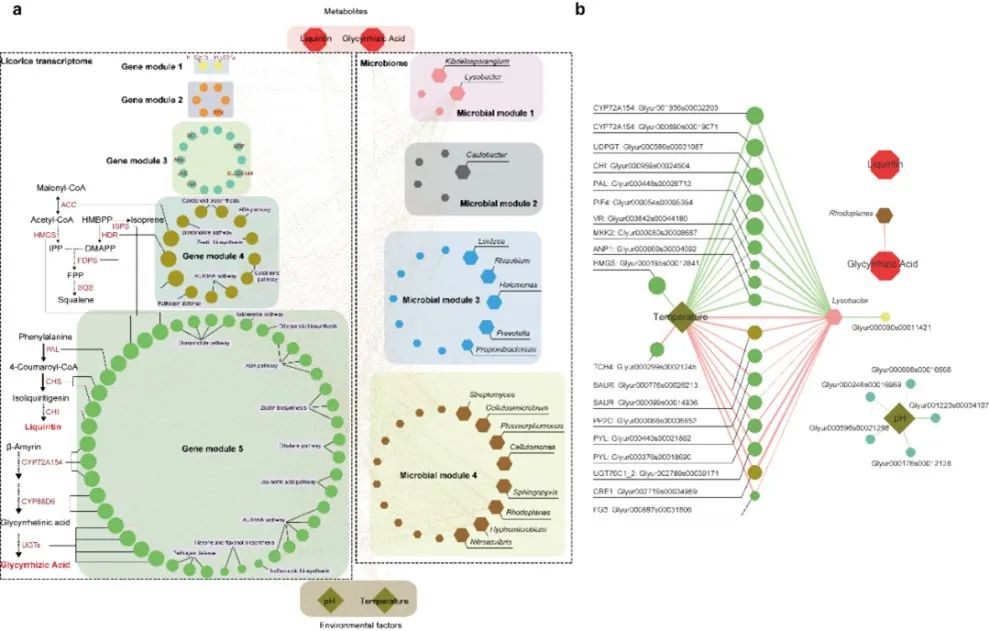
图2 甘草基因表达-微生物群落-代谢物调控网络
华中科技大学生命学院钟朝芳博士为论文的第一作者,华中科技大学宁康教授和白虹高级工程师为论文的共同通讯作者。该研究得到国家自然科学基金(81774008,81573702,32071465,31871334,31671374和62072283),科技部国家重点研发计划(2018YFC0910502)等的资助。特别感谢宁夏回族自治区药品检验所王英华老师在药材采集及鉴定方面给予的大力帮助和支持。
此项工作是宁康教授和白虹高级工程师共同带领的“药用植物组学大数据研究方向”又一重要研究成果。此系列工作还包括:2022年4月于国际权威期刊Environmental Microbiome发表了题为“Glycyrrhiza uralensis Fisch. Root-associated microbiota: the multifaceted hubs associated with environmental factors, growth status and accumulation of secondary metabolites”的论文。该文章提出了甘草根际微生物在调控甘草生长和重要代谢产物积累方面扮演着重要的“多面手”角色。此外, 2019年于国际权威期刊Frontiers in Pharmacology发表了题为“Network Pharmacology Databases for Traditional Chinese Medicine: Review and Assessment”的论文,为药用植物组学研究领域提供了大数据基础,为SCI高被引论文和热点论文。
宁康教授长期从事微生物组大数据和人工智能方面的研究,已在iMeta、PNAS、Gut、Genome Biology、Genome Medicine、Nucleic Acids Research等高水平学术期刊发表多篇学术论文。担任iMeta、Microbiology Spectrum、Genomics Proteomics Bioinformatics、Scientific Reports等国际期刊编委。担任中国生物信息学学会(筹)-基因组信息学分会副主任,中国生物工程学会-计算生物学与生物信息学专业委员会委员,中国计算机协会-生物信息学专业委员会委员等。欢迎对相关研究方向感兴趣的研究生和博士后加盟,团队网站:
http://www.microbioinformatics.org/。
猜你喜欢
iMeta简介 高引文章 高颜值绘图imageGP 网络分析iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文