原创: 林二狗 宇宙实验媛
宏基因组
宏基因组测序是将环境总DNA提取出来,随机打断成300/500bp的小片段,然后在片段两端加入通用引物进行PCR扩增测序,然后对测序数据进行质控,再将高质量序列拼接,根据数据库参考信息,对基因序列进行预测和功能注释,最终获得重要的宏基因组信息,如序列组成(GC含量、基因组大小等)、物种组成、功能组成和群落特征等。
相比于16S扩增子测序,宏基因组测序能够使物种鉴定深度达到“种”,而前者往往只能达到“属”的级别。另外,基于16S扩增子测序的基因预测结果(预测方法请参见往期内容“微生物组学研究手段概览——扩增子测序”)是依据数据库中的参考序列得到的,宏基因组测序则提供了菌群实际的基因信息。所以,如果不考虑经费的限制,宏基因组测序是能够更准确地研究微生物组及其功能的方法。
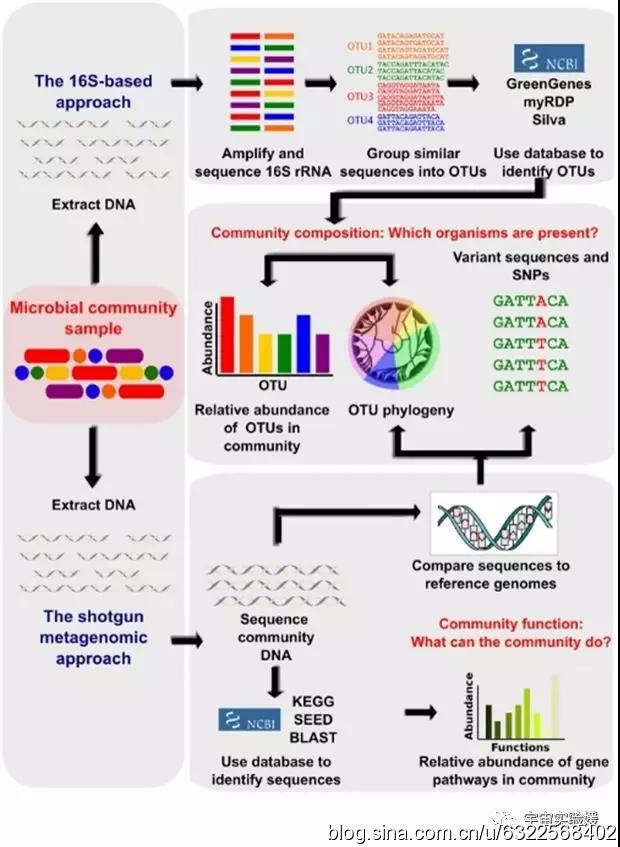
图1. 宏基因组学研究的生物信息工作内容(doi: 10.1371/journal.pcbi.1002808)。
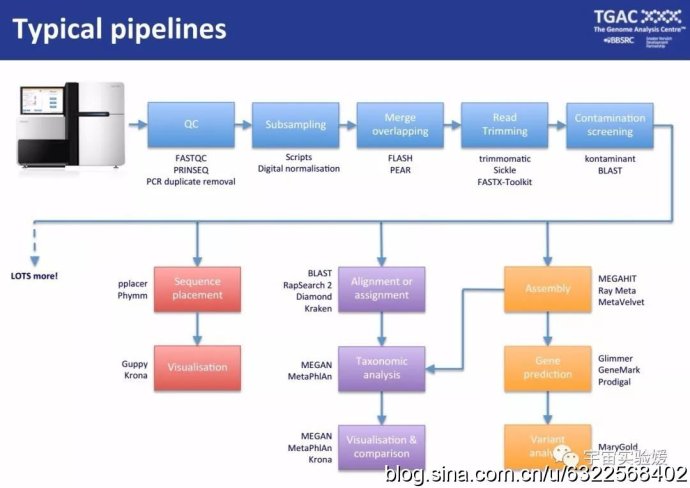
图2. 宏基因组数据处理的基本流程和需用软件(有具体操作需求的同学可以通过下面网址下载英文相关教程https://github.com/TGAC/361Division/tree/master/Metagenomics 2015)
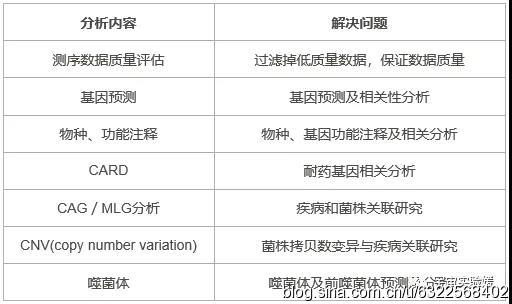
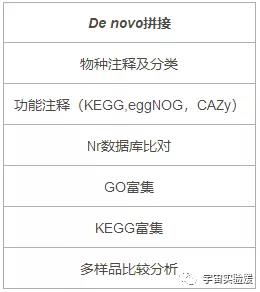
表1. 测序公司提供的基于宏基因组测序数据的标准分析内容
目前很多研究都聚焦在人体肠道微生物组的宏基因组学研究,期望从中挖掘出多种疾病的因果关系。已经采集到的样本来自不同国家、遗传背景、生活习惯、身体状况的人群。同时,环境微生物的宏基因组学研究也在如火如荼地开展中。相比于宿主样本,环境样本(如水体、土壤等)中存在大量未知或不可培养的微生物,这些微生物有很多是尚未被鉴定或者深入研究的。宏基因组测序直接研究环境中的总DNA,为开发新的生物活性物质、发现新的基因和物种、研究特定环境中微生物群落结构与功能的关系(功能网络与互作)、微生物对环境变化的响应与反馈(比如,农耕土壤质量的恶化与恢复)、微生物群落的演替与进化、微生物区域分布与生物地理学等开辟了一条新的途径,为解释和解决一些重大农业和环境问题提供重要依据。
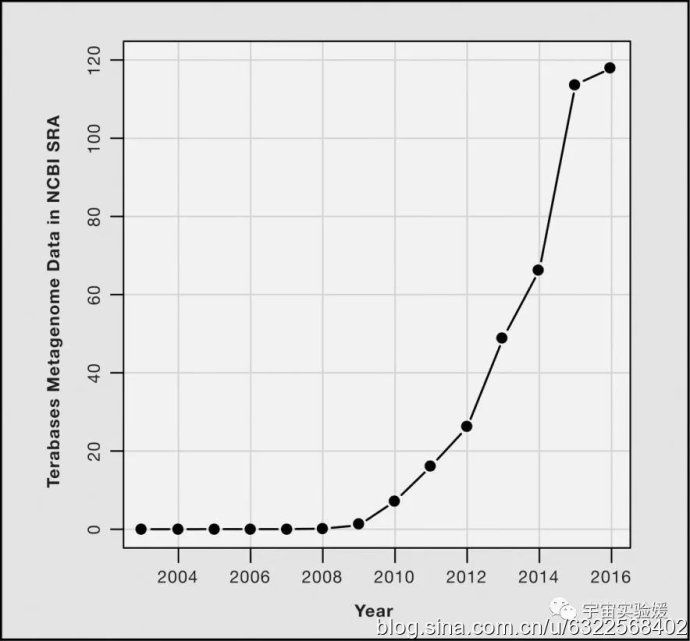
图3. NCBI Sequence Read Archive鸟枪法宏基因组测序数据的增长(doi: 10.1016/j.cell.2016.08.007)
Nature文章实例
以Crits-Christoph和Diamond等人2018年6月发表于Nature的文章为例。研究人员用宏基因组测序的方式,从草地土壤样本中获得了376株细菌基因组,的基因组序列,发现了完全新颖的生物合成基因簇,并描述了它们的基因组学、系统发育学和生态学状况。他们共鉴定出了1599个生物合成基因簇(biosynthetic gene clusters),推测这些基因簇可能合成非核糖体多肽、聚酮化合物等,很多非核糖体多肽合成酶(NPRS)和聚酮合成酶(PKS)都能够合成抗生素、抗真菌素、嗜铁素或免疫抑制剂,所以该研究中着重关注这两类基因簇(图4),以期发现新的抗生素和药用化合物。
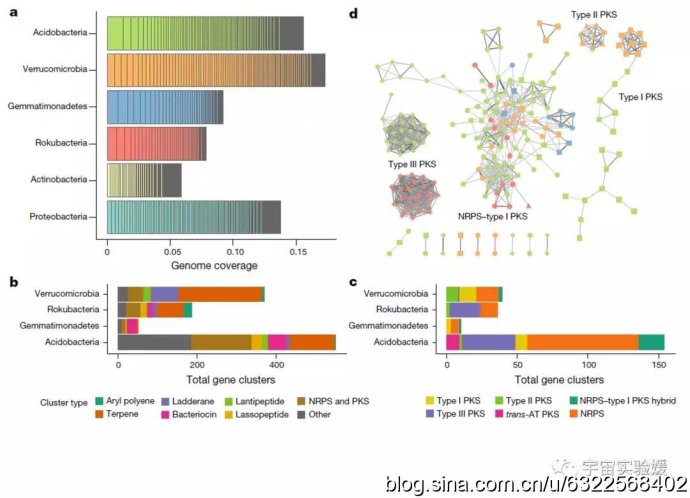
图4. 样本中提取到的基因组和其生物合成基因簇的情况差异展示。a,几个菌门的平均相对丰度。b,生物合成基因簇在几个菌门之间的分布,不同颜色代表由antiSMASH(用于生物合成基因簇的分析;http://antismash.secondarymetabolites.org)推测的产物类型c,研究鉴定到了240个NRPS、PKS(根据酶结构域的组成差异分为型、型和型)和NRPS-PKS混合型基因簇,以及86个可能不完整的基因簇。尽管这些基因含量水平各异,但是由于这些酶具有保守的结构域,因此他们的生物合成途径是可识辨的。d,生物合成基因簇的网络图,连线代表两端具有共有基因,共有基因所占比例越大连线越粗、颜色越深。由该图可知,Verrucomicrobia、Acidobacteria和Rokubacteria中存在不同且稀疏的NRPS和PKS系统,而多数稀有型NRPS基因簇之间的距离较为疏远。在Rokubacteria和Acidobacteria的一支中保守的型PKS基因簇形成了一个密集的网络集群,高度的保守性可能意味着一类新的代谢物的广泛分布。
图5. Acidobacteria中的生物合成NRPS/PKS基因簇。Acidobacteria中发现了两个几乎含有完整NRP和PKS基因簇的基因组,它们分别被命名为Candidatus Eelbacter (Eelbacter_gp4_AA13)和Candidatus Angelobacter (Angelobacter_gp1_AA117)。通过对Acidobacteria基因组中核糖体蛋白序列进行系统发育学分析,发现两个基因组均进化出独特的生物合成操纵子(5a)。Candidatus Angelobacter基因组中含有多种抗生素合成蛋白、一种细菌素合成簇、型和型分泌系统的多基因操纵子元件和一些含有RHS重复结构的大蛋白;Candidatus Eelbacter基因组包含6个长度超过45kb的复合型NRPS-PKS混合型基因簇(5b)。
宏转录组
宏转录组测序是指从整体水平上研究某一特定环境、特定时期群体生命全部基因组转录情况以及转录调控规律的研究手段。它以生态环境中的全部RNA为研究对象,提取环境微生物群落中的全部转录本,进行高通量测序和生物信息学分析,与宏基因组研究相辅相成,能够很好地揭示复杂微生物群落的变化,有效地扩展微生物资源的利用空间。
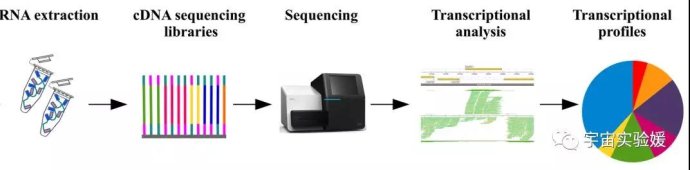
图6. 宏转录组分析简化流程(doi: 10.1007/s00253-018-8976-7)
表2. 测序公司提供的基于宏转录组测序数据的标准分析内容
Nature文章实例
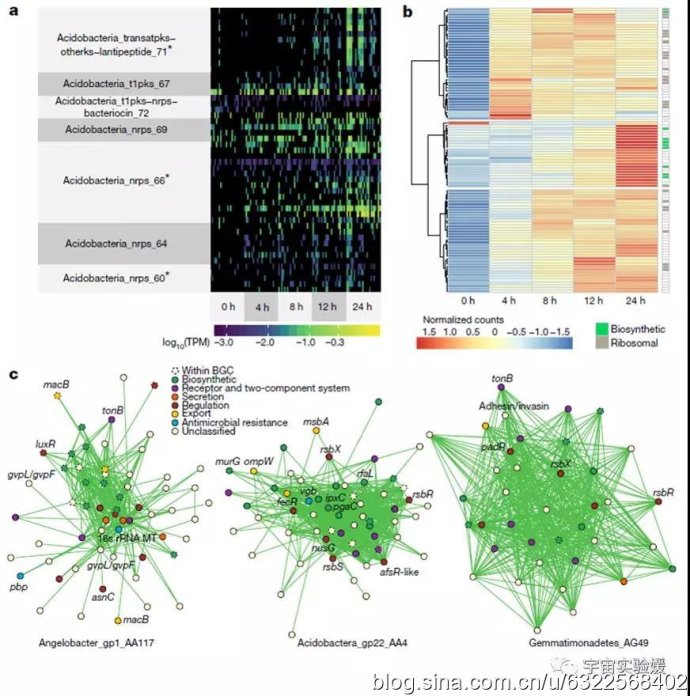
还是同一篇文章,结合宏转录组测序和Kallisto量化分析发现,在133个差异表达的NRPS和PKS基因簇中,共检测到198个NRPS/PKS基因的表达。在4个微生物门类中均检测到了NRPS和PKS的表达,并且酸杆菌门有84个活性基因簇产生了表达。本文还检测了Candidatus Eelbacter的10个生物合成基因簇(含11个NRPS/PKS结构域基因)和Candidatus Angelobacter的25个生物合成基因簇(含25个NRPS/PKS结构域基因),发现7个基因组中有10个NRPS/PKS基因簇在24h修正试验过程中,表现出时间依赖性。
Candidatus Angelobacter中有5个生物合成基因簇与多种感知、响应环境的基因具有共表达模型,如ToB(嗜铁素摄取受体)、MacB(大环内酯转运物)、pbp(青霉素结合蛋白)、16s rRNA MT(16s rRNA甲基转移酶)和gvp(气泡蛋白)等(图7c)。Angelobacter多个基因簇的生物合成基因表达是同步的,表明这些基因能够协同响应生态竞争。此外,Acidobactera_gp22_AA4和Gemmatimonadetes_AG49也具有多个重要的共表达基因,如rsb X/R/S(应激响应调节操纵子)、vgb(维及霉素同源酶B)和ToB等(图7c)。
图7. 生物合成基因的宏转录组。在添加了底物后12-24h,Candidatus Angelobacter基因组中几个基因簇的基因表达水平显著升高(7a),并且一些生物合成基因的表达与核心核糖体基因的表达不一致(7b)。表明Candidatus Angelobacter能够对水和基质的添加产生响应,并且在核心代谢基因的表达量上升数小时后单独调控次级代谢产物合成相关基因的表达。从基因共表达模型来看,7个基因组中有4个发生了显著的次级代谢基因共表达富集(p<0.05)(7c)。
简单来说,宏基因组能够说明菌群“能做什么”,而宏转录组则是“要做什么”。尤其是在不同取样时间点之间和实验条件处理前后,宏转录组往往会发生明显变化,据此可以建立菌群功能与影响因子之间的关联性。后面我们还会介绍表征菌群“做了什么”和“做出了什么”的研究方法,敬请关注。
(部分定义源自网络)