题目背景
大家都知道,基因可以看作一个碱基对序列。它包含了4种核苷酸,简记作A,C,G,T。生物学家正致力于寻找人类基因的功能,以利用于诊断疾病和发明药物。
在一个人类基因工作组的任务中,生物学家研究的是:两个基因的相似程度。因为这个研究对疾病的治疗有着非同寻常的作用。
题目描述
两个基因的相似度的计算方法如下:
对于两个已知基因,例如AGTGATG和GTTAG,将它们的碱基互相对应。当然,中间可以加入一些空碱基-,例如:
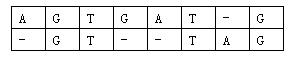
这样,两个基因之间的相似度就可以用碱基之间相似度的总和来描述,碱基之间的相似度如下表所示:
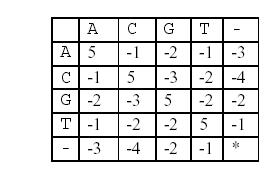
那么相似度就是:(-3)+5+5+(-2)+(-3)+5+(-3)+5=9。因为两个基因的对应方法不唯一,例如又有:
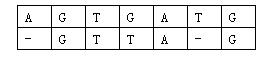
相似度为:(-3)+5+5+(-2)+5+(-1)+5=14。规定两个基因的相似度为所有对应方法中,相似度最大的那个。
输入输出格式
输入格式:
共两行。每行首先是一个整数,表示基因的长度;隔一个空格后是一个基因序列,序列中只含A,C,G,T四个字母。1<=序列的长度<=100。
输出格式:
仅一行,即输入基因的相似度。
输入输出样例
输出样例#1:
14
Solution
这道题的思路算比较简单.
状态定义可以很快想出来.即用 f[ i ][ j ] 表示匹配到前一个DNA序列的 第 i 个和第二个DNA串的 第 j 个的最大相似度.
然后前导状态也很容易想:
1. f [ i-1 ][ j-1 ] 此时即用当前两个DNA排在一起. 加上这两个的相似度.
2. f [ i-1 ][ j ] 此时即 前一个DNA序列的第 i 个和空格匹配.
3. f [ i ][ j-1 ] 此时即 后一个DNA序列的第 j 个和空格匹配.
然后这道题的预处理其实才是最坑的.
因为它有可能前面一整个字符串都用空格匹配.
代码
#include<bits/stdc++.h> using namespace std; int c[5][5]={ 0,-3,-4,-2,-1, -3,5,-1,-2,-1, -4,-1,5,-3,-2, -2,-2,-3,5,-2, -1,-1,-2,-2,5, }; int n,m; int nn[108],mm[108]; int f[108][108]; int main() { cin>>n; for(int i=1;i<=n;i++) { char ch; cin>>ch; if(ch=='A')nn[i]=1; if(ch=='C')nn[i]=2; if(ch=='G')nn[i]=3; if(ch=='T')nn[i]=4; } cin>>m; for(int i=1;i<=m;i++) { char ch; cin>>ch; if(ch=='A')mm[i]=1; if(ch=='C')mm[i]=2; if(ch=='G')mm[i]=3; if(ch=='T')mm[i]=4; } for(int i=1;i<=n;i++) f[i][0]=f[i-1][0]+c[0][nn[i]]; for(int i=1;i<=m;i++) f[0][i]=f[0][i-1]+c[0][nn[i]]; //预处理部分 for(int i=1;i<=n;i++) for(int j=1;j<=m;j++) f[i][j]=max(f[i-1][j-1]+c[nn[i]][mm[j]],max(f[i-1][j]+c[0][nn[i]],f[i][j-1]+c[0][mm[j]])); cout<<f[n][m]<<endl; }