题目描述
两个基因的相似度的计算方法如下:
对于两个已知基因,例如AGTGATG和GTTAG,将它们的碱基互相对应。当然,中间可以加入一些空碱基-,例如:
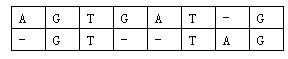
这样,两个基因之间的相似度就可以用碱基之间相似度的总和来描述,碱基之间的相似度如下表所示:
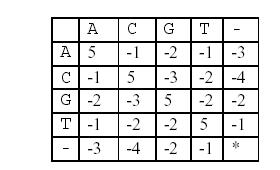
那么相似度就是:(−3)+5+5+(−2)+(−3)+5+(−3)+5=9。因为两个基因的对应方法不唯一,例如又有:
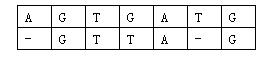
相似度为:(−3)+5+5+(−2)+5+(−1)+5=14。规定两个基因的相似度为所有对应方法中,相似度最大的那个。
输入输出格式
输入格式:
共两行。每行首先是一个整数,表示基因的长度;隔一个空格后是一个基因序列,序列中只含A,C,G,T四个字母。1≤1≤序列的长度≤100。
输出格式:
仅一行,即输入基因的相似度。
输入输出样例
输入样例#1:
7 AGTGATG
5 GTTAG
输出样例#1:
14
思路:
我们设dp【i】【j】为第一个基因的1~i位和第二个基因的1~j位的最大相似值,那么可以得到递推公式dp【i】【j】=max(dp【i-1】【j】+ maps【i】【-】,dp【i】【j-1】 + maps【-】【j】,dp【i - 1】【j - 1】+ maps【i】【j】)
代码:
#include <stdio.h>
#include <algorithm>
#include <map>
using namespace std;
int maps[5][5] = {
5, -1, -2, -1, -3,
-1, 5, -3, -2, -4,
-2, -3, 5, -2, -2,
-1, -2, -2, 5, -1,
-3, -4, -2, -1, 0
};
char s1[105], s2[105];
int dp[105][105] = {0};
map<char, int>zcy;
int main () {
int n, m;
zcy['A'] = 0;
zcy['C'] = 1;
zcy['G'] = 2;
zcy['T'] = 3;
scanf("%d%s", &n, s1 + 1);
scanf("%d%s", &m, s2 + 1);
for (int i = 1; i <= n; i++) {
dp[i][0] = dp[i - 1][0] + maps[zcy[s1[i]]][4];
}
for (int i = 1; i <= m; i++) {
dp[0][i] = dp[0][i - 1] + maps[4][zcy[s2[i]]];
}
for (int i = 1; i <= n; i++) {
for (int j = 1; j <= m; j++) {
dp[i][j] = max(max(dp[i - 1][j] + maps[zcy[s1[i]]][4],
dp[i][j - 1] + maps[4][zcy[s2[j]]]),
dp[i - 1][j - 1] + maps[zcy[s1[i]]][zcy[s2[j]]]);
}
}
printf("%d\n", dp[n][m]);
return 0;
}
如果有写的不对或者不全面的地方 可通过主页的联系方式进行指正,谢谢