简介
Enrichr是一个基于网页端的综合性的基因集富集分析工具。富集分析是用于分析由全基因组实验产生的基因集合的流行方法。在这里,我们介绍一个重要的更新到这个域名为Enrichr的工具(此为第二版)。 Enrichr目前包含大量可用于分析和下载的基因组库。 Enrichr总共包含来自102个基因集库的180 184个注释基因集。 Enrichr增加了新功能,包括提交模糊集,上传BED文件,改进的应用程序编程接口以及将结果可视化为簇的能力。总的来说,Enrichr是一个综合资源,用于策划的基因组和搜索引擎,为进一步的生物学发现积累生物学知识。
Enrichr网页的链接为http://amp.pharm.mssm.edu/Enrichr
Enrichr网站的主页:
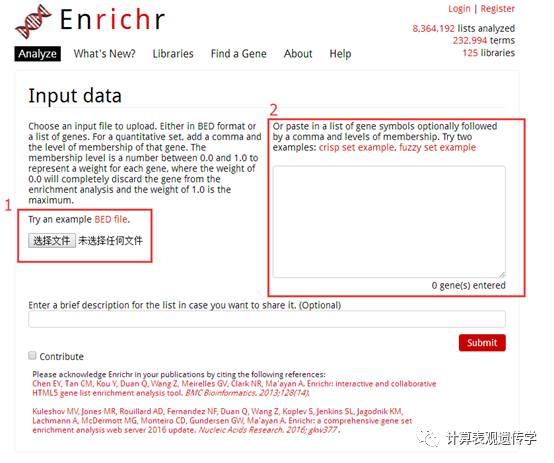
其中“1”部分需要提交.bed文件,.bed文件中应该包含染色体号,基因的起始位置和终止位置以及正负链的信息,如下是一个示例的.bed文件
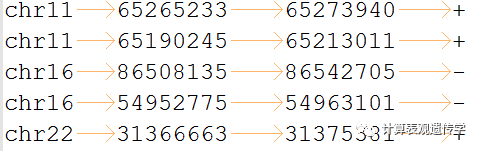
在我们提交这个.bed文件后,会出现如下信息

这里注意“3”的选项,要选择和你的.bed文件版本号相同的注释。
Ps:由于网络或者网页端服务器的问题,有的时候“3”并不会在你提交文件之后立马弹出,需要耐心等待一会。一定要选择对应的版本哦。
当然我们也可以直接提交自己的基因列表进行富集分析,这里使用enrichr提供的样本进行分析,结果如下:

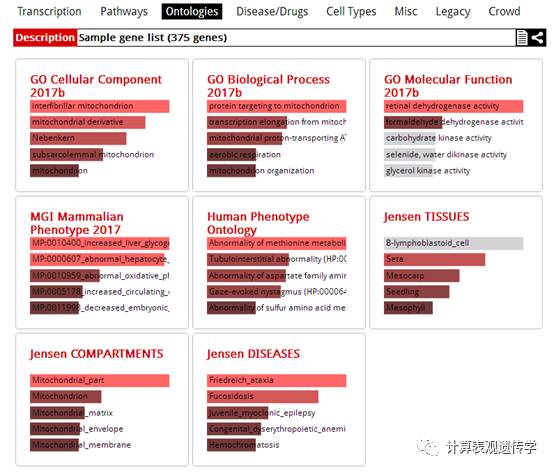
可以看到,里面包含了几乎所有的基因集富集分析所能分析到的方面,对于我们常用的GO,KEGG等分析都包含在内,点开单独的矩形。

会出现该模块的详细信息,而且这个图是可以直接导出的哦。
点击“Table”。
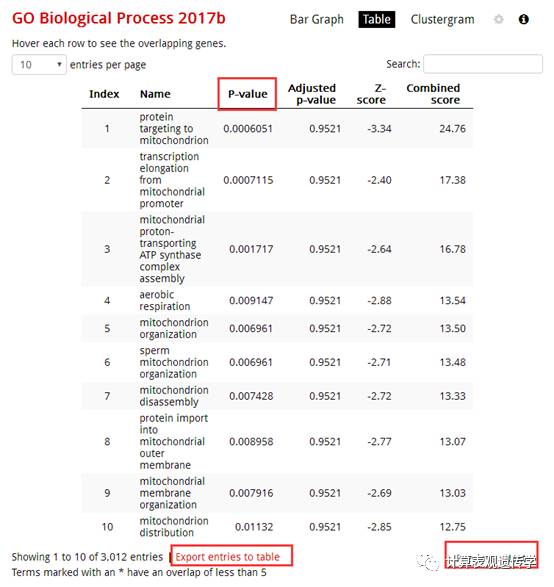
可以看到详细的参数,点击“P-value”,可以按照p值对表格进行排序。同时我们也可以点击“exprot”讲表格里的内容导出进筛选。
Ps:从小编自己的使用经历来说,这个“export”有时候会失灵,如果失灵请同学们自己复制网页上的内容即可。
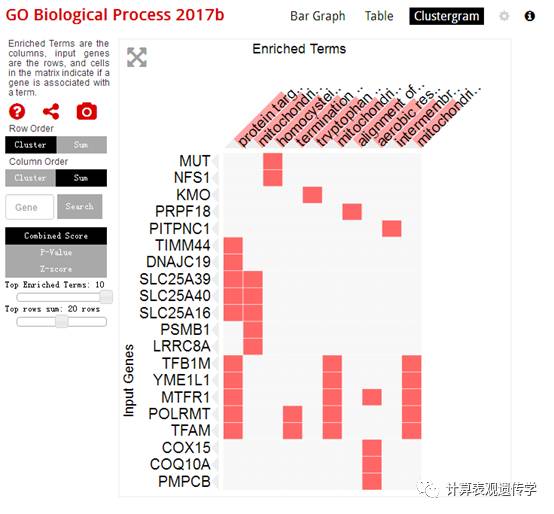
同时,“enrichr”还提供了对基因集聚类的功能,点击“Clustergram”可以得到聚类图:
小结:
enrichr这个网站提供了全面的,便捷的基因集富集分析的功能,几乎可能做到“一站在手,富集我有”。除了本文提到的常用功能外,还有很多其他的功能,推荐同学们读一遍这篇文献,文献在pubmed上是免费的哟。
全文链接:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4987924/
参考文献:
Kuleshov, M.V., et al., Enrichr:a comprehensive gene set enrichment analysis web server 2016 update.Nucleic Acids Res, 2016. 44(W1): p.W90-7.
往期「精彩内容」,点击回顾
DNA测序历史 | CircRNA数据库 | Epigenie表观综合 | 癌症定位
BWA介绍 | 源码安装R包 | CancerLocator | lme4 | 450K分析
乳腺癌异质性 | BS-Seq | 隐马模型 | Circos安装 | Circos画图
KEGG标记基因 | GDSC | Meta分析 | R线性回归和相关矩阵
精彩会议及课程,点击回顾