文章:Genome-reconstruction for eukaryotes from complex natural microbial communities | bioRxiv
推荐使用conda进行安装:
conda create -y -n eukrep-env -c bioconda scikit-learn==0.19.2 eukrep或者通过pip安装(需预先安装scikit-learn版本0.19.2):
pip install EukRep示例用法
-
从fasta文件中识别并输出预测为真核起源的序列:
EukRep -i <Sequences in Fasta format> -o <Eukaryote sequence output file> -
同时识别并分别从fasta文件中输出真核和原核起源的序列:
EukRep -i <Sequences in Fasta format> -o <Eukaryote sequence output file> --prokarya <Prokaryote sequence output file>
获取真核生物bins EukRep设计用于作为更大规模分析流程的一部分。如需根据“从复杂自然微生物群落中重建真核生物基因组”(West等人,待审阅)一文中所述的方法获得高质量的基因预测及对已识别的真核contigs进行分箱,请参阅以下方法部分: Genome-reconstruction for eukaryotes from complex natural microbial communities | bioRxiv
- 或者
查看提供的示例工作流程(正在进行中): https://github.com/patrickwest/EukRep_Pipeline
调整筛选严格度 可以通过-m参数调整识别真核contig的严格度。以下是严格、平衡和宽松模式下的假阳性率(FPR)和假阴性率(FNR)。默认设置为平衡模式。在0.6.5版本之前,默认设置为宽松模式。
在对模拟新型门类基因组生成的20kb和5kb片段化支架运行EukRep后,获得了如下所示的数据:
20kb

5kb
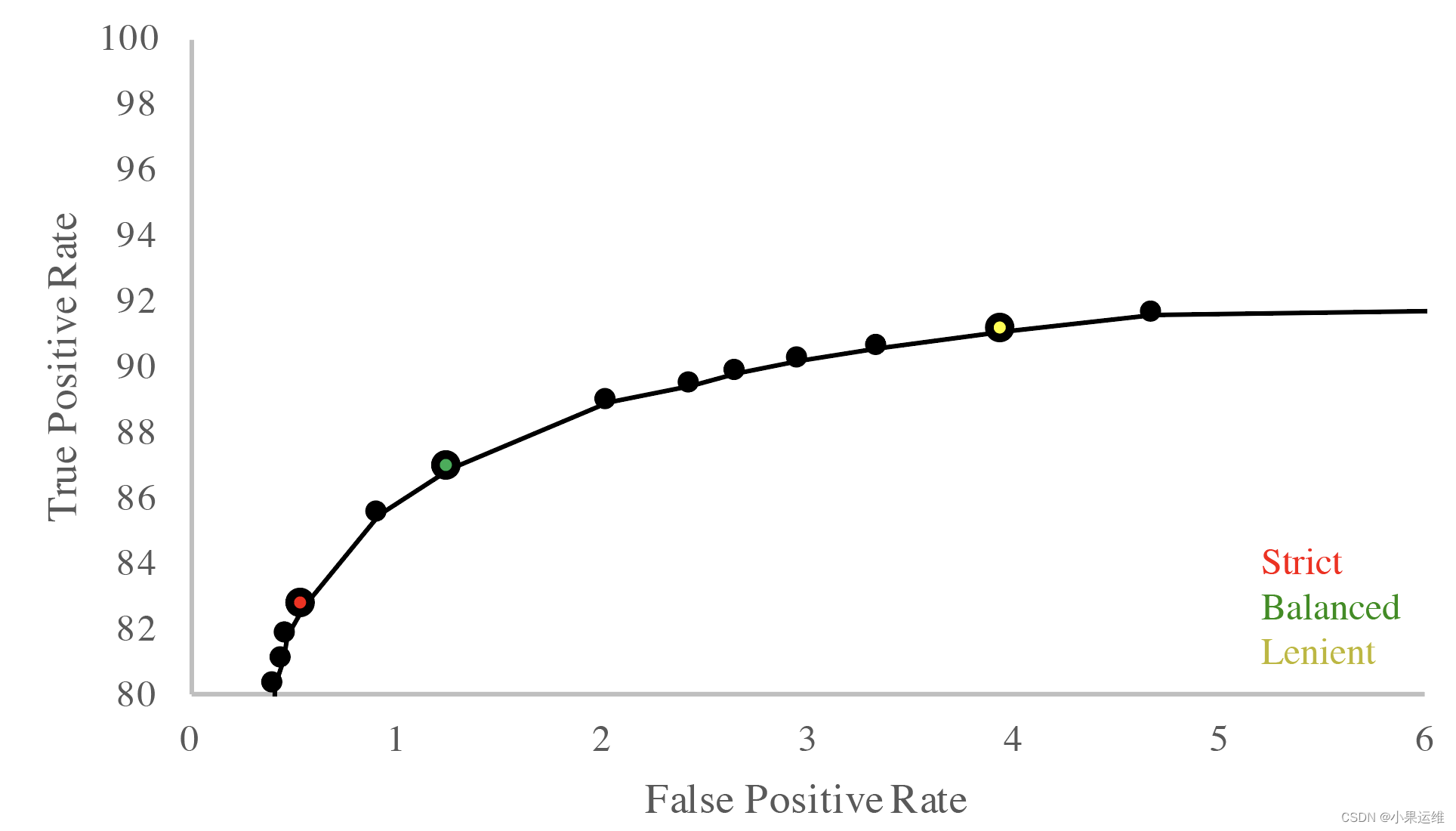
此处未给出具体的数据内容,但可根据上述描述理解,在不同长度的支架上应用EukRep,可以得到不同筛选严格度下对应的假阳性和假阴性结果。
典型使用流程:官方推荐
patrickwest/EukRep_Pipeline (github.com)
EukRep_Pipeline 这是一个结合EukRep从元基因组中分箱真核生物基因组的示例工作流程。其中包含了一个示例bash脚本euk_pipeline.sh,整合了以下所有步骤。
要求:
- 预先组装完成的shotgun元基因组样本及其每条scaffold覆盖度信息
- EukRep工具
- CONCOCT或metabat工具
- genemark-ES
- MAKER2
- BUSCO
- 可选但推荐:pyenv
使用EukRep分类 在预先组装的shotgun元基因组样本上运行EukRep:
EukRep -i metagenome.fa -o euk_contigs.fa如果你处理的是高度复杂或片段化的元基因组,建议降低最小contig长度阈值:
EukRep -i metagenome.fa -o euk_contigs.fa --min 1000自动分箱 此步骤对于分离样本中的多个真核生物基因组至关重要。 为了获得尽可能高质量的基因预测结果,在进行基因预测前必须将基因组分开。 需要每条scaffold的覆盖度信息 使用CONCOCT执行:
concoct --coverage_file euk_contig_cov.txt --composition_file euk_contigs.fa
mkdir clusters
python /path/to/CONCOCT/scripts/extract_fasta_bins.py --output_path ./clusters/ euk_contigs.fa clustering_gt1000.csv使用metabat执行:
metabat -a euk_contig_cov.txt -i euk_contigs.fa -o bin -t 6按bin大小过滤 我们发现在此阶段过滤掉小于2.5 Mbp的bin非常有用。这种过滤可以去除大部分假阳性结果,尤其是当使用CONCOCT时,因为CONCOCT会对每一条scaffold进行分箱,往往会产生许多非常小的bin。训练GeneMark-ES
perl gmes_petap.pl --ES -min_contig 10000 --sequence bin_1.fa-min_contig选项指定了用于训练指定bin基因预测模型的最小contig长度。并非需要该bin中的每个contig都被用于训练,但如果超过阈值的contig过少,训练可能失败。由于许多来自元基因组的bin通常十分碎片化,所以可能需要调整这个选项。
使用训练好的GeneMark-ES模型和MAKER2预测基因 MAKER使用控制文件。至少建议以以下方式修改它们,以便使用RepeatMasker和GeneMark-ES进行基因预测: 在'maker_opts.ctl'文件中:
keep_preds=1
gmhmm=/path/to/output/gmhmm.mod然后使用6个核心运行MAKER:
maker -g bin_1.fa -c 6 cd *.maker.output fasta_merge -d *_master_datastore_index.log -o bin_1为了进一步提高基因预测质量,MAKER能够整合来自相关物种参考基因组的同源蛋白质、转录组证据以及其他如AUGUSTUS等的ab initio基因预测器。为了获取高质量的基因预测,通常最好利用所有可用的这些证据来源。
对许多元基因组样本而言,进行ab initio基因预测可能是唯一可选择的方法。运行BUSCO
python3 BUSCO.py -i *.maker.proteins.fasta -l eukaryota_odb9 -o bin_1 -m protBUSCO将在你的bin内寻找单拷贝直系同源基因(SCGs),提供一个完整性估计(以及粗略的重复单拷贝基因污染评估)。 -l参数指定了要使用的SCG谱系集。我们通常使用eukaryota_odb9因为它最通用,然而当你对你的bin所属的生物类型有了更清晰的认识后,也可以选择使用更具体的谱系集。