从NCBI下载数据本来是一件很简单的事情,但是今天碰到几个坑:
1、paper里没有提供SRA数据号、也没有提供路径;
2、不知道文件在ftp的地址,不能直接用wget下载
所以通过在NCBI官网,直接在SRA搜索栏里:
 输入paper的title关键词NIFTY BGI
输入paper的title关键词NIFTY BGI

搜索结果:

选一个文件点击进去
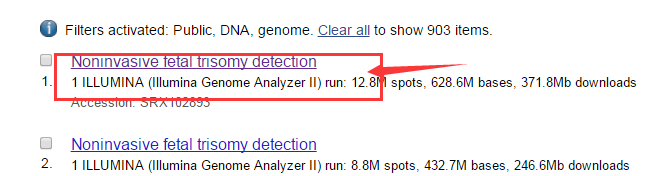
进去之后,再点击SRP
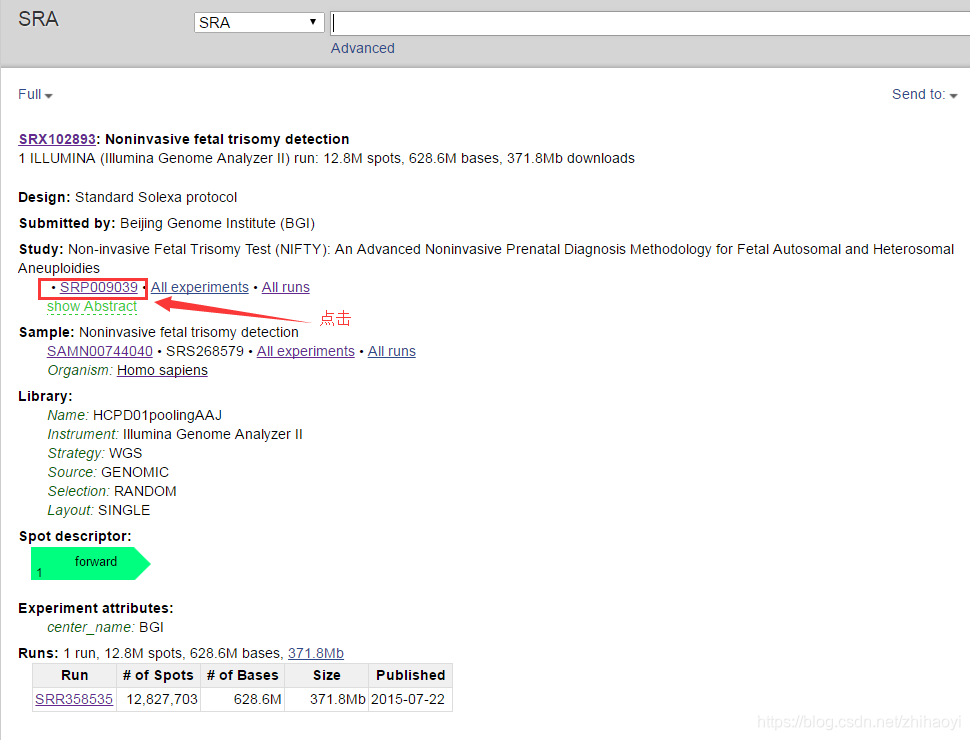
然后:
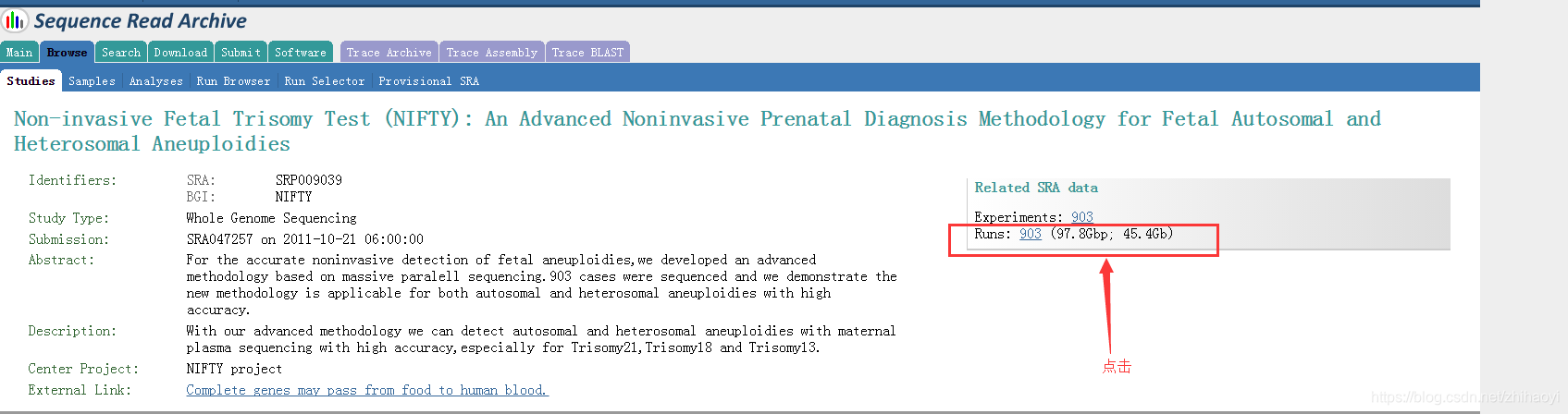
出现如下内容:
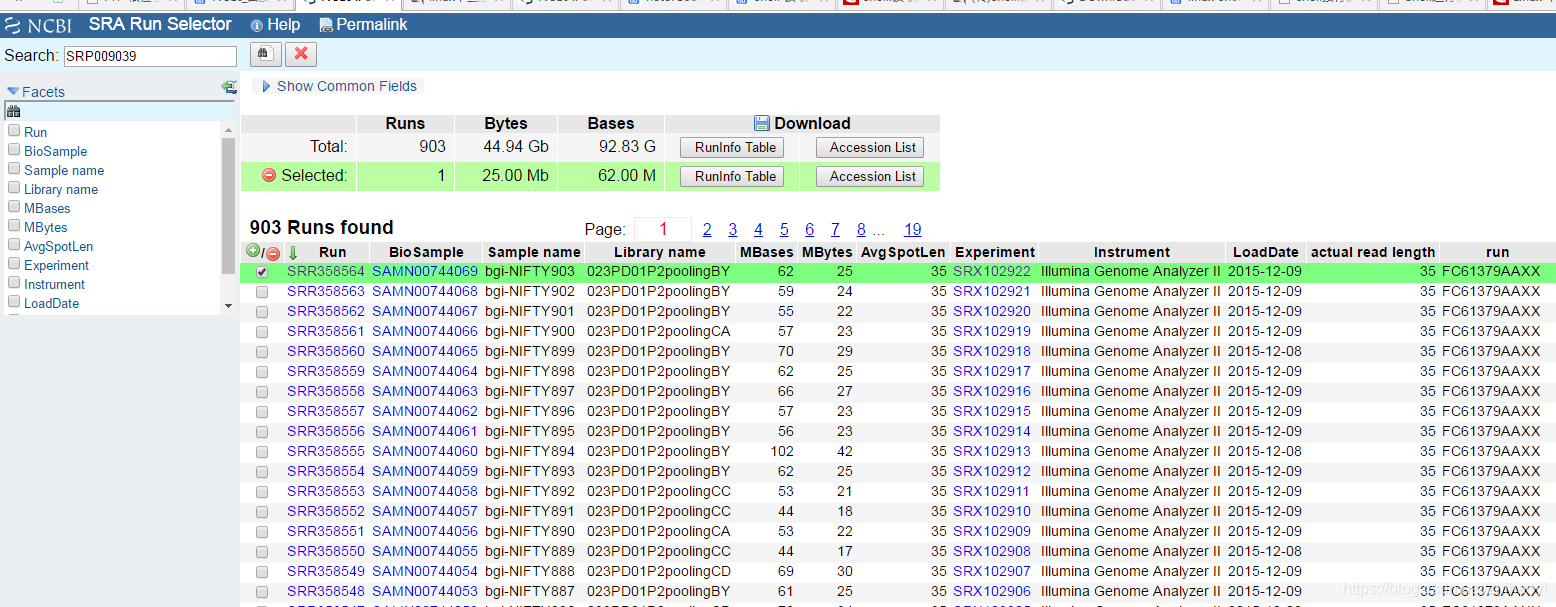
然后选择所有SRR文件:
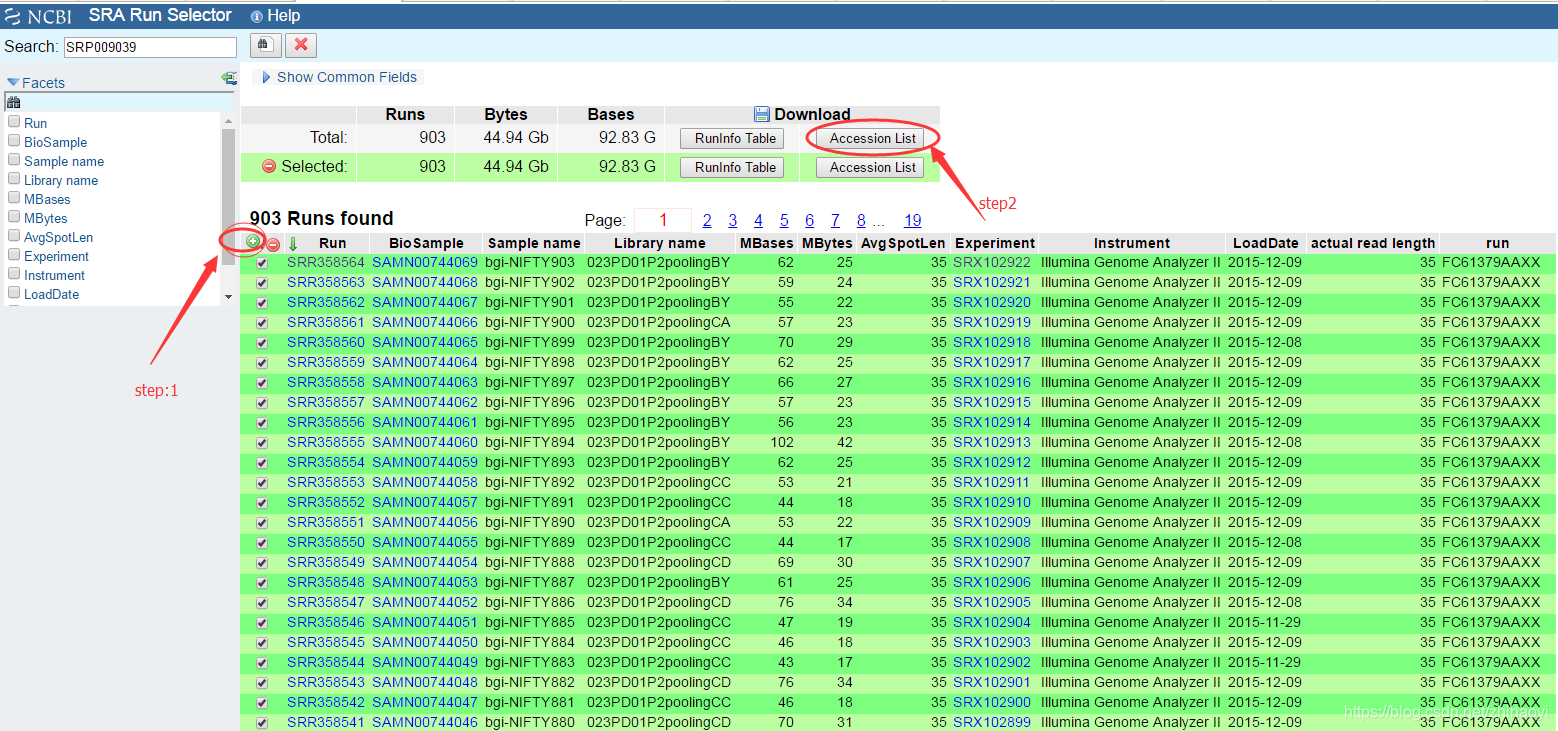
下载Accession list之后得到文件列表:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
SRR354208
SRR357358
SRR357397
SRR357398
SRR357666
SRR357667
SRR357668
SRR357669
SRR357670
SRR357671
SRR357672
SRR357673
SRR357674
SRR357675
SRR357676
然后根据这个列表在linux下载:
1
2
3
4
5
[wuzengding@mn01 NIFTY_BGI_samp]$ cat /data1/Medicine/WZD/SRR_Acc_List.txt | while read line
do
echo $line
/home/wuzengding/biosoftware/sratoolkit.2.8.2-1-centos_linux64/bin/fastq-dump.2.8.2 ${line}
done
下载成功!!
原文 https://www.cnblogs.com/zdwu/p/8473986.html
扫描二维码关注公众号,回复:
9634485 查看本文章


