简介
- 序列读取存档(sequence Read Archive,SRA)数据是可通过多个云提供商和NCBI服务器获得的,它是最大的可公开获得的高通量测序数据存储库。
- SRA接受来自生活各个部门以及宏基因组学和环境调查的数据。
- SRA存储原始测序数据和比对信息,以提高可重复性并通过数据分析促进新发现。
- NCBI网站储存二代测序原始数据的数据库
- SRA官网
从Entrez搜索结果中下载SRA序列
例如查找SRA Entrez中BALB / c小鼠淋巴结组织的RNA-Seq记录
获取搜索结果
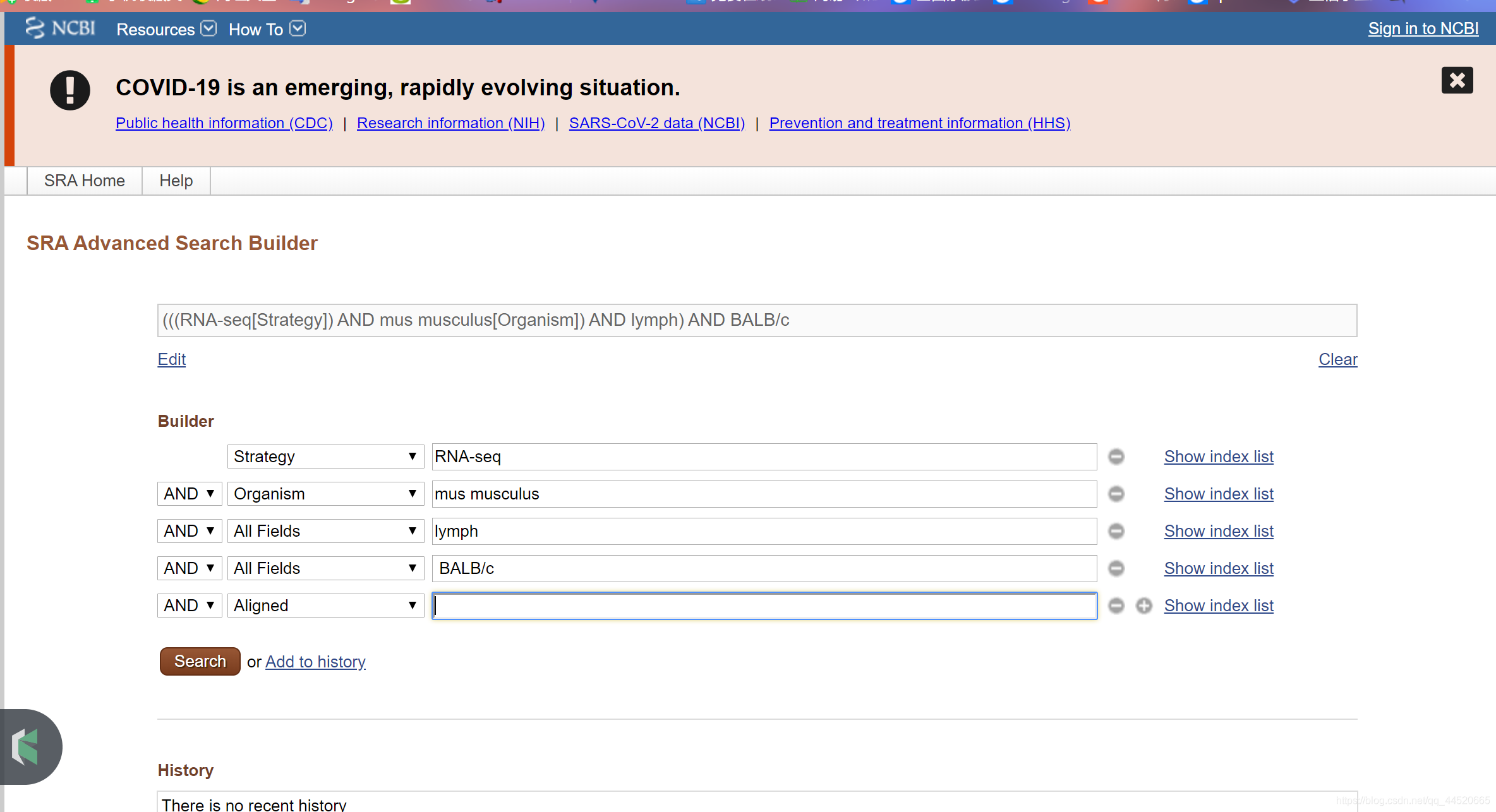
- SRA search中高级搜索: (((“mus musculus”[Organism]) AND BALB/c*) AND “lymph*”) AND “rna seq”[Strategy]
- 要将您的搜索限制为仅aligned数据,请添加到上述查询中AND alignment data [属性]。
- 单击记录(实验)旁边的复选框以选择感兴趣的数据。 取消选中所有复选框,以从搜索中选择所有记录(实验)。
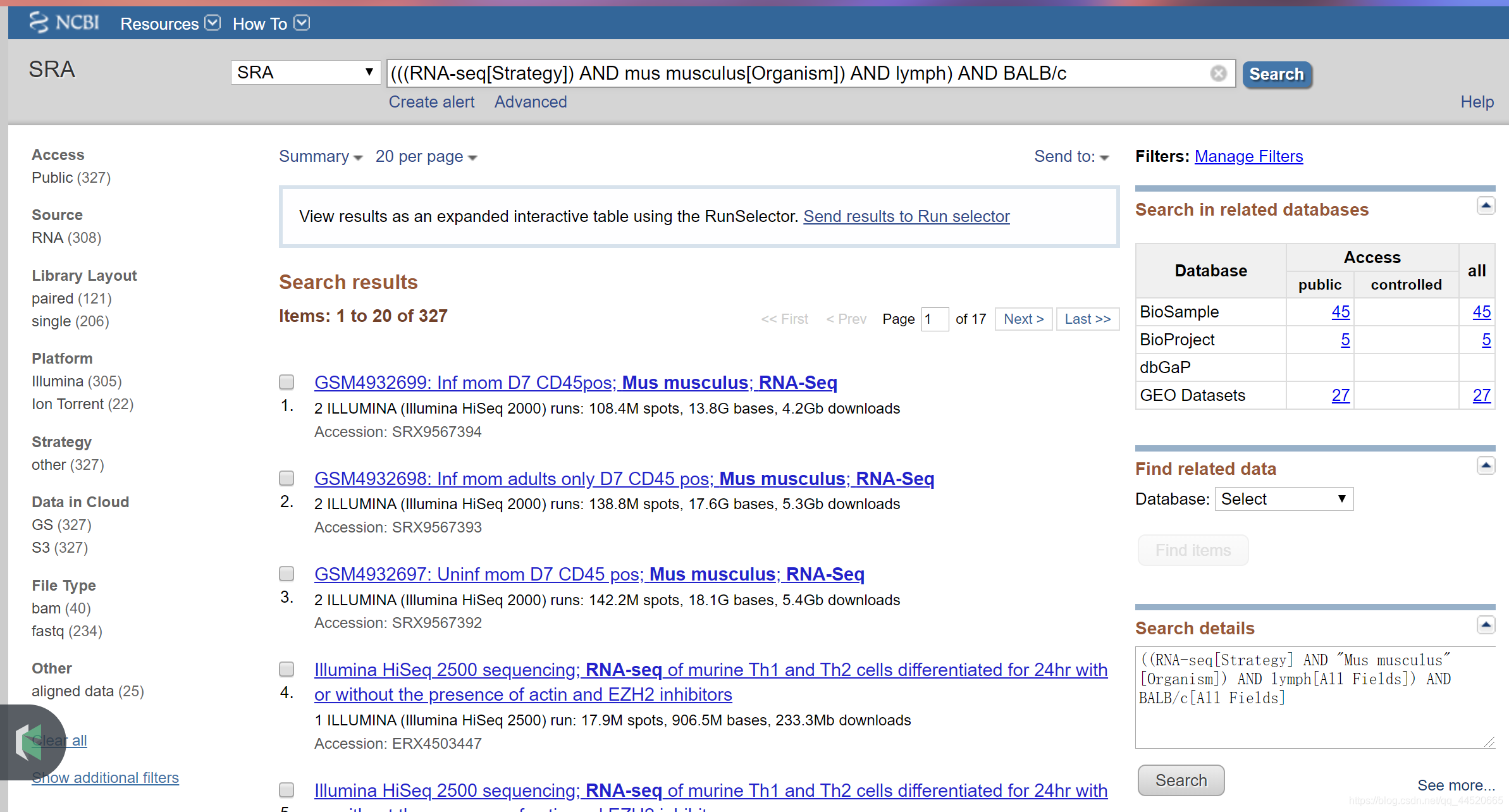
获得run accessions
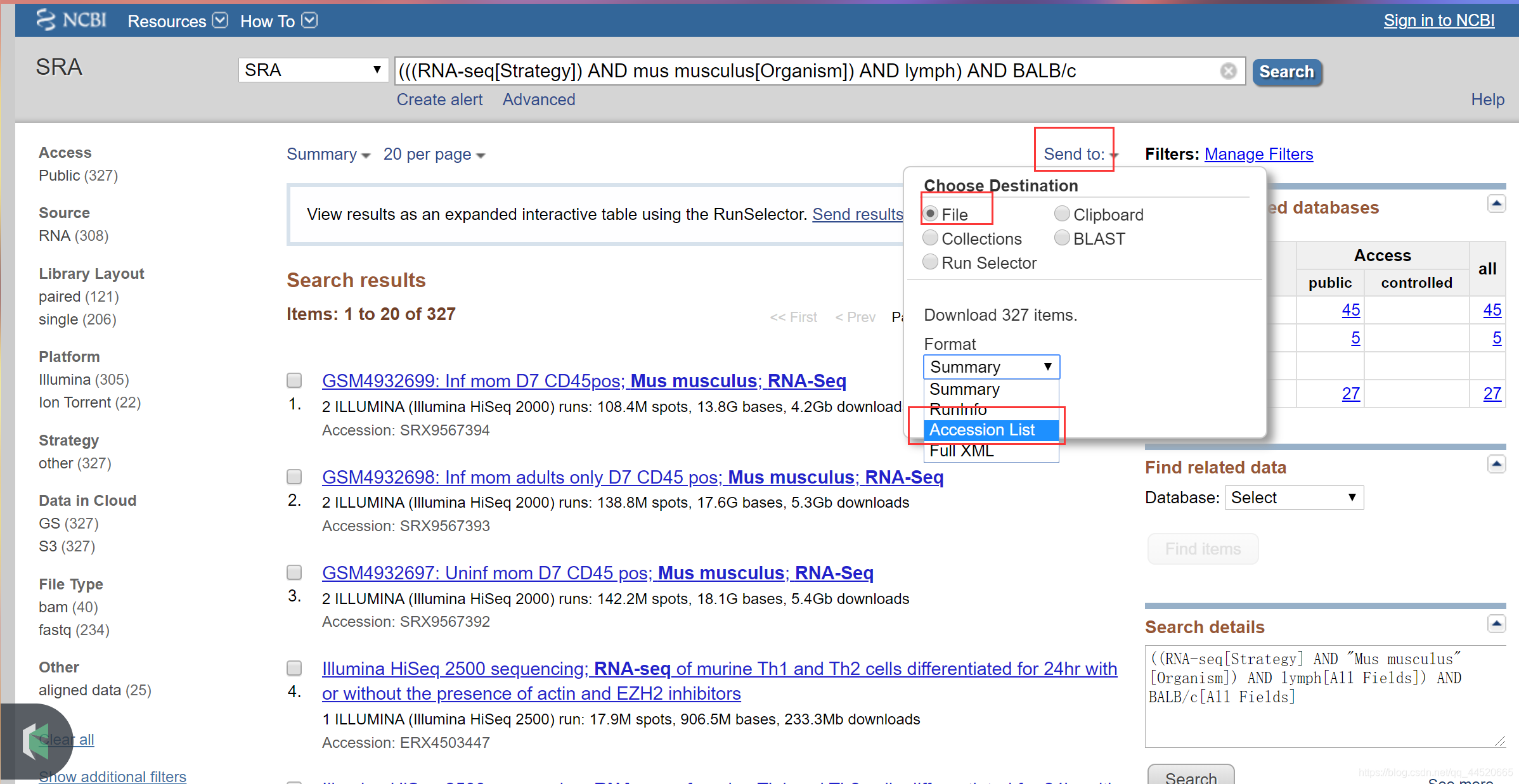
run accessions用于下载SRA数据,要下载你的Entrez搜索中选择的run accessions的列表,并执行以下操作:
- 点击页面顶部的send to,选中单选按钮File,选择Accession List
- 将此文件保存在运行SRA工具包的位置
Sraacclist.txt 文件的格式如下图:
SRR11192680
SRR11192681
SRR11192682
SRR11192683
SRR11192684
使用SRA工具包下载序列数据文件
- SRA运行文件仅包含序列数据,不包含链接到运行的任何元数据(样品信息等)
- 请确保你正在运行该工具包的最新版本,因为较早的版本可能与最新加载的数据或最新的网络协议不兼容
安装SRA Toolkit:
- 下载sratoolkit.current-win64.zip:http://ftp-trace.ncbi.nlm.nih.gov/sra/sdk/current/sratoolkit.current-win64.zip
- 路径:C:\Users\xuyunfeng\Desktop\sratoolkit.current-win64\sratoolkit.2.10.9-win64
- 打开command shell, 例如开始或运行== cmd.exe==
- cd到你解压zip文件的目录
- cd bin

配置SRA Toolkit
- 只有一小部分选项需要启用才能访问云中的公共和受控访问数据。 要开始配置,请运行:vdb-config -i
- 您将看到一个屏幕,您可以在其中操作按钮,方法是按红色突出显示的字母,或者按Tab键直到到达所需的按钮,然后按空格键或Enter键。
- 您要在主屏幕上启用“Remote access”选项。
- 转到“高速缓存Cache”选项卡,您将在其中启用“本地文件缓存”,并设置“用户存储库的位置”。
- 存储库目录需要设置为空文件夹。 这是预取将存放文件的文件夹。
- 转到您的云提供商标签并接受“报告云实例身份”
- 云实例身份仅报告您正在使用的云(AWS v GCP),因此您可以免费访问数据。
检查toolkit是否可用
fastq-dump --stdout -X 2 SRR390728
几秒后,命令生成如下输出
Read 2 spots for SRR390728
Written 2 spots for SRR390728
@SRR390728.1 1 length=72
CATTCTTCACGTAGTTCTCGAGCCTTGGTTTTCAGCGATGGAGAATGACTTTGACAAGCTGAGAGAAGNTNC
+SRR390728.1 1 length=72
;;;;;;;;;;;;;;;;;;;;;;;;;;;9;;665142;;;;;;;;;;;;;;;;;;;;;;;;;;;;;96&&&&(
@SRR390728.2 2 length=72
AAGTAGGTCTCGTCTGTGTTTTCTACGAGCTTGTGTTCCAGCTGACCCACTCCCTGGGTGGGGGGACTGGGT
+SRR390728.2 2 length=72
;;;;;;;;;;;;;;;;;4;;;;3;393.1+4&&5&&;;;;;;;;;;;;;;;;;;;;;<9;<;;;;;464262
下载公共数据
- Prefetch是SRA工具包的一部分。 该程序下载Runs(压缩的SRA格式的序列文件)以及将Run从SRA格式转换为更常用格式所需的所有其他数据。 Prefetch可用于更正和完成不完整的Run下载
- 使用此prefetch命令以SRA格式从上一示例下载Run
$ prefetch SRR000001
Runs列表:
prefetch --option-file SraAccList.txt
- fastq-dump和sam-dump也是SRA工具包的一部分,可用于将预提取的运行从压缩的SRA格式转换为fastq或sam格式,例如:
fasterq-dump --split-files SRR11180057.sra
- 你还可以通过在fastqq-dump或sam-dump命令中仅输入不带.sra扩展名的Run accession来避免预取步骤并一步一步下载和转换运行:
fasterq-dump --split-files SRR11180057
下载原始提交的文件
- 如果你希望使用原始提交的文件而不是从存档中转储标准化数据,则SRA已将原始提交的文件存放在可通过prefetch命令访问的云存储桶中。
- 参阅使用Amazon Web Services(AWS)下载SRA序列数据
- 例如prefetch命令
prefetch --type fastq SRR11180057
使用==–type==命令可以指定要下载的文件的类型。 你可以在BigQuery的SRA或“运行浏览器”中的“数据访问”选项卡中查找原始文件的文件类型,或使用any获取所有可用格式。
下载受保护的数据
有关如何下载dbGaP数据的信息,请参见:受保护的数据使用指南
下载与SRA数据相关的元数据
从搜索结果页面
- SRA Run文件不包含有关链接到数据本身的元数据的任何信息(示例信息等)。
- 要下载Entrez查询中每个Run的元数据,请单击页面顶部的Send to,选中File单选按钮,然后在下拉菜单中选择RunInfo。
- 这将生成表格形式的SraRunInfo.csv文件,其中包含每个运行可用的元数据。
从Run Selector
可以从Run Selector中以制表符分隔的文件中下载一组稍有不同的元数据
要为Entrez查询中的每个Run下载元数据,请执行以下操作:
- 点击发送到页面的顶部,检查运行选择单选按钮,然后单击按钮进入。
- 如有必要,请使用“ 运行选择器 ”界面提供的各种过滤器来优化结果。
- 单击“ 运行信息表”按钮。这将生成一个表格SraRunTable.txt文件,其中包含每个运行可用的元数据。
从Run Browser下载序列数据
Run Browser允许对HTTP未对齐和对齐的序列进行有限的下载
未对齐序列示例
- 在Run Browser中打开选定的运行。
- 单击Reads选项卡。
通过应用Filter查找某些读物或将 过滤条件字段清空。
点击Filtered Download按钮。
选择可用的下载格式,然后单击Download链接。
比对序列示例
在Run Browser中打开选定的运行。
单击Alignment选项卡。
在下拉菜单中选择可用的下载格式,然后单击Screen或File按钮以将运行输出到屏幕或文件中。