1、数据库简介
UniVec是一个数据库,可用于快速识别核酸序列中可能来自载体来源(载体污染)的片段。使用UniVec进行筛选是高效的,因为已经消除了大量冗余子序列,从而创建一个只包含来自大量载体的每个惟一序列段的一个副本数据库。除了载体序列,UniVec还包含用于克隆cDNA或基因组DNA过程中常用的adpter、linkers和引物的序列。这使得在载体筛选过程中可以发现这些寡核苷酸序列的污染。UniVec可以从NCBI FTP目录获得:ftp://ftp.ncbi.nlm.nih.gov/pub/UniVec
2、VecScreen
VecScreen是一个系统,它可以快速找到核酸序列的片段,这些片段可能来自于载体。它帮助研究人员在分析或提交序列之前识别和删除任何载体源片段。研究人员被鼓励使用VecScreen搜索页面上的表单对其序列进行载体污染筛选。
无法识别序列中的外源片段可以:
导致对该序列生物学意义的错误结论
浪费时间和精力分析污染序列
延迟在公共数据库中释放序列
用受污染的序列污染公共数据库
GenBank注释人员使用VecScreen验证提交给数据库的序列是否不受载体污染。VecScreen在一个查询序列中搜索匹配UniVec中任何序列的段。UniVec是一个专用的非冗余载体数据库。该搜索使用带有预设参数的BLAST对载体污染进行最优检测。匹配载体序列的查询段将根据匹配的强度进行分类,并显示它们的位置(参见一个正结果示例)。
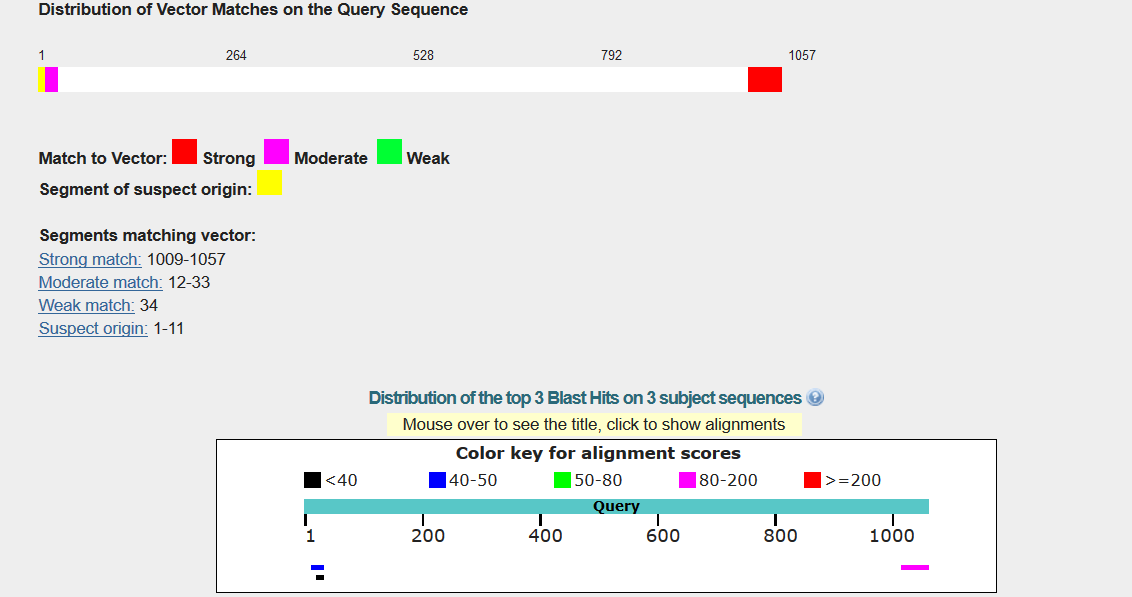
关于结果的解释https://www.ncbi.nlm.nih.gov/tools/vecscreen/interpretation/
3)VecScreen Search Parameters
理论上,任何向量污染的序列都应该与已知向量序列相同。在实践中,偶尔的差异被认为是由测序错误引起的,较少的情况下,是由工程变异或自发突变引起的。因此,选择用于VecScreen的搜索参数是为了找到与已知向量序列相同的序列段,或者只与已知序列略有偏离的序列段。
用于VecScreen的blastn参数比默认的blastn参数严格得多。主要差异有:
增加对不匹配的惩罚,这严重限制了不匹配的频率。
间隙惩罚更容忍单碱基插入或删除,这适应了添加或删除碱基的排序错误类型。
只对初始命中进行低复杂度过滤,这可以防止在低复杂度区域中启动对齐,同时允许跨区域的对齐
使用blastn选项预先设置VecScreen参数:-task blastn -reward 1 -penalty -5 -gapopen 3 -gapextend 3 -dust yes -soft_mask true - value 700 -searchsp 1750000000000
4)VecScreen Match Categories
载体污染通常发生在序列的开始或结束;因此,对终端和内部匹配使用不同的标准。如果匹配在查询序列开始的25个碱基内开始,或者在序列结束的25个碱基内停止,VecScreen将该匹配视为终端。在另一个匹配的25个碱基内开始或停止的匹配也被视为终端匹配。匹配根据随机序列之间发生的具有相同得分的比对的预期频率进行分类。
强匹配向量:(期望在1,000,000个长度为350kb的查询中有一个随机匹配。)
终端匹配,得分≥24。
内部匹配,得分≥30。
向量适度匹配:(期望在1000个长度为350 kb的查询中有一个随机匹配。)
终场比分19比23。
内部比赛得分25比29。
弱匹配向量:(期望在40个长度为350 kb的查询中有一个随机匹配。)
终场比分16比18。
内部比赛得分23比24。
可疑来源序列
Any segment of fewer than 50 bases between two vector matches or between a match and an end.
参考:
https://www.ncbi.nlm.nih.gov/tools/vecscreen/about/
https://www.ncbi.nlm.nih.gov/tools/vecscreen/univec/#Overview
https://www.ncbi.nlm.nih.gov/tools/vecscreen/contam/#Definition