遗传算法
基本原理
遗传算法(Genetic Algorithm, GA)起源于对生物系统所进行的计算机模拟研究。它是模仿自然界生物进化机制发展起来的随机全局搜索和优化方法,借鉴了达尔文的进化论和孟德尔的遗传学说。其本质是一种高效、并行、全局搜索的方法,能在搜索过程中自动获取和积累有关搜索空间的知识,并自适应地控制搜索过程以求得最佳解。
基本流程
总结出遗传算法的一般步骤:
开始循环直至找到满意的解。
1.评估每条染色体所对应个体的适应度。
2.遵照适应度越高,选择概率越大的原则,从种群中选择两个个体作为父方和母方。
3.抽取父母双方的染色体,进行交叉,产生子代。
4.对子代的染色体进行变异。
5.重复2,3,4步骤,直到新种群的产生。
结束循环。
代码实现
连点画图函数
function plot_route(a,R)
scatter(a(:,1),a(:,2),'rx');
hold on;
plot([a(R(1),1),a(R(length(R)),1)],[a(R(1),2),a(R(length(R)),2)]);
hold on;for i=2:length(R)
x0=a(R(i-1),1);
y0=a(R(i-1),2);
x1=a(R(i),1);
y1=a(R(i),2);
xx=[x0,x1];
yy=[y0,y1];
plot(xx,yy);
hold on;
end
end
染色体的路程代价函数(用于计算每个染色体的长度)
function len=myLength(D,p)%p是一个排列
[N,NN]=size(D);
len=D(p(1,N),p(1,1));
for i=1:(N-1)
len=len+D(p(1,i),p(1,i+1));
end
end
适应度函数(每次迭代都要计算每个染色体在本种群内部的优先级别,类似归一化参数。越大越好!)
function fitness=fit(len,m,maxlen,minlen)
fitness=len;
for i=1:length(len)
fitness(i,1)=(1-(len(i,1)-minlen)/(maxlen-minlen+0.0001)).^m;
end
变异函数
function a=Mutation(A)
index1=0;index2=0;
nnper=randperm(size(A,2));
index1=nnper(1);
index2=nnper(2);
%fprintf('index1=%d ',index1);
%fprintf('index2=%d ',index2);
temp=0;
temp=A(index1);
A(index1)=A(index2);
A(index2)=temp;
a=A;
end
对调函数
function [x,y]=exchange(x,y)
temp=x;
x=y;
y=temp;
end
交叉操作函数
function [A,B]=cross(A,B)
L=length(A);
if L<10
W=L;
elseif ((L/10)-floor(L/10))>=rand&&L>10
W=ceil(L/10)+8;
else
W=floor(L/10)+8;
end
%%W为需要交叉的位数
p=unidrnd(L-W+1);%随机产生一个交叉位置
%fprintf('p=%d ',p);%交叉位置
for i=1:W
x=find(A==B(1,p+i-1));
y=find(B==A(1,p+i-1));
[A(1,p+i-1),B(1,p+i-1)]=exchange(A(1,p+i-1),B(1,p+i-1));
[A(1,x),B(1,y)]=exchange(A(1,x),B(1,y));
end
end
主函数(main函数)
%main
clear;
clc;
%%%%%%%%%%%%%%%输入参数%%%%%%%%
N=25; %%城市的个数
M=100; %%种群的个数
ITER=2000; %%迭代次数
%C_old=C;m=2; %%适应值归一化淘汰加速指数
Pc=0.8; %%交叉概率
Pmutation=0.05; %%变异概率
%%生成城市的坐标pos=randn(N,2);
%%生成城市之间距离矩阵
D=zeros(N,N);
for i=1:N
for j=i+1:N
dis=(pos(i,1)-pos(j,1)).^2+(pos(i,2)-pos(j,2)).^2;
D(i,j)=dis^(0.5);
D(j,i)=D(i,j);
endend %%生成初始群体
popm=zeros(M,N);
for i=1:M
popm(i,:)=randperm(N);%随机排列,比如[2 4 5 6 1 3]
end
%%随机选择一个种群
R=popm(1,:);
figure(1);
scatter(pos(:,1),pos(:,2),'rx');%画出所有城市坐标
axis([-3 3 -3 3]);figure(2);plot_route(pos,R); %%画出初始种群对应各城市之间的连线a
xis([-3 3 -3 3]);%%初始化种群及其适应函数
fitness=zeros(M,1);
len=zeros(M,1);
for i=1:M%计算每个染色体对应的总长度
len(i,1)=myLength(D,popm(i,:));
end
maxlen=max(len);%最大回路
minlen=min(len);%最小回路
fitness=fit(len,m,maxlen,minlen);
rr=find(len==minlen);%找到最小值的下标,赋值为rr
R=popm(rr(1,1),:);%提取该染色体,赋值为R
for i=1:N fprintf('%d ',R(i));%把R顺序打印出来
end
fprintf('\n');
fitness=fitness/sum(fitness);
distance_min=zeros(ITER+1,1); %%各次迭代的最小的种群的路径总长
nn=M;
iter=0;
while iter<=ITER
fprintf('迭代第%d次\n',iter); %%选择操作
p=fitness./sum(fitness);
q=cumsum(p);%累加
for i=1:(M-1)
len_1(i,1)=myLength(D,popm(i,:));
r=rand;
tmp=find(r<=q);
popm_sel(i,:)=popm(tmp(1),:);
end
[fmax,indmax]=max(fitness);%求当代最佳个体
popm_sel(M,:)=popm(indmax,:); %%交叉操作
nnper=randperm(M);
% A=popm_sel(nnper(1),:);
% B=popm_sel(nnper(2),:);
%% for i=1:M*Pc*0.5
A=popm_sel(nnper(i),:);
B=popm_sel(nnper(i+1),:);
[A,B]=cross(A,B);
% popm_sel(nnper(1),:)=A;
% popm_sel(nnper(2),:)=B;
popm_sel(nnper(i),:)=A;
popm_sel(nnper(i+1),:)=B;
end
%%变异操作
for i=1:M
pick=rand;
while pick==0
pick=rand;
end
if pick<=Pmutation
popm_sel(i,:)=Mutation(popm_sel(i,:));
end
end
%%求适应度函数
NN=size(popm_sel,1);
len=zeros(NN,1);
for i=1:NN
len(i,1)=myLength(D,popm_sel(i,:));
end
maxlen=max(len);
minlen=min(len);
distance_min(iter+1,1)=minlen;
fitness=fit(len,m,maxlen,minlen);
rr=find(len==minlen);
fprintf('minlen=%d\n',minlen);
R=popm_sel(rr(1,1),:);
for i=1:N
fprintf('%d ',R(i));
end
fprintf('\n');
popm=[];
popm=popm_sel;
iter=iter+1;
%pause(1);
end
%end of while figure(3)plot_route(pos,R);
axis([-3 3 -3 3]);
figure(4)plot(distance_min);
结果分析
一、种群个数M对算法的影响:
参数ITER=2000,m=2,Pc=0.8,Pm=0.05保持不变,调整种群个数M的值,运行结果如下:
M=50
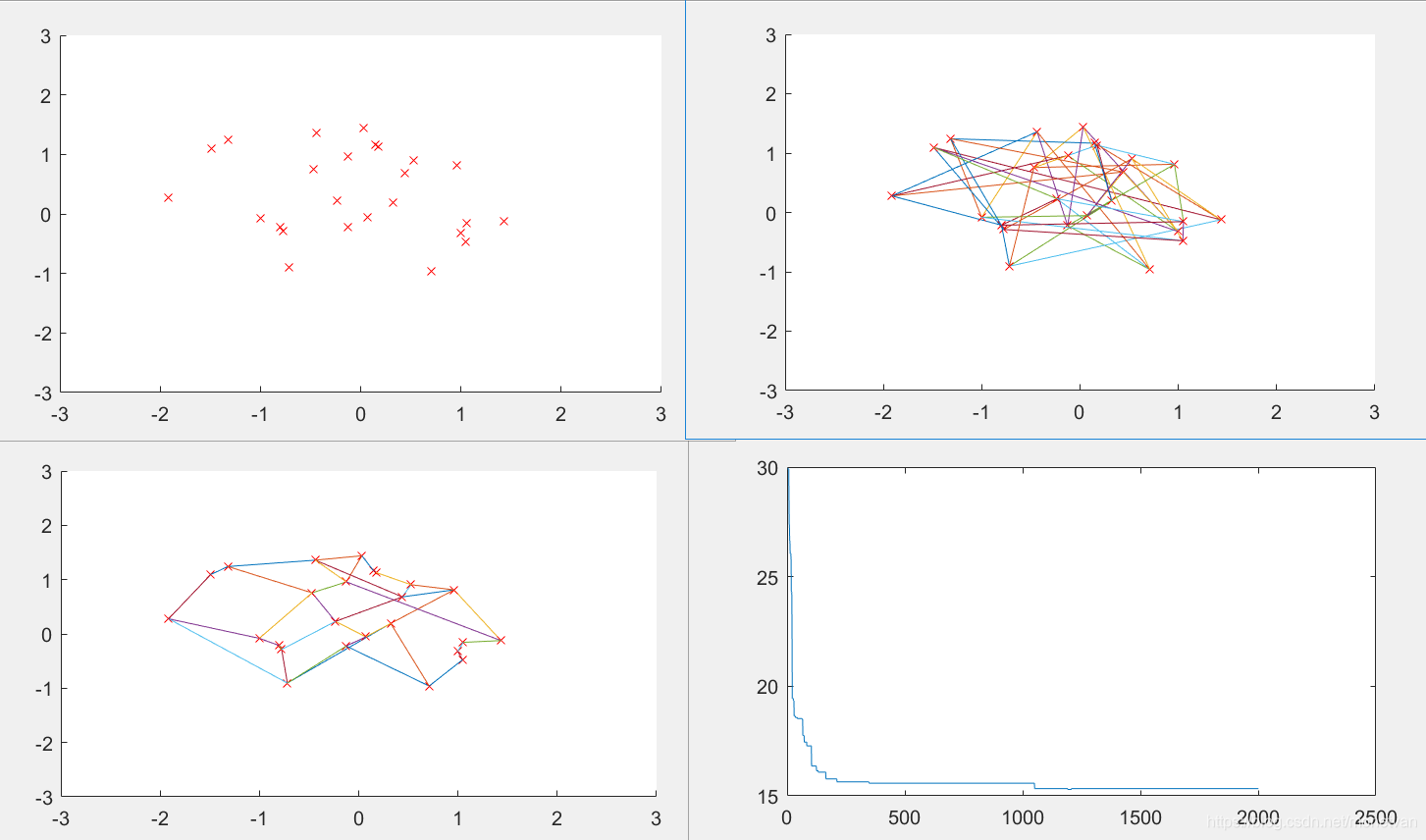
M=100
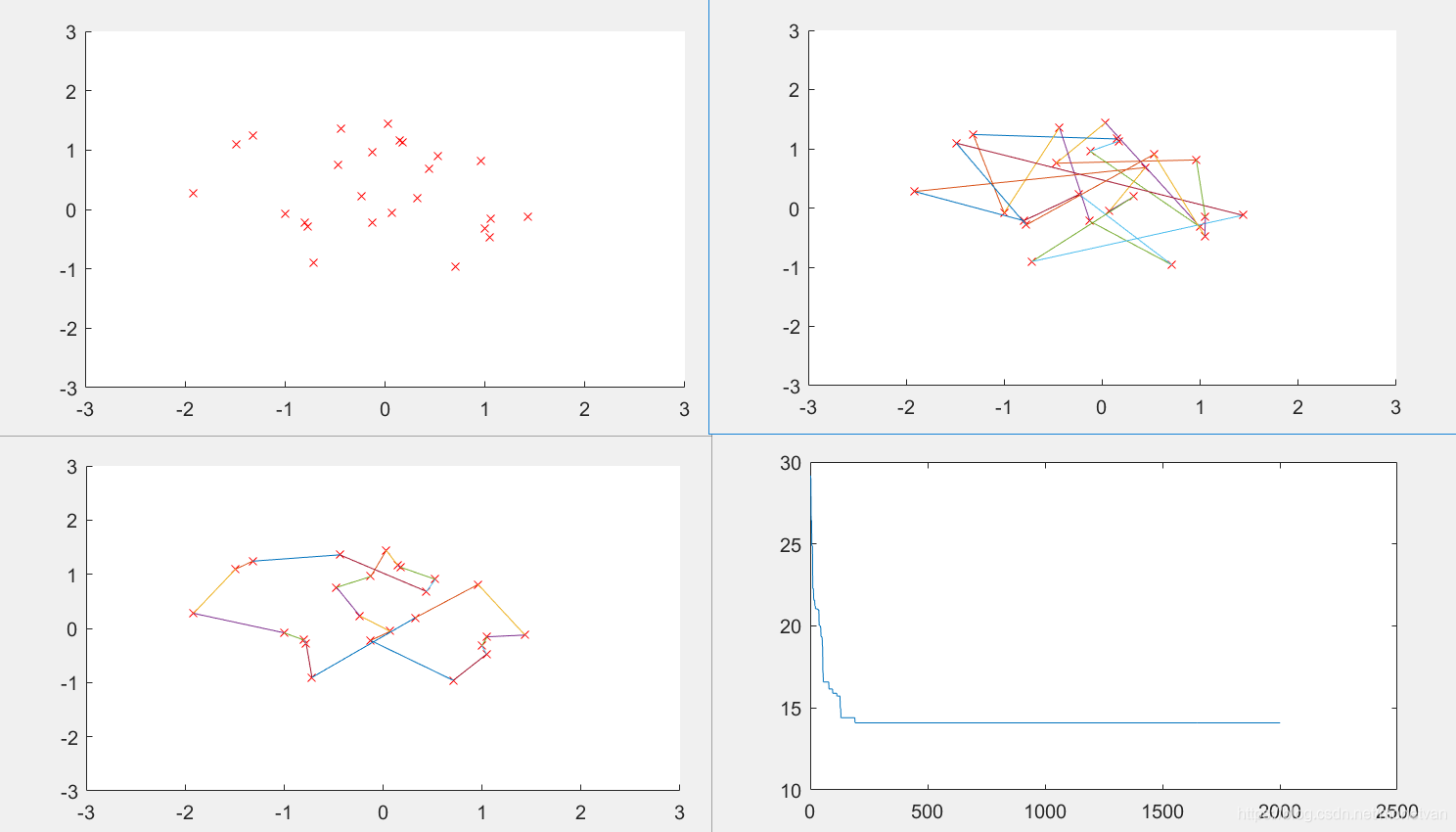
M=200
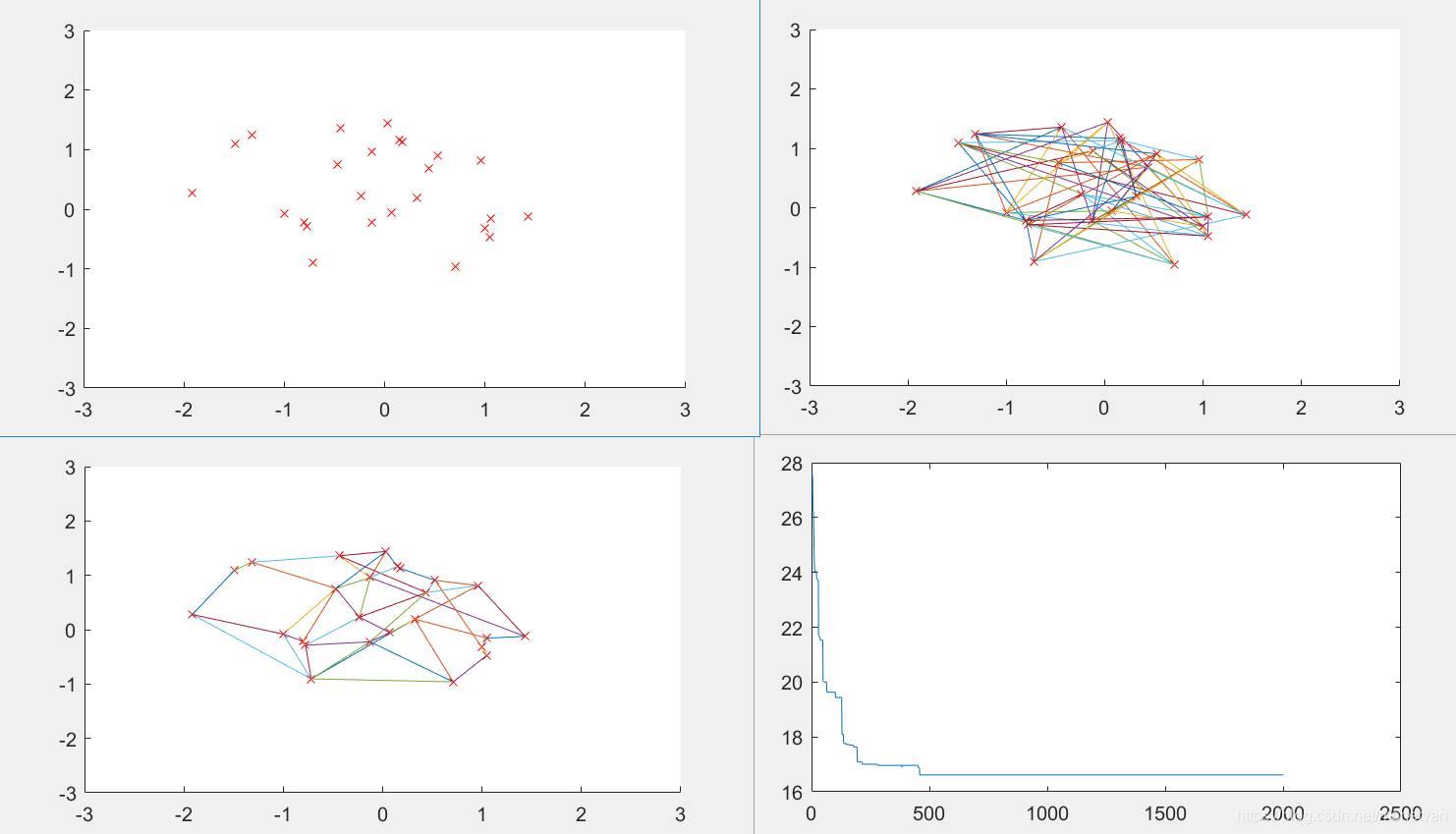
M=400
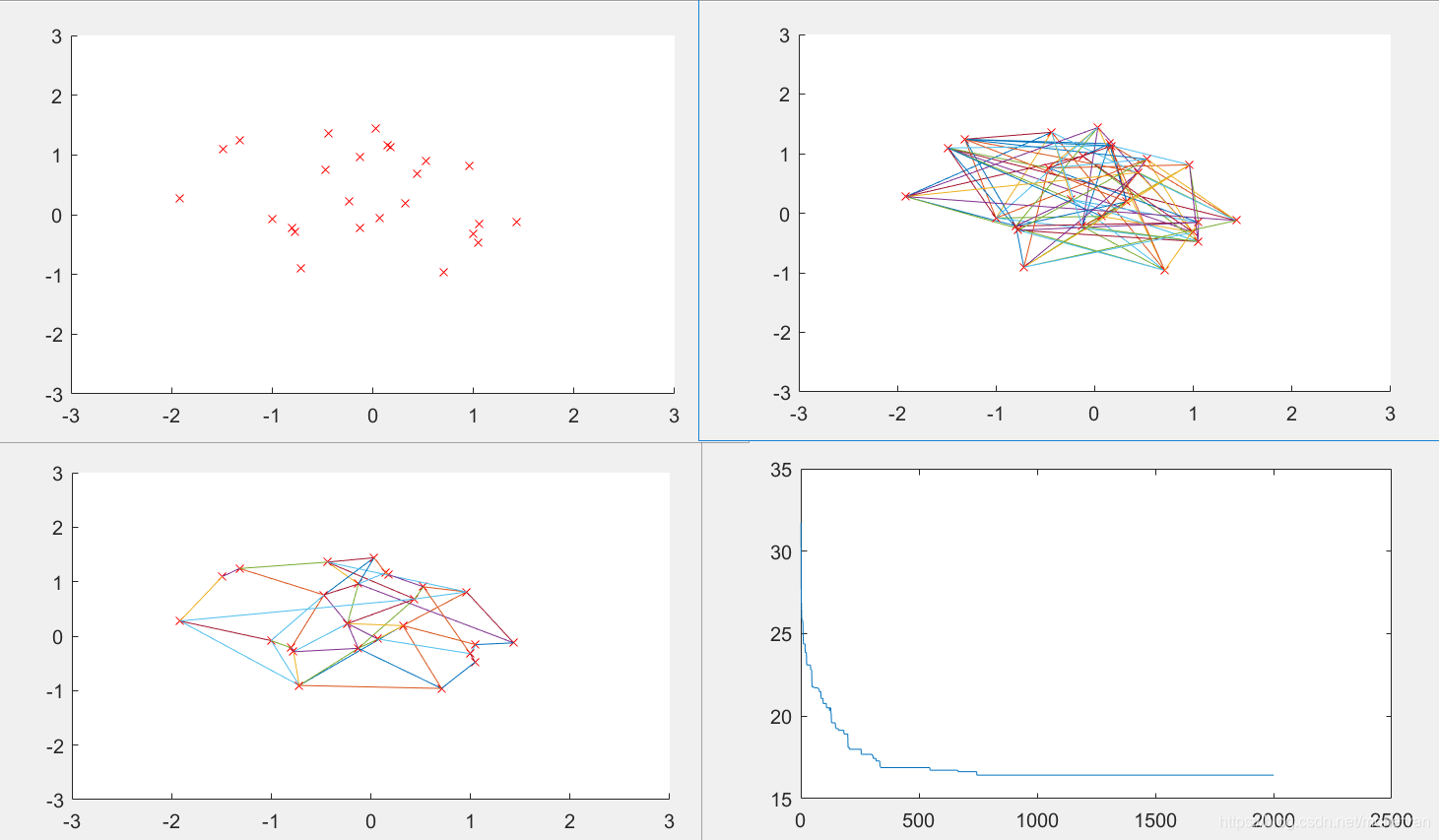 群体个数M参数设置的时候不宜过大也不宜过小。当固定城市坐标,改变种群数量,种群数量过小时,输出的最短路径较长,迭代得到的最短路径次数大,说明算法搜索能力不够,效率较低,且较难找到实际最短路径;种群数量过大时,运行时间较长,迭代达到最优次数逐渐下降,最短路径收敛趋势加快,易陷入局部最优,过早得到最短路径。
群体个数M参数设置的时候不宜过大也不宜过小。当固定城市坐标,改变种群数量,种群数量过小时,输出的最短路径较长,迭代得到的最短路径次数大,说明算法搜索能力不够,效率较低,且较难找到实际最短路径;种群数量过大时,运行时间较长,迭代达到最优次数逐渐下降,最短路径收敛趋势加快,易陷入局部最优,过早得到最短路径。
二、迭代次数ITER对算法的影响:
参数m=2,M=100,Pc=0.8,Pm=0.05保持不变,调整迭代次数ITER的值,运行结果如下:
ITER=500
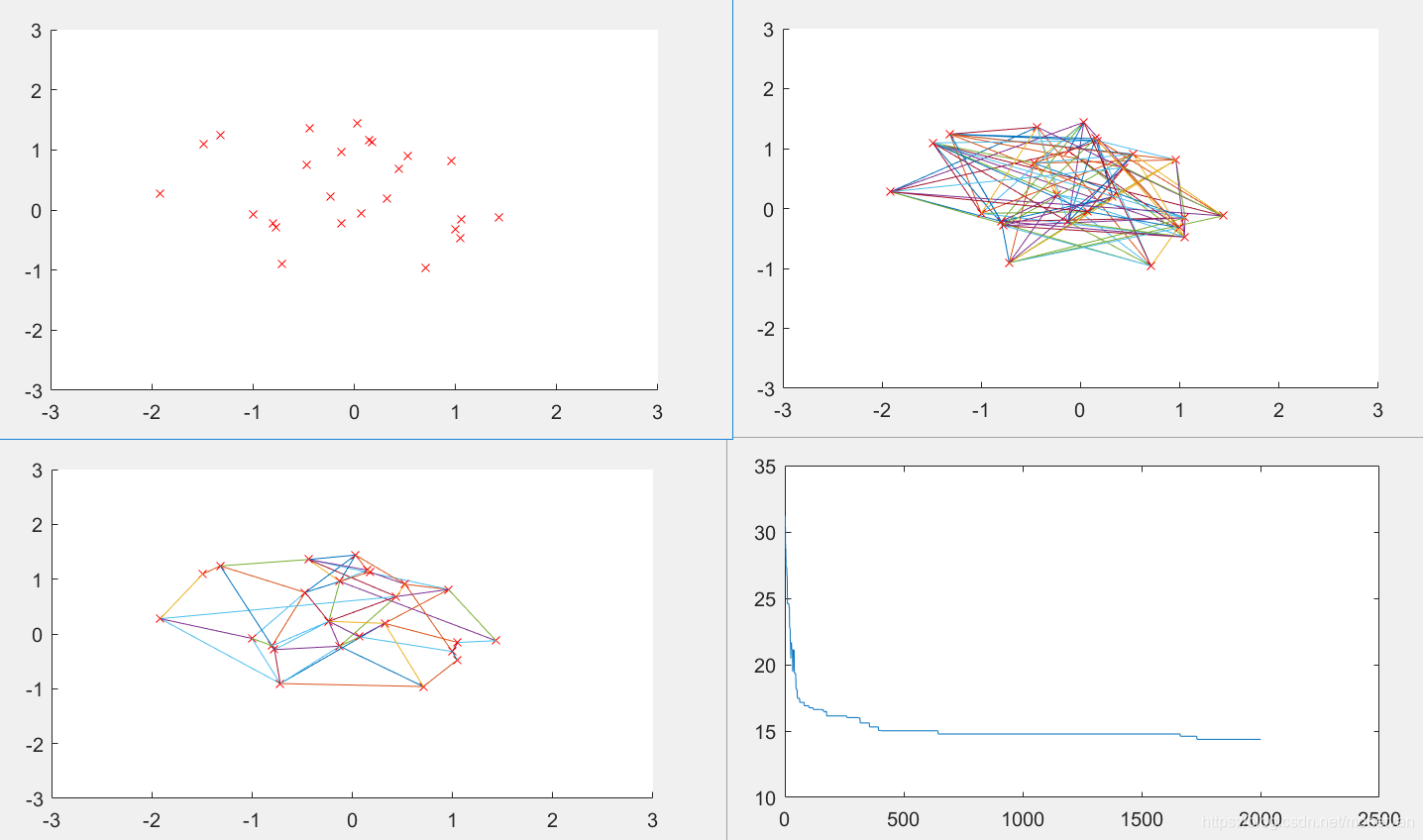
ITER=1000
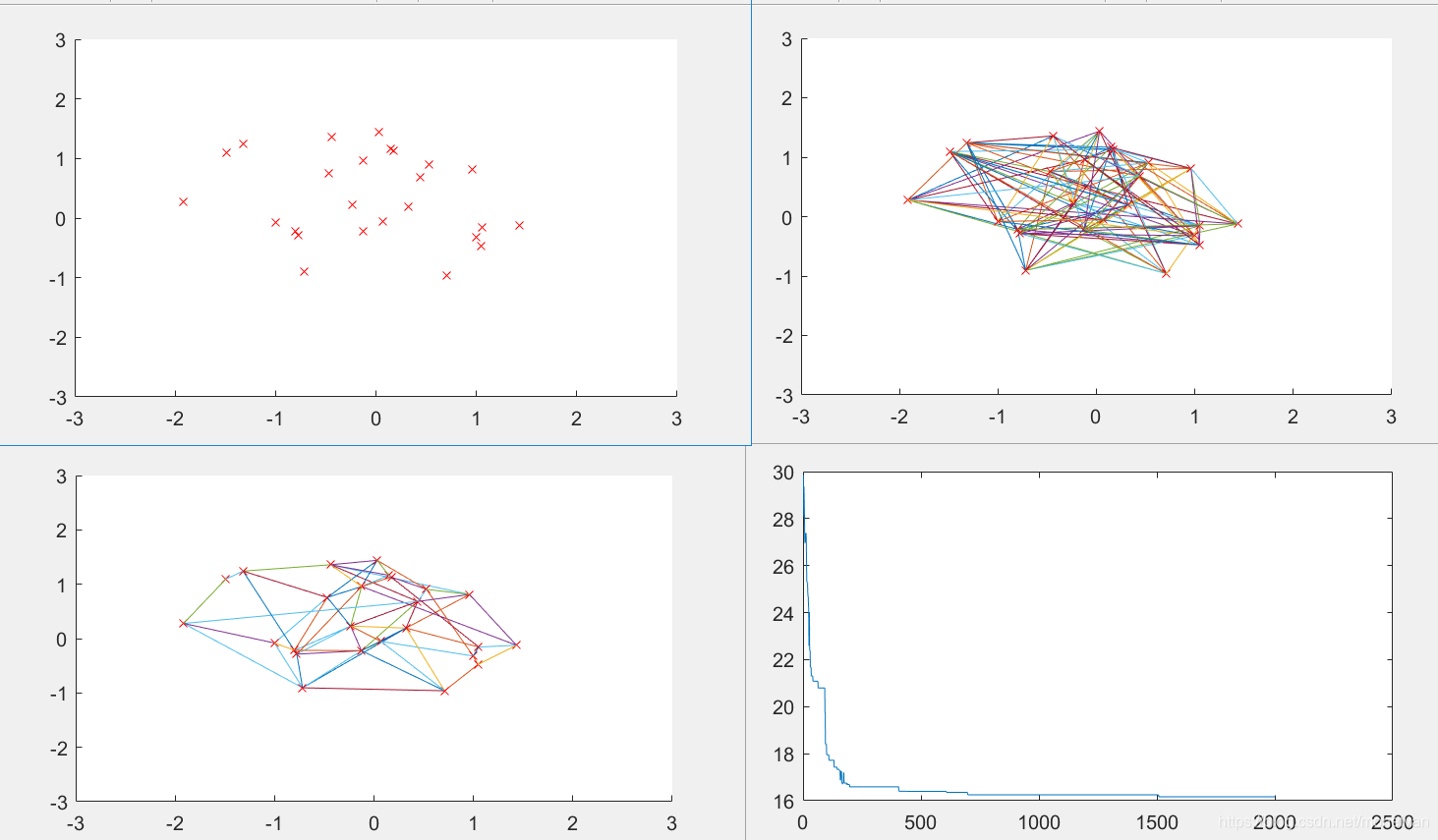
ITER=2000
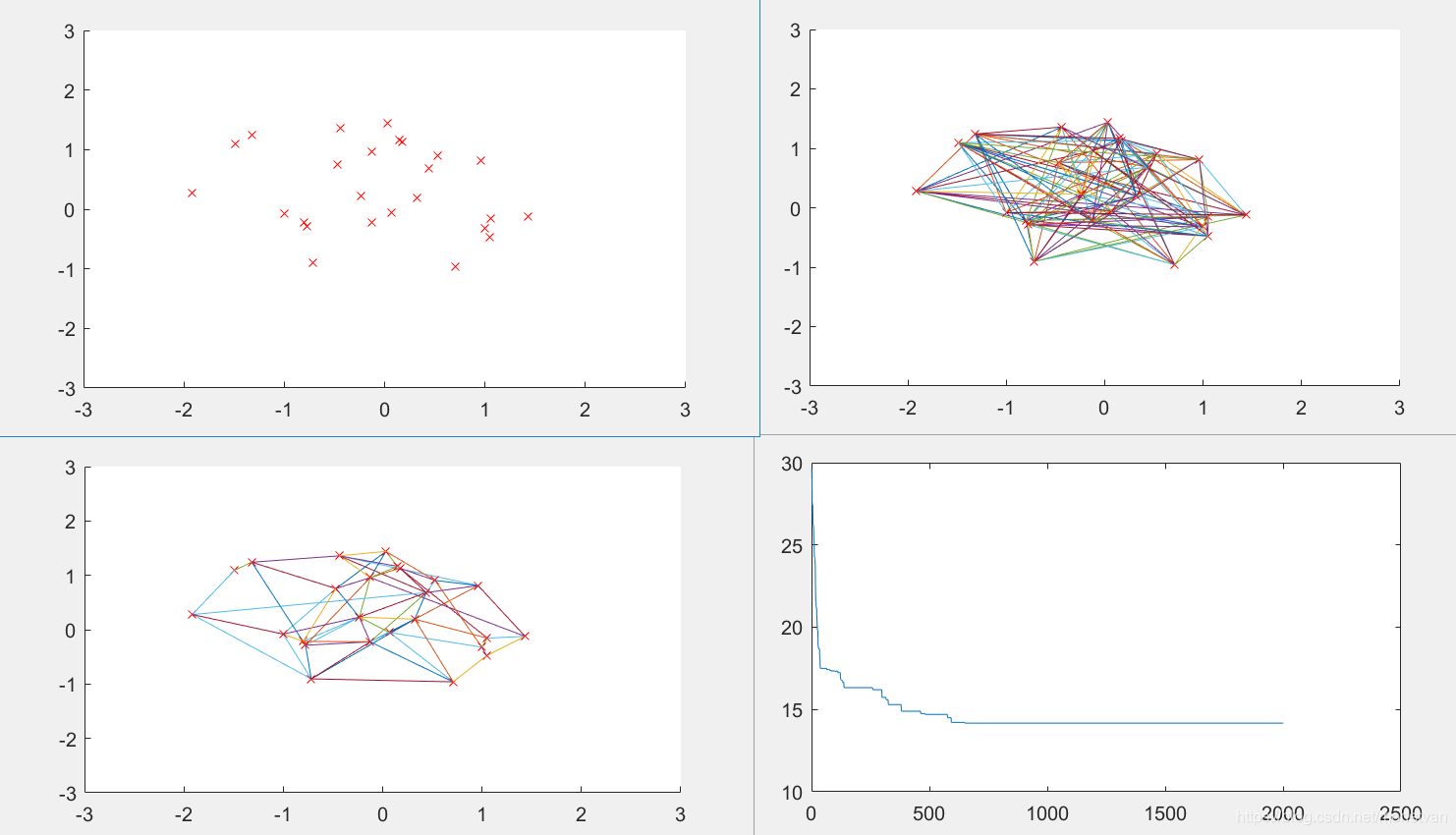
ITER=4000
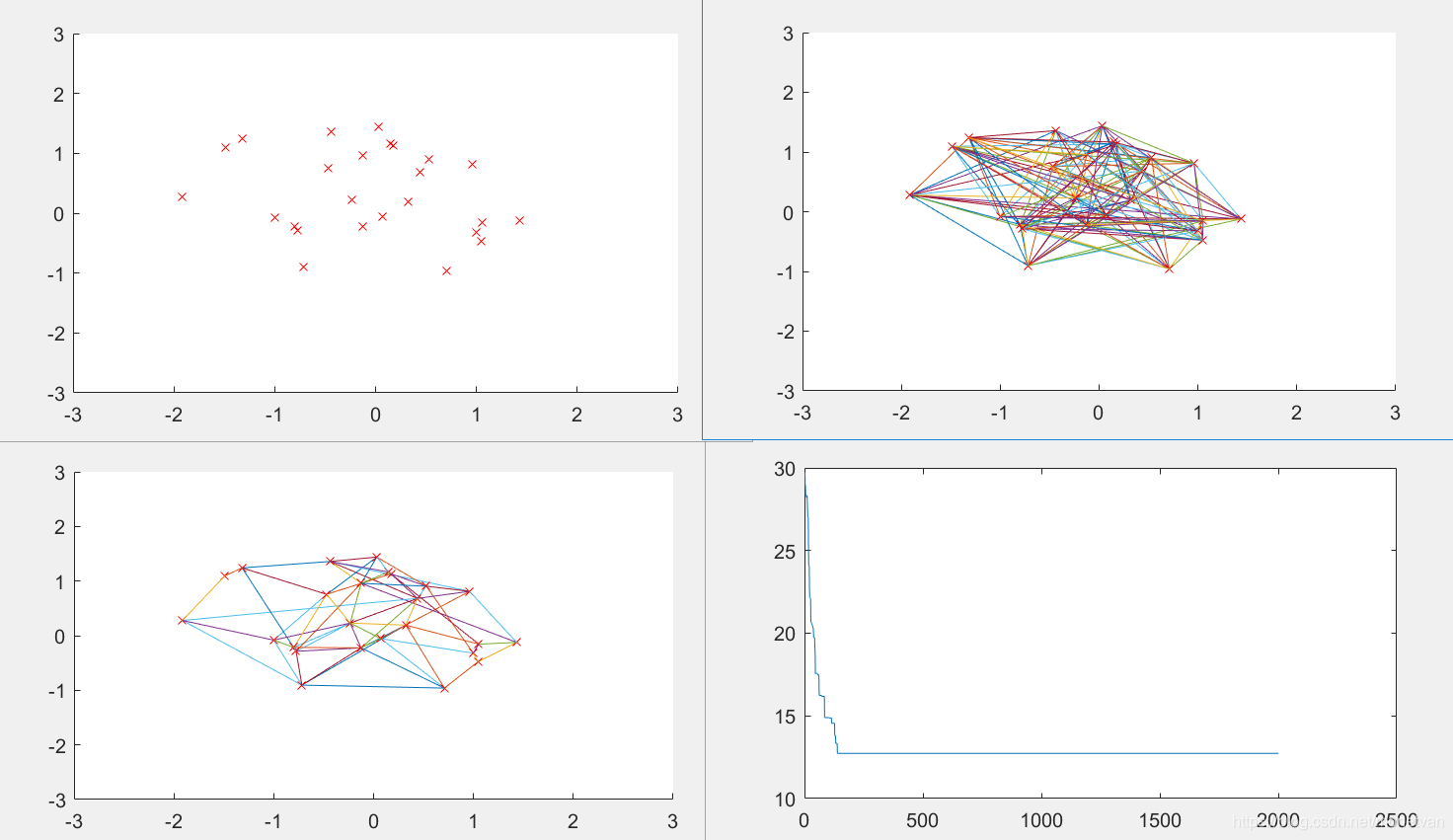 迭代次数过小时,难以收敛到最优、最短路径曲线多波动。迭代次数增大时,收敛速度曲线加快,曲线波动变小。当迭代次数过大时,会降低算法的运行效率,最短路径过早收敛,难以得到全局最短路径。
迭代次数过小时,难以收敛到最优、最短路径曲线多波动。迭代次数增大时,收敛速度曲线加快,曲线波动变小。当迭代次数过大时,会降低算法的运行效率,最短路径过早收敛,难以得到全局最短路径。
三、交叉概率Pc对算法的影响:
参数m=2,M=100,ITER=2000,Pm=0.05保持不变,调整交叉概率Pc的值,运行结果如下:
Pc=0.1
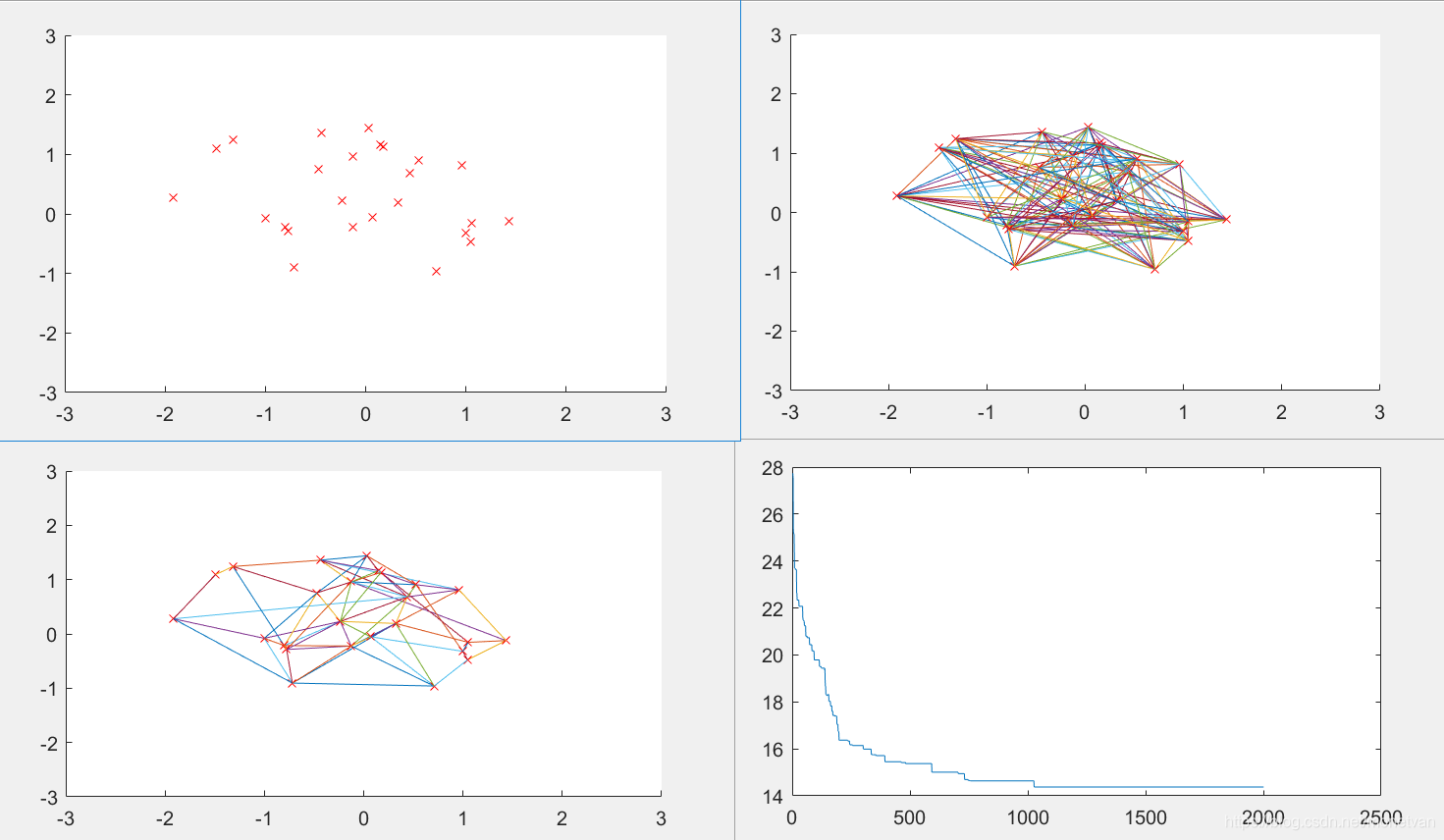
Pc=0.2
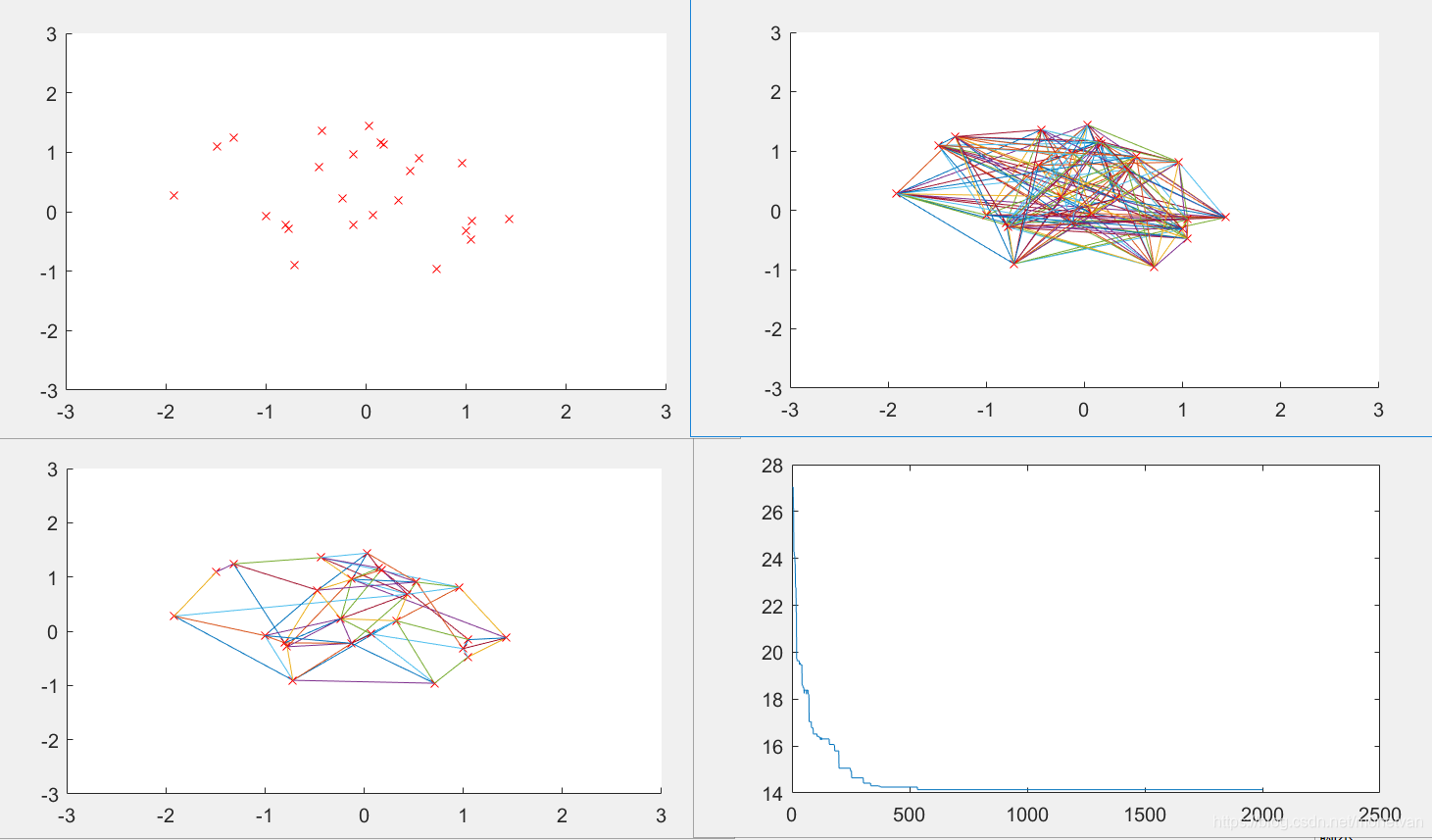
Pc=0.5
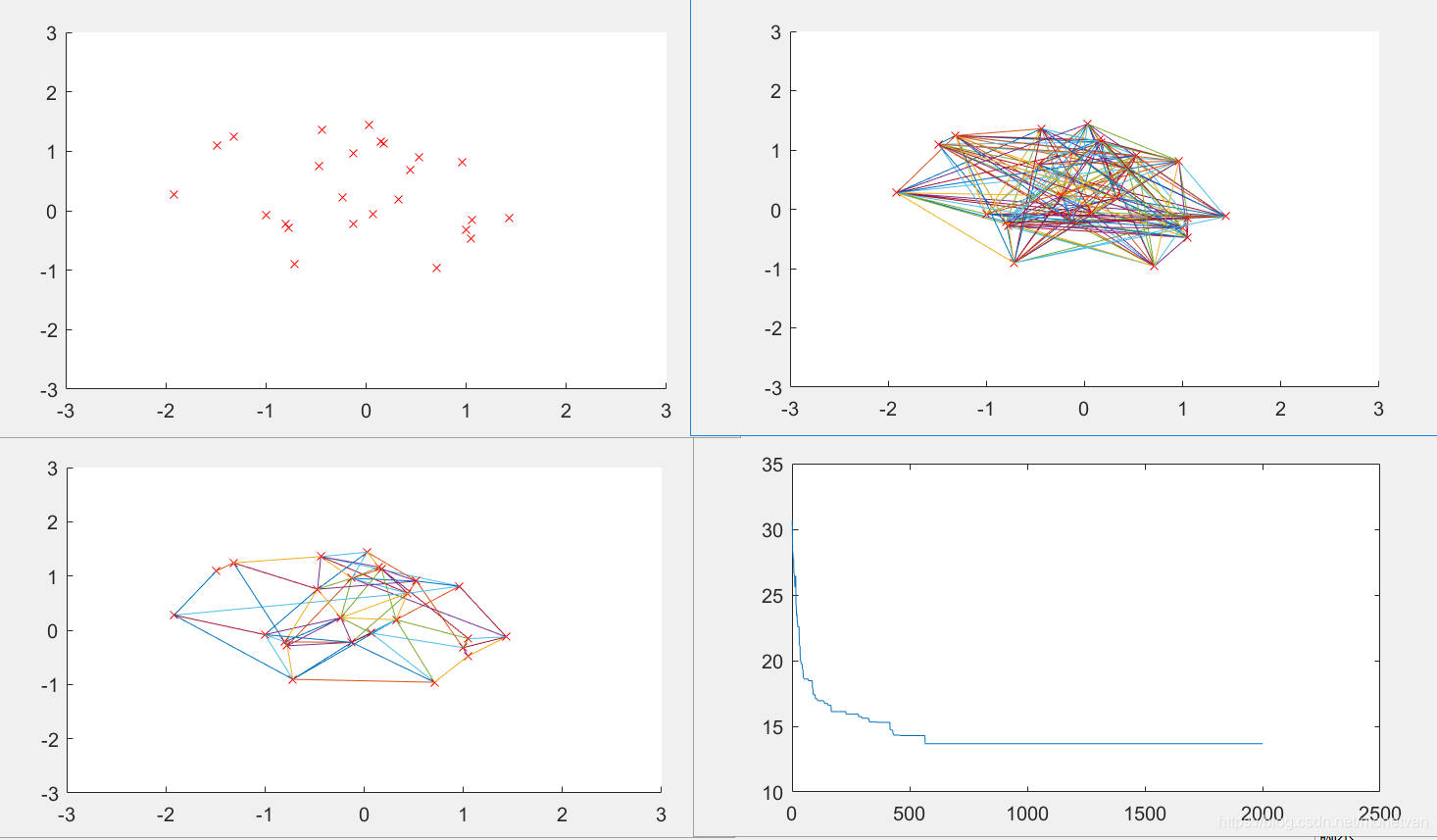
Pc=0.8
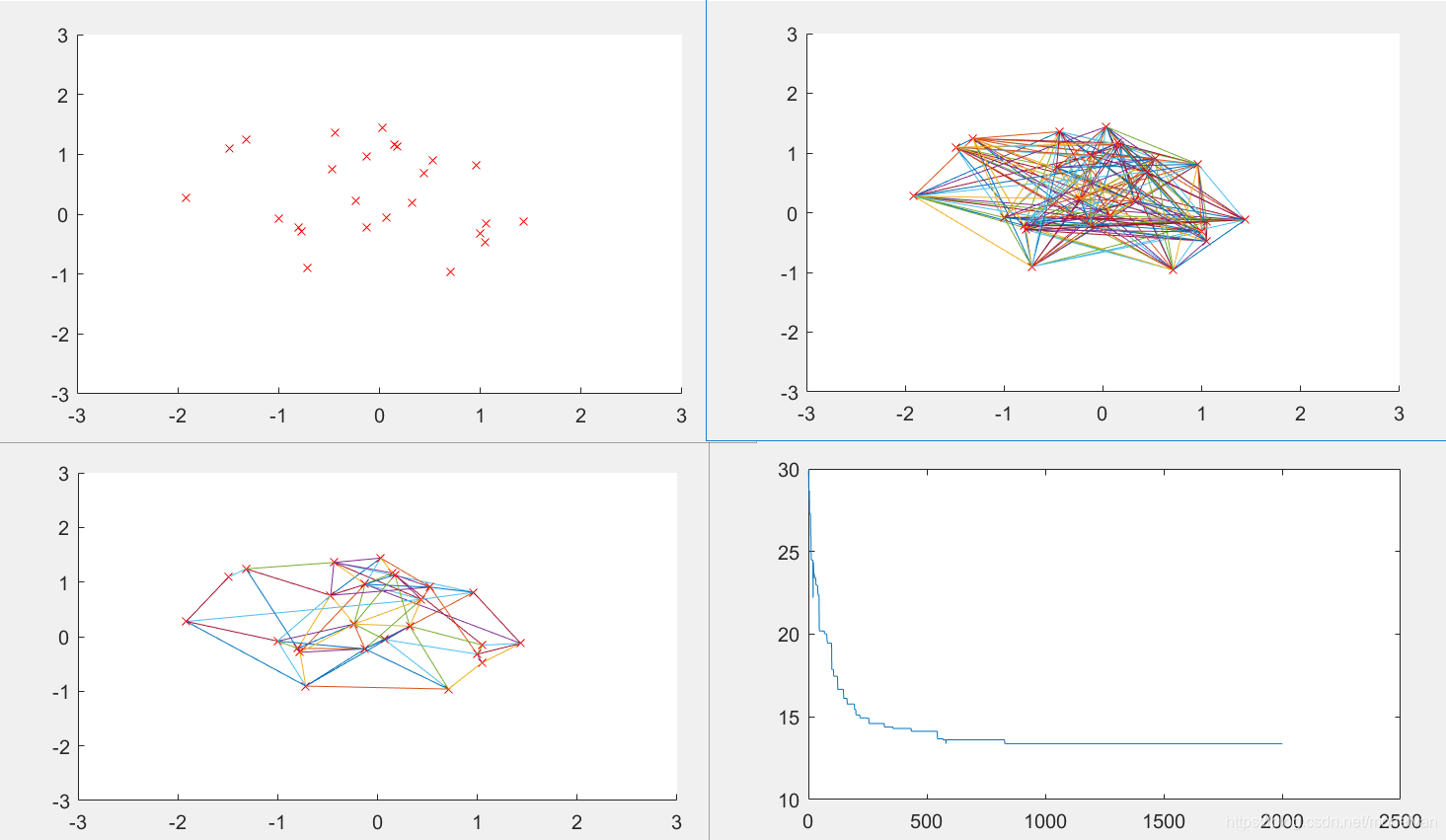 交叉概率决定了进化过程种群参加交配的染色体平均数目,过大或过小都难以得到最短路径最短路径。交叉概率在0.4-0.8时较合适,当交叉概率过小时最短路径收敛较慢,且陷入局部最优解。过大时难以收敛得到最优解。
交叉概率决定了进化过程种群参加交配的染色体平均数目,过大或过小都难以得到最短路径最短路径。交叉概率在0.4-0.8时较合适,当交叉概率过小时最短路径收敛较慢,且陷入局部最优解。过大时难以收敛得到最优解。
四、变异概率Pm对算法的影响:
参数m=2,M=100,ITER=2000,Pc=0.8保持不变,调整变异概率Pm的值,运行结果如下:
Pm=0.01
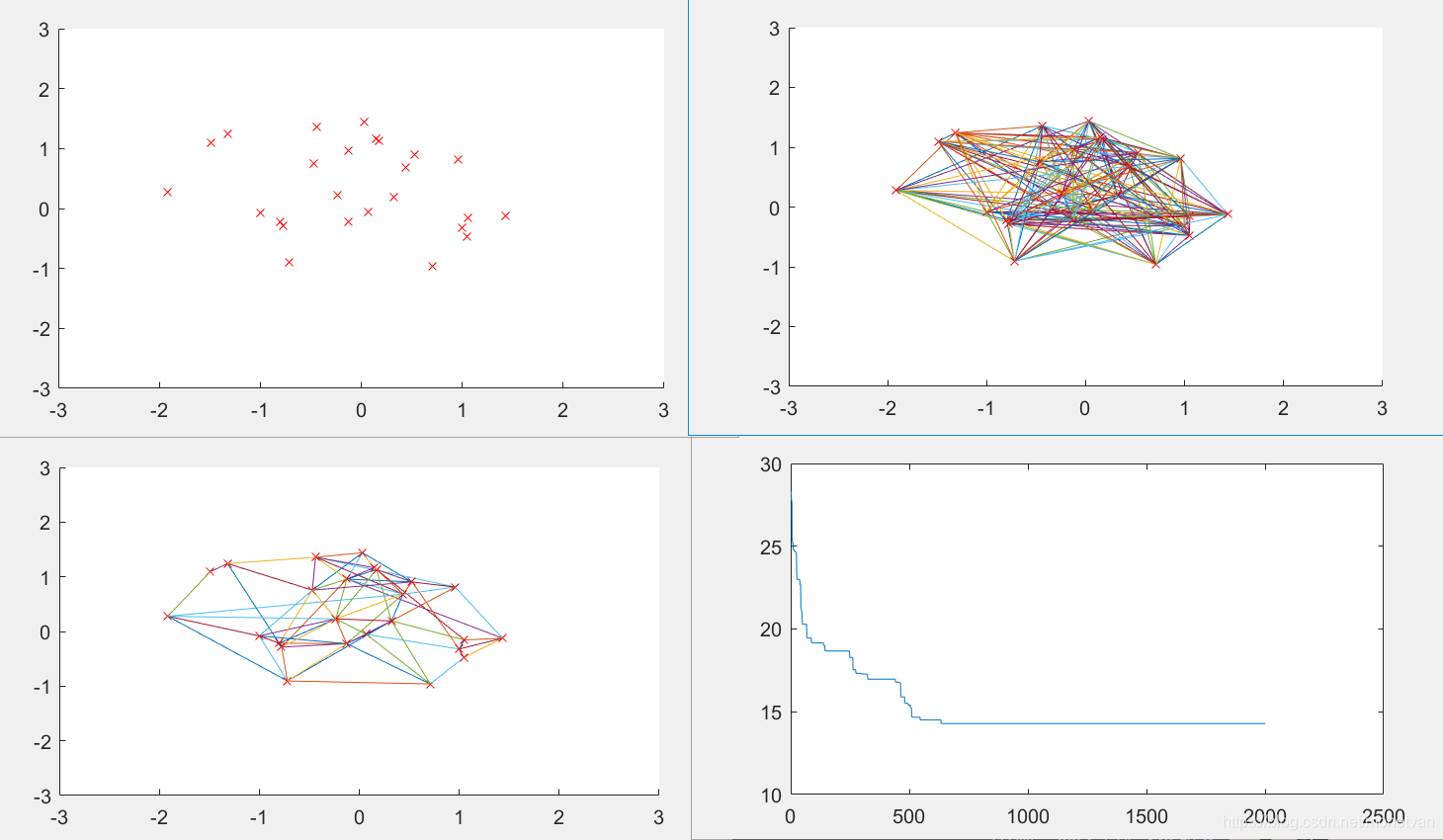
Pm=0.05
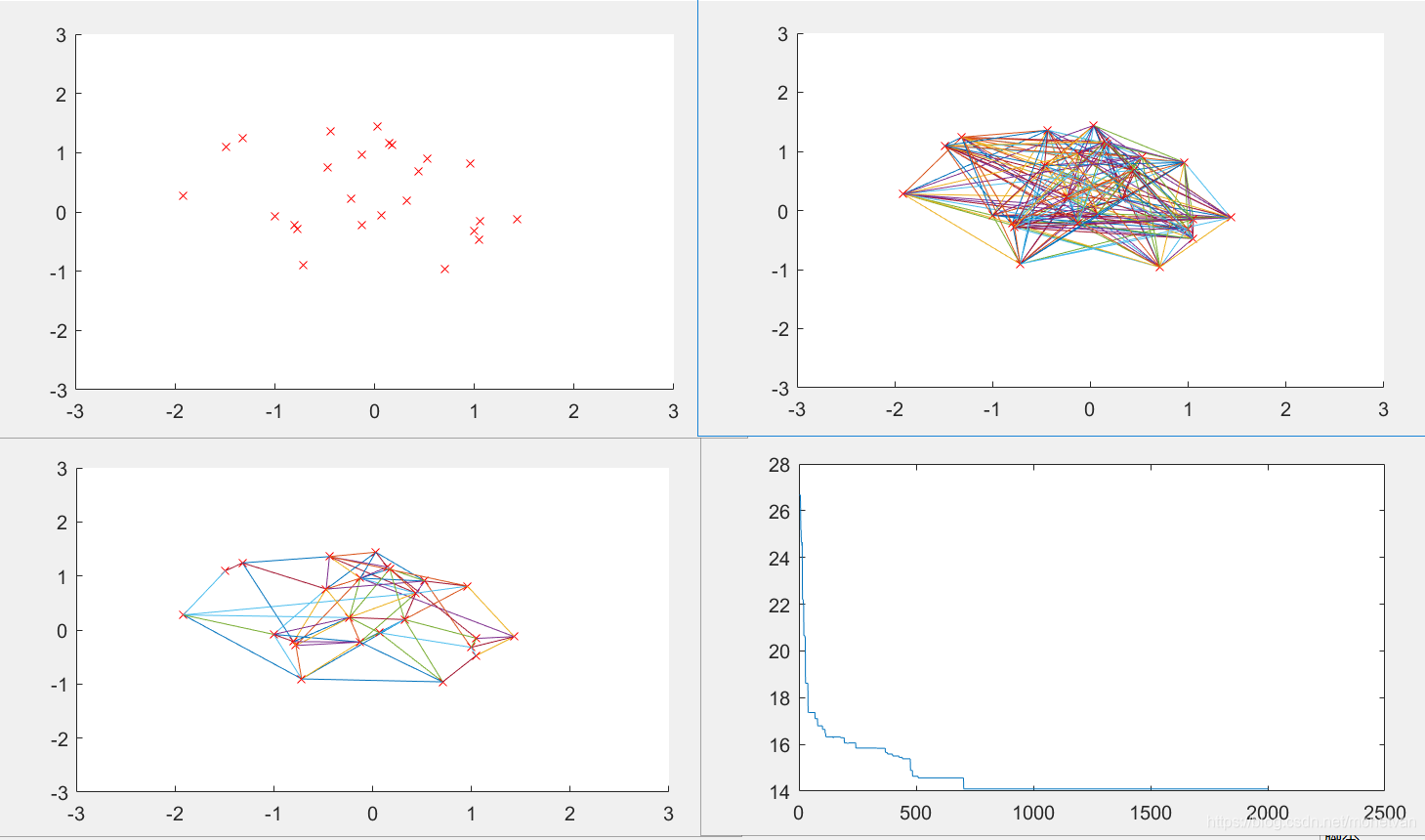
Pm=0.1
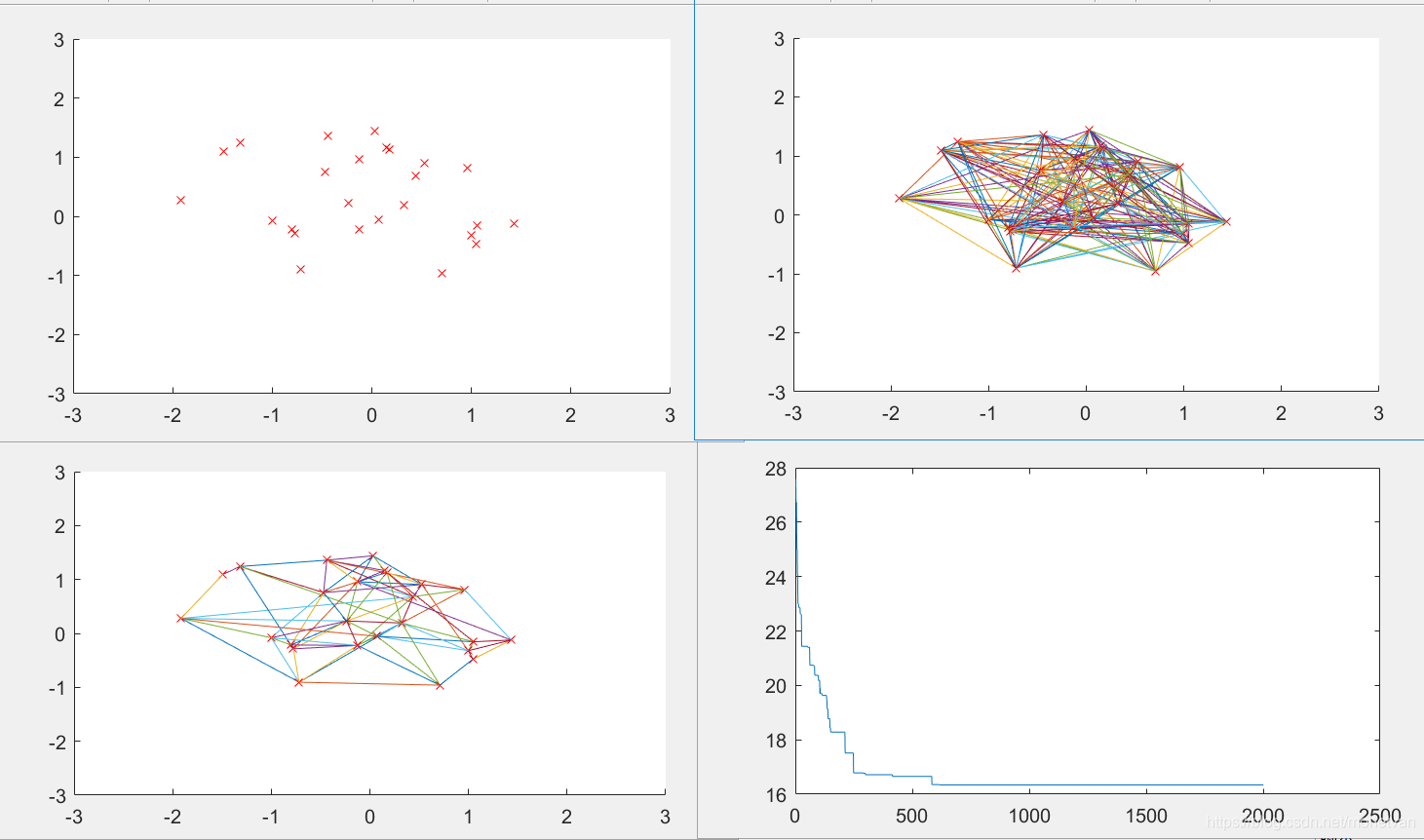
Pm=0.2
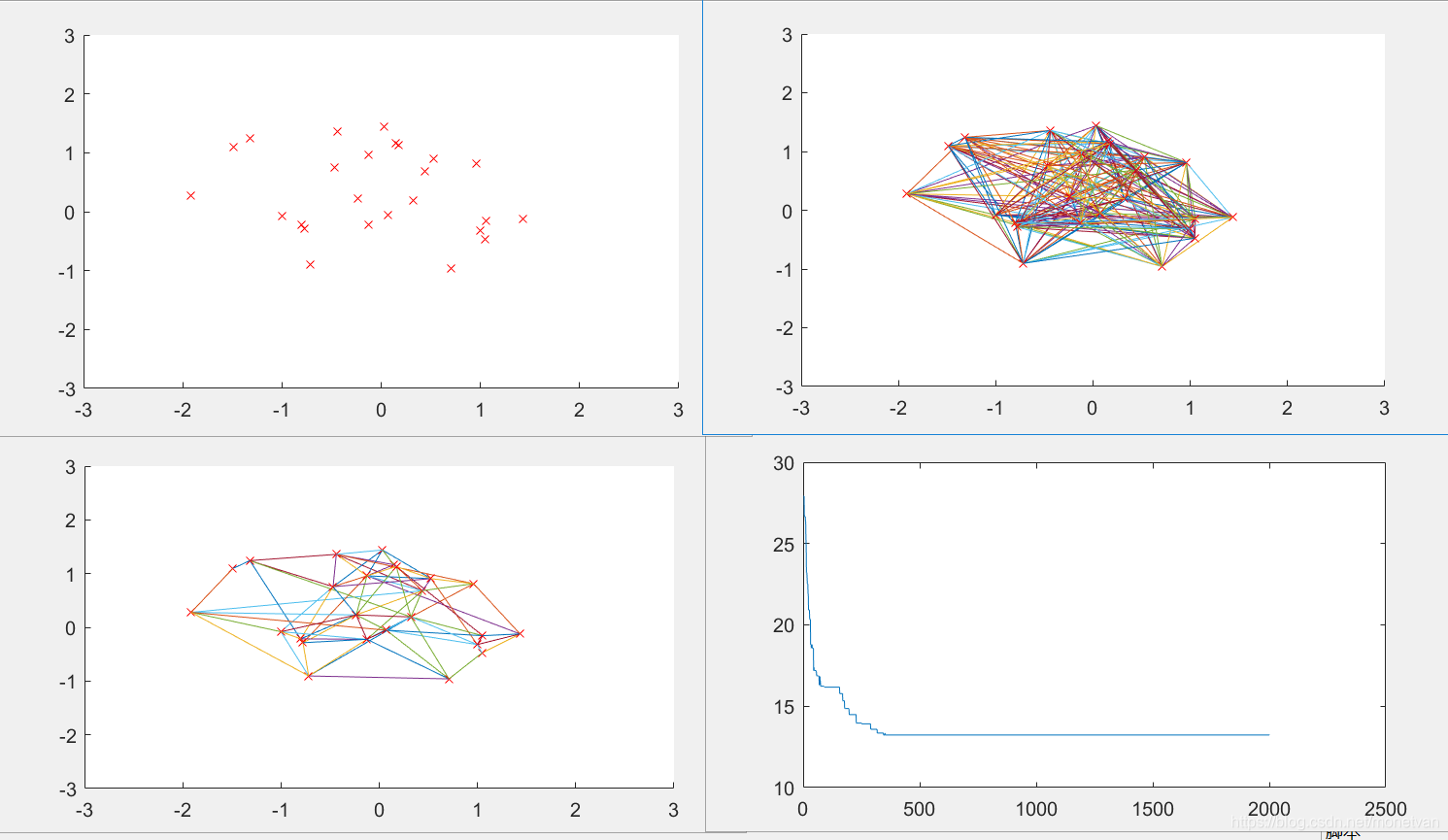
Pm值不宜过大,因为变异对已找到最优解具有一定的破坏作用,如果Pm的值太大,可能会导致算法目前所处的较好的搜索状况倒退会原本较差的情况。变异概率越小,收敛速度越慢;变异概率越大,基因变异较多,难以得到最短路径。
