文章目录
1、TSP问题
假设有一个旅行商人要拜访N个城市,要求他从一个城市出发,每个城市最多拜访一次,最后要回到出发的城市,保证所选择的路径长度最短。
2、算法描述
2.1、算法简介
遗传算法(GeneticAlgorithm)是模拟达尔文生物进化论的自然选择和遗传学机理的生物进化过程的计算模型,通过模拟自然进化过程搜索最优解。遗传算法是从代表问题可能潜在的解集的一个种群(population)开始的,初代种群产生之后,按照适者生存和优胜劣汰的原理,逐代(generation)演化产生出越来越好的近似解,在每一代,根据问题域中个体的适应度(fitness)大小选择个体,并借助于自然遗传学的遗传算子(genetic operators)进行组合交叉(crossover)和变异(mutation),产生出代表新的解集的种群。这个过程将导致种群像自然进化一样的后生代种群比前代更加适应于环境,末代种群中的最优个体经过解码(decoding),可以作为问题近似最优解。
2.2、遗传算子
遗传算法中有选择算子、交叉算子和变异算子。
选择算子用于在父代种群中选择进入下一代的个体。
交叉算子用于对种群中的个体两两进行交叉,有Partial-Mapped Crossover、Order Crossover、Position-based Crossover等交叉算子。
变异算子用于对种群中的个体进行突变。
2.3、算法步骤描述
1、计算开始时,随机初始化一定数目的个体,并计算每个个体的适应度值,产生第一代(初始种群)。
2、如果不满足优化准则,开始新一代的计算:按照适应度值选择个体,产生下一代;父代按一定概率进行交叉操作,产生子代;所有的子代按一定概率变异,形成新的一代。计算新子代的适应度值。
3、这一过程循环执行,直到满足优化准则为止。
3、代码
main
clear;
clc;
%%%%%%%%%%%%%%%输入参数%%%%%%%%
N=25; %%城市的个数
M=100; %%种群的个数
ITER=2000; %%迭代次数
%C_old=C;
m=2; %%适应值归一化淘汰加速指数
Pc=0.8; %%交叉概率
Pmutation=0.05; %%变异概率
%%生成城市的坐标
pos=randn(N,2);
%%生成城市之间距离矩阵
D=zeros(N,N);
for i=1:N
for j=i+1:N
dis=(pos(i,1)-pos(j,1)).^2+(pos(i,2)-pos(j,2)).^2;
D(i,j)=dis^(0.5);
D(j,i)=D(i,j);
end
end
%%生成初始群体
popm=zeros(M,N);
for i=1:M
popm(i,:)=randperm(N);%随机排列,比如[2 4 5 6 1 3]
end
%%随机选择一个种群
R=popm(1,:);
figure(1);
scatter(pos(:,1),pos(:,2),'rx');%画出所有城市坐标
axis([-3 3 -3 3]);
figure(2);
plot_route(pos,R); %%画出初始种群对应各城市之间的连线
axis([-3 3 -3 3]);
%%初始化种群及其适应函数
fitness=zeros(M,1);
len=zeros(M,1);
for i=1:M%计算每个染色体对应的总长度
len(i,1)=myLength(D,popm(i,:));
end
maxlen=max(len);%最大回路
minlen=min(len);%最小回路
fitness=fit(len,m,maxlen,minlen);
rr=find(len==minlen);%找到最小值的下标,赋值为rr
R=popm(rr(1,1),:);%提取该染色体,赋值为R
for i=1:N
fprintf('%d ',R(i));%把R顺序打印出来
end
fprintf('\n');
fitness=fitness/sum(fitness);
distance_min=zeros(ITER+1,1); %%各次迭代的最小的种群的路径总长
nn=M;
iter=0;
while iter<=ITER
fprintf('迭代第%d次\n',iter);
%%选择操作
p=fitness./sum(fitness);
q=cumsum(p);%累加
for i=1:(M-1)
len_1(i,1)=myLength(D,popm(i,:));
r=rand;
tmp=find(r<=q);
popm_sel(i,:)=popm(tmp(1),:);
end
[fmax,indmax]=max(fitness);%求当代最佳个体
popm_sel(M,:)=popm(indmax,:);
%%交叉操作
nnper=randperm(M);
% A=popm_sel(nnper(1),:);
% B=popm_sel(nnper(2),:);
%%
for i=1:M*Pc*0.5
A=popm_sel(nnper(i),:);
B=popm_sel(nnper(i+1),:);
[A,B]=cross(A,B);
% popm_sel(nnper(1),:)=A;
% popm_sel(nnper(2),:)=B;
popm_sel(nnper(i),:)=A;
popm_sel(nnper(i+1),:)=B;
end
%%变异操作
for i=1:M
pick=rand;
while pick==0
pick=rand;
end
if pick<=Pmutation
popm_sel(i,:)=Mutation(popm_sel(i,:));
end
end
%%求适应度函数
NN=size(popm_sel,1);
len=zeros(NN,1);
for i=1:NN
len(i,1)=myLength(D,popm_sel(i,:));
end
maxlen=max(len);
minlen=min(len);
distance_min(iter+1,1)=minlen;
fitness=fit(len,m,maxlen,minlen);
rr=find(len==minlen);
fprintf('minlen=%d\n',minlen);
R=popm_sel(rr(1,1),:);
for i=1:N
fprintf('%d ',R(i));
end
fprintf('\n');
popm=[];
popm=popm_sel;
iter=iter+1;
%pause(1);
end
%end of while
figure(3)
plot_route(pos,R);
axis([-3 3 -3 3]);
figure(4)
plot(distance_min);
%交叉操作函数 cross.m
function [A,B]=cross(A,B)
L=length(A);
if L<10
W=L;
elseif ((L/10)-floor(L/10))>=rand&&L>10
W=ceil(L/10)+8;
else
W=floor(L/10)+8;
end
%%W为需要交叉的位数
p=unidrnd(L-W+1);%随机产生一个交叉位置
%fprintf('p=%d ',p);%交叉位置
for i=1:W
x=find(A==B(1,p+i-1));
y=find(B==A(1,p+i-1));
[A(1,p+i-1),B(1,p+i-1)]=exchange(A(1,p+i-1),B(1,p+i-1));
[A(1,x),B(1,y)]=exchange(A(1,x),B(1,y));
end
%对调函数 exchange.m
function [x,y]=exchange(x,y)
temp=x;
x=y;
y=temp;
end
%适应度函数fit.m,每次迭代都要计算每个染色体在本种群内部的优先级别,类似归一化参数。越大约好!
function fitness=fit(len,m,maxlen,minlen)
fitness=len;
for i=1:length(len)
fitness(i,1)=(1-(len(i,1)-minlen)/(maxlen-minlen+0.0001)).^m;
end
%变异函数 Mutation.m
function a=Mutation(A)
index1=0;index2=0;
nnper=randperm(size(A,2));
index1=nnper(1);
index2=nnper(2);
%fprintf('index1=%d ',index1);
%fprintf('index2=%d ',index2);
temp=0;
temp=A(index1);
A(index1)=A(index2);
A(index2)=temp;
a=A;
end
%染色体的路程代价函数 mylength.m
function len=myLength(D,p)%p是一个排列
[N,NN]=size(D);
len=D(p(1,N),p(1,1));
for i=1:(N-1)
len=len+D(p(1,i),p(1,i+1));
end
end
%连点画图函数 plot_route.m
function plot_route(a,R)
scatter(a(:,1),a(:,2),'rx');
hold on;
plot([a(R(1),1),a(R(length(R)),1)],[a(R(1),2),a(R(length(R)),2)]);
hold on;
for i=2:length(R)
x0=a(R(i-1),1);
y0=a(R(i-1),2);
x1=a(R(i),1);
y1=a(R(i),2);
xx=[x0,x1];
yy=[y0,y1];
plot(xx,yy);
hold on;
end
4、实验
4.1、种群大小改变
N=25; %%城市的个数
M=?; %%种群的个数
ITER=2000; %%迭代次数
m=2; %%适应值归一化淘汰加速指数
Pc=0.8; %%交叉概率
Pmutation=0.05; %%变异概率
实验1 M=100

实验2 M=70

实验3 M=50

结果分析
当M设置的比较大时,种群个数过多,导致运算量增大,影响算法的搜索能力
和运行效率
4.2 迭代次数改变
N=25; %%城市的个数
M=100; %%种群的个数
ITER=?; %%迭代次数
%C_old=C;
m=2; %%适应值归一化淘汰加速指数
Pc=0.8; %%交叉概率
Pmutation=0.05; %%变异概率
实验1 ITER=2000
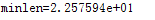

实验2 ITER=1000
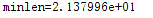

实验3 ITER=3000
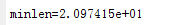

结果分析
若迭代次数过多,会降低算法的运行效率,会陷入已经求解到最优值,但因为迭代次数还未达到设置的值,因此继续进行迭代,降低了算法的运行效率。
若迭代次数过少,会直接导致算法的失败,当算法还未查找到最优值时,已经到达了迭代次数的最大值,退出迭代过程。
4.3、适应值归一化淘汰加速指数改变
N=25; %%城市的个数
M=100; %%种群的个数
ITER=2000; %%迭代次数
%C_old=C;
m=?; %%适应值归一化淘汰加速指数
Pc=0.8; %%交叉概率
Pmutation=0.05; %%变异概率
实验1 m=2
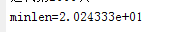

实验2 m=3
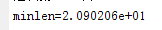

结果分析
适应值归一化淘汰加速指数最好设置为较小
4.4、交叉概率改变
N=25; %%城市的个数
M=100; %%种群的个数
ITER=2000; %%迭代次数
%C_old=C;
m=2; %%适应值归一化淘汰加速指数
Pc=?; %%交叉概率
Pmutation=0.05; %%变异概率
实验1 Pc=0.8
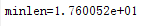

实验2 Pc=0.4
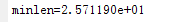

实验3 Pc=0.6


结果分析
从实验结果可以看出,Pc=0.6比其他参数设置所得到的最短路径结果会更好。但是当交叉概率较小时,染色体难以发生交配会使得较难获得较优解。当Pc=0.8时迭代次数最少。
4.5、变异概率改变
N=25; %%城市的个数
M=100; %%种群的个数
ITER=2000; %%迭代次数
%C_old=C;
m=2; %%适应值归一化淘汰加速指数
Pc=0.6; %%交叉概率
Pmutation=?; %%变异概率
实验1 Pmutation=0.05
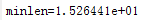

实验2 Pm=0.001
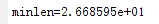

实验3 Pm=0.2
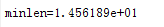

结果分析
当变异概率比较低,且种群较小时,几乎不会有变异的情况
当变异概率比较大时,可能难以找到较优解或者直接破坏较优解的结果
5、结论
从多次的实验结果可以看出,基本上每次实验的迭代次数稳定下来都是在500次上下,而设置不同的参数会直接影响较优解的结果,下面是我从网上查到的每个参数的范围。
(1)种群大小:20~100
(2)迭代代数:1000~2000
(3)交叉概率:0.4~0.99
(4)变异概率:0.0001~0.1
