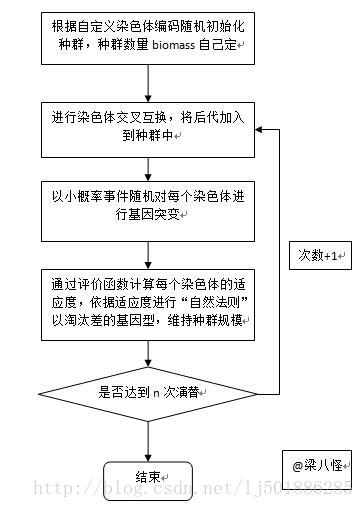
概述
遗传算法(英文缩写为GA)算得上是自然人工系统中比较出名的算法了,属于进化算法的一种,在函数极值、生产调度、航空航天、军事等多方面有着广泛的应用。可以这么说,哪里存在问题待优化,哪里就有GA的用武之地。同类中还有蚁群算法、模拟退火和粒子群算法等等。GA最先由密歇根大学的霍兰德和他的同事学生团队提出(博主接触到GA是由于有幸观得梅拉尼·米歇尔的《复杂》一书),其主要思想来源于达尔文的进化论与自然选择学说,相信各位在高中的时候一定印象很深吧!那么这生物学上的自然演变更替怎么就能与算法牵扯上关系并且还能解决著名的旅行商问题呢?
GA的结构
GA是一种抽象的算法步骤,不存在特定的GA能通用的解决所有的问题,也就是说它具有专一性,即一种GA只能解决某一特定的问题,但所有的GA的步骤都可分为以下五步:
①自定义染色体编码
每一条染色体就意味着一个问题的解,所以我们需要将问题的解编码,编码就是一个数字长串或字符长串都行。例如在求函数极值时我们可以将数字19960610编码成1001100001001001100100010(简单的十进制转二进制即可)。再例如本篇博客的旅行商问题,假若有20个城市,我们给城市A贴上标签1,城市B贴上标签2,依次类推。这样一个长度为20的集合{20 16 13 19 12 18 1 14 3 9 10 6 2 7 5 17 11 15 8 4}即可为旅行商问题中染色体的一种(解)。其排列代表着先后顺序。
②自定义评价函数
染色体编码好了我们该如何评价染色体的优劣的?所以我们必须要根据实际问题自定义一个评价函数。例如在求函数极值时我们可以自然而然把该函数作为评价函数。而在旅行商问题中我们可以把染色体序列按顺序将城市间的距离计算函数当作评价函数。
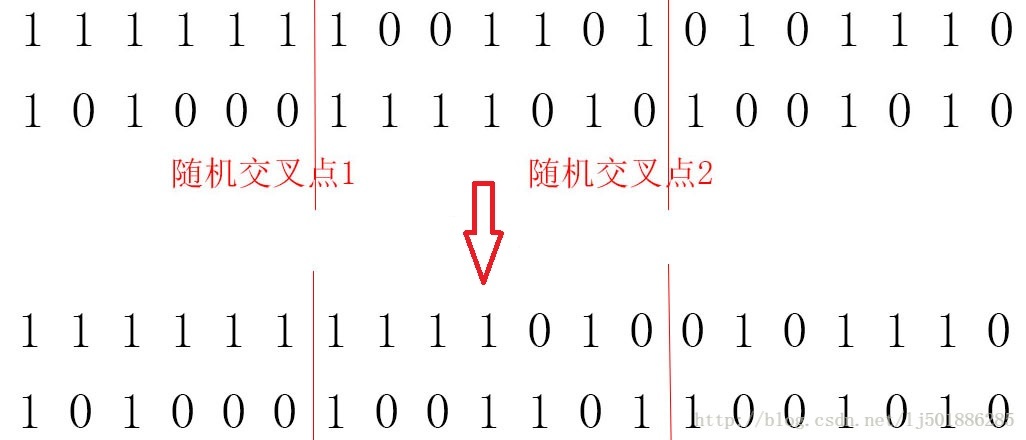
③模拟染色体交叉互换
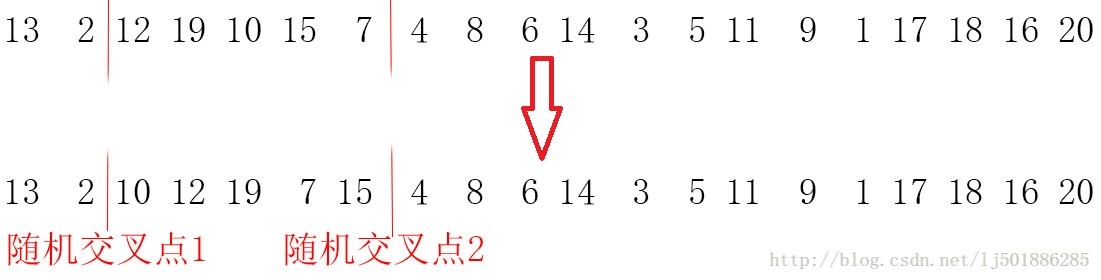
正如大部分生物今天能够很好的适应环境全得益于有性生殖一样,我们所编码的染色体要想不断地进化,就必须得对染色体进行模拟交叉互换。这里我只讲解2点交换,(单点交换只有一个交叉点、n点交换n个交叉点):随机抽取两个染色体匹配,随机选择两个交叉点α、β,将这两个染色体的αβ点间的片段互相交换即为后代。
由于在旅行商问题中,如果单纯的染色体交换会导致某些城市的重复或缺失,所以这里我采取在交叉点间进行随机自乱顺序的办法:
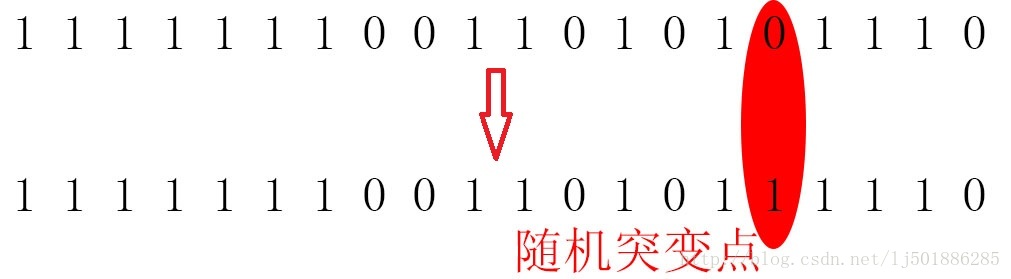
④模拟基因突变
基于突变是保持物种多样性的重要原因之一,这里以一定小概率随机发生基因突变:
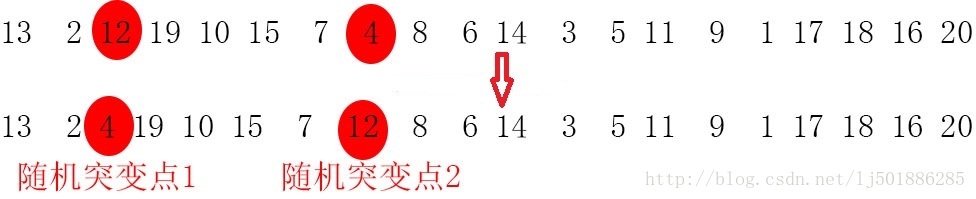
而在旅行商问题中,我们不能只突变一个点,这样同样会造成某些城市的重复和消失,所以这里我们以小概率随机产生两个突变点,将这两个突变点互换即可
⑤根据评价函数淘汰差的基因型
通过评价函数计算出每个染色体的适应度,再根据适应度来淘汰差的基因型。假设有5个染色体,适应度分别为1、2、3、4、5,以适应度高为选择目标的话则这五个染色体能够生存的概率为1/15,2/15 , 3/15,4/15 , 5/15。
在旅行商问题中,我们以距离最短为目标,所以我们将适应度适当调整为1/distance,这样距离越高,其生存概率越低。
算法流程
算法正确性检验
初代种群染色体适应度的分布
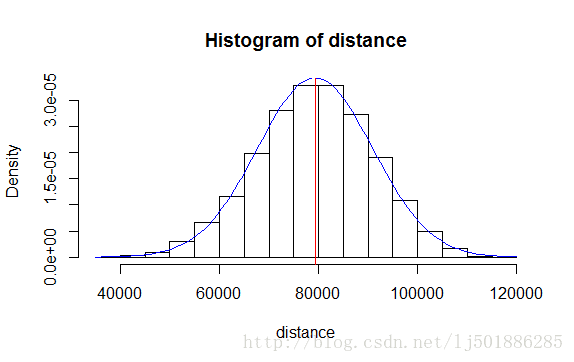
很明显初代种群染色体的适应度满足正态分布,这恰恰验证了我们的模拟很接近自然种群的连续变量性状的分布,正如人类的身高一样。
在自然演替过程中种群染色体适应度的变化
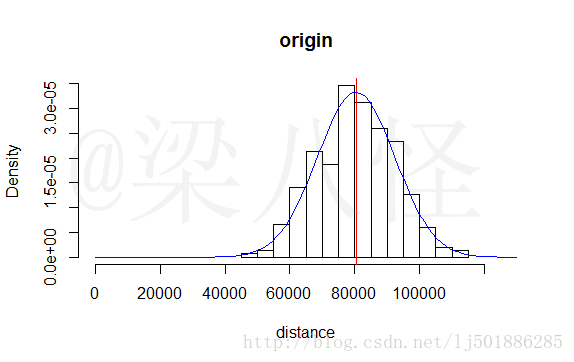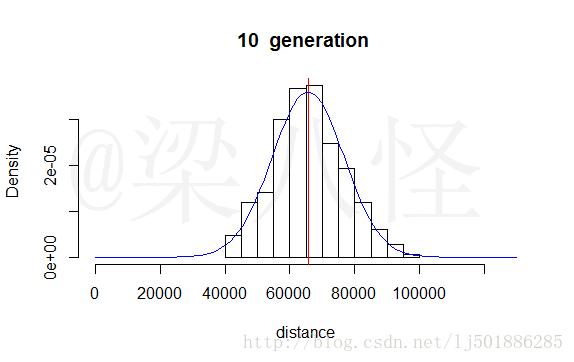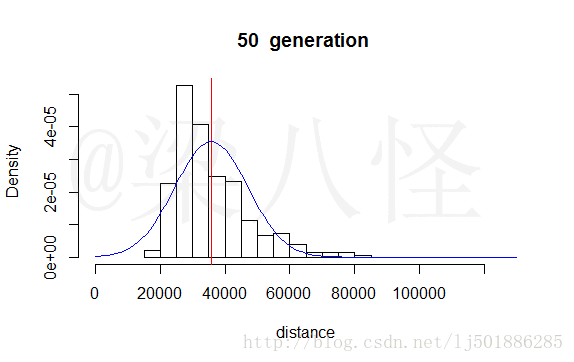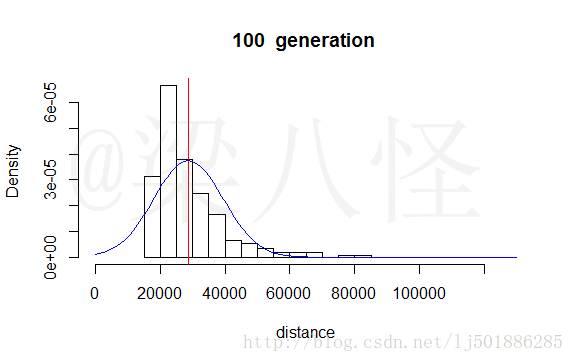
通过初代、5、10、20、25、50、100代的演替我们能明显发现种群的适应度朝着我们的目标方向演替,种群适应度的均值(城市距离和)越来越小。
算法效果图
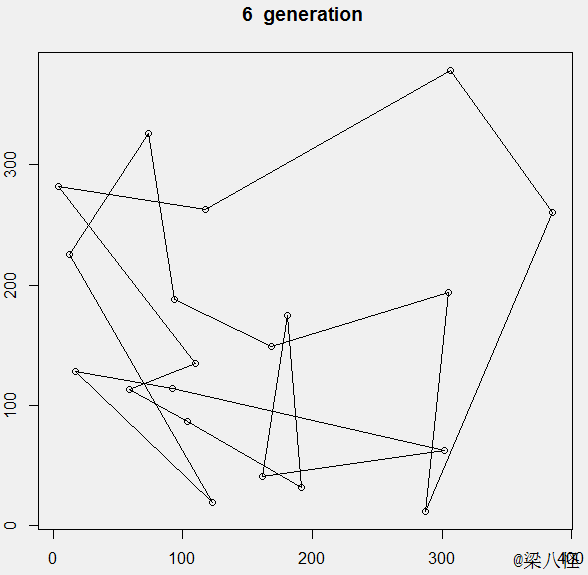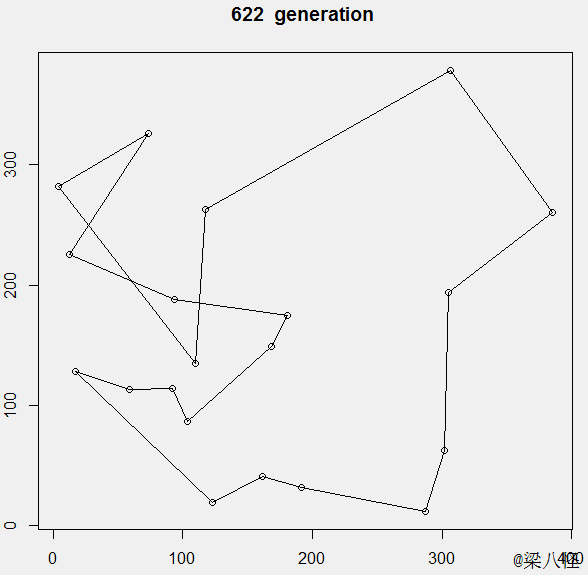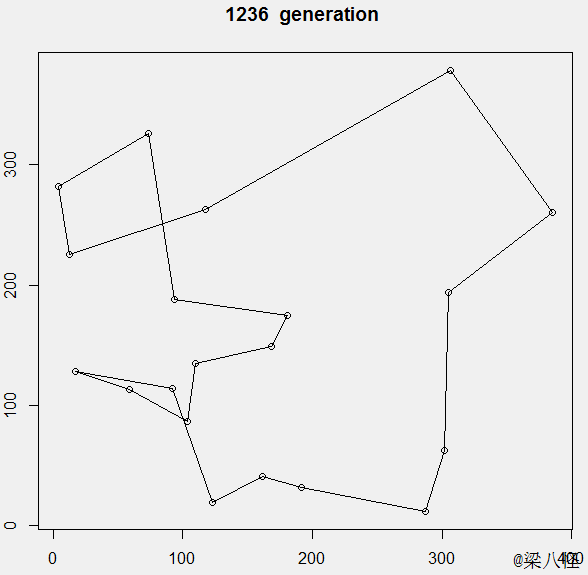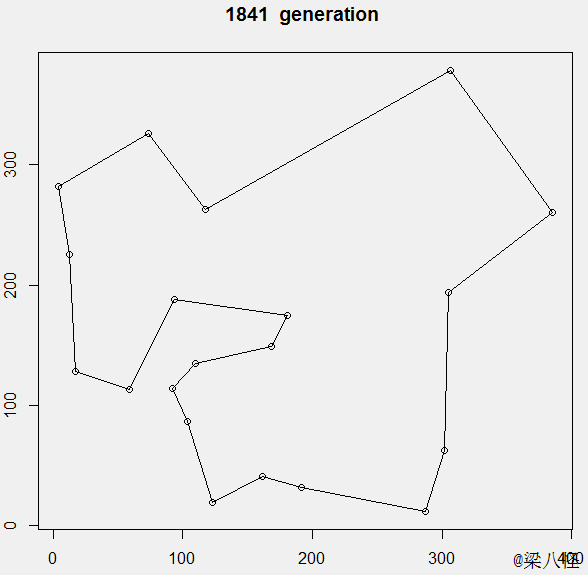
最终大概在1600代左右进化出了最优值
适应度曲线变化图
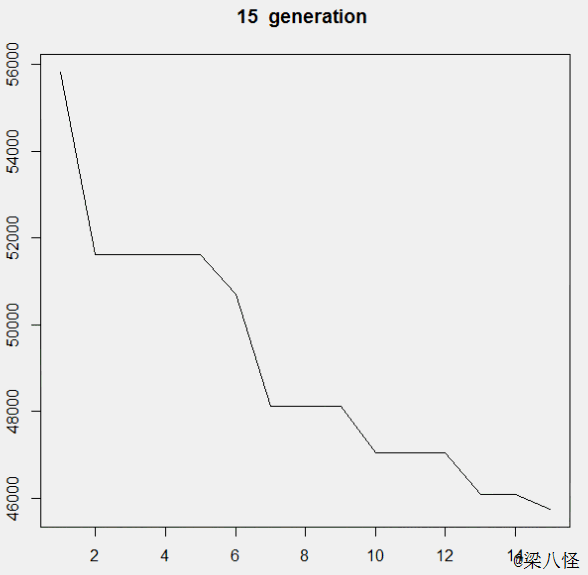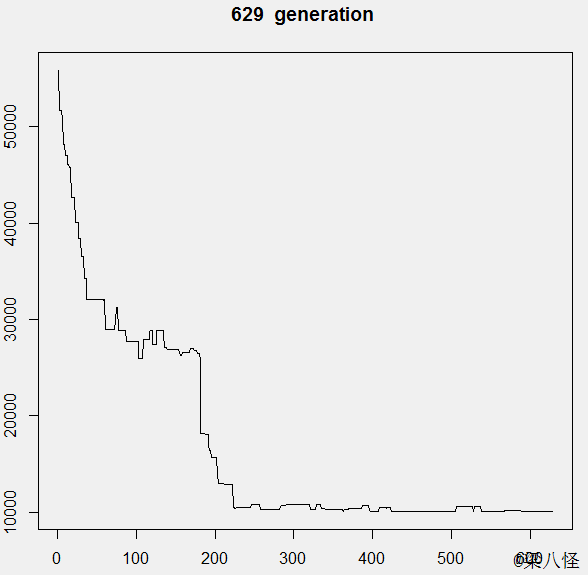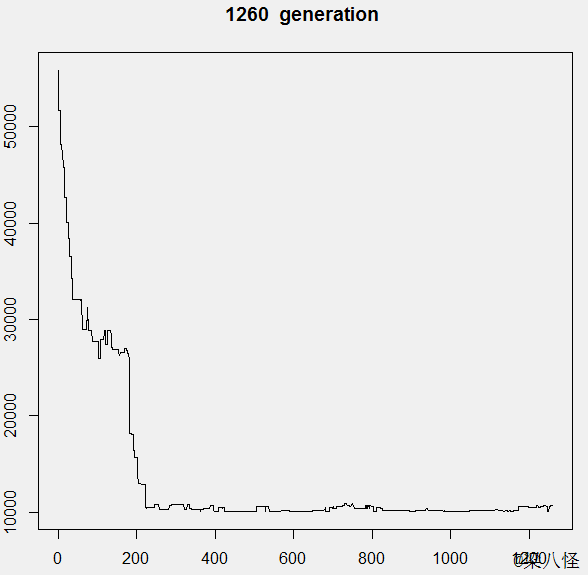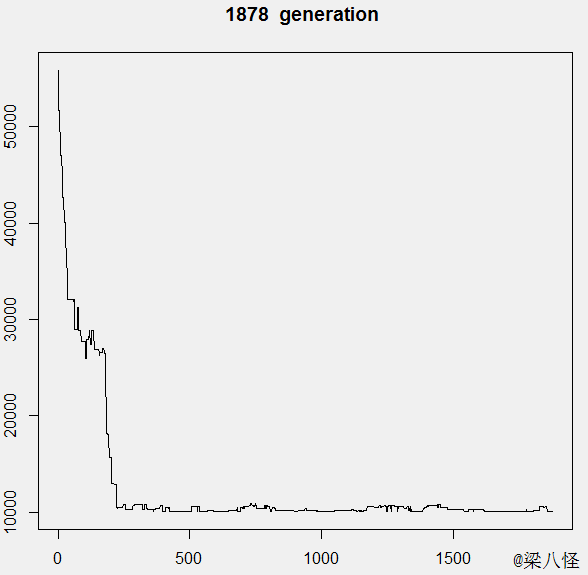
通过曲线的变化,我们能发现在演替的开始时进化较明显,随着演替次数的增多,种群适应值趋于稳定
R代码
GA_travel<-function(cities,biomass = 200,n = 100000,mutation = 0.01){
sourceCpp(file = 'MyDataMining/Cpp/GA.cpp')
ancestor<-c()#初始化种群
for (i in 1:biomass)
ancestor<-c(ancestor,sample(x = 1:nrow(cities),size = nrow(cities),replace = F))
ancestor<-matrix(ancestor,nrow = biomass,byrow = T)
best<-ancestor[1,]
best_value<-getTotalDistance(cities = cities,seq = ancestor[1,])
for (i in 1:n) {#种群n次遗传进化
for (k in 1:biomass) {#交叉互换
point<-sort(sample(x = 1:nrow(cities),size = 2,replace = F))#随机生成2个交叉点
new<-ancestor[k,]#产生新后代
new[point[1]:point[2]]<-sample(new[point[1]:point[2]],size = point[2]-point[1]+1,replace = F)#交叉点间乱序
ancestor<-rbind(ancestor,new)
}
for (k in 1:(2*biomass)) {#基于突变
rate<-runif(n = 1,min = 0,max = 1)#随机数生成概率
if (rate <= mutation) {
point<-sort(sample(x = 1:nrow(cities),size = 2,replace = F))#随机生成2个突变点
temp<-ancestor[k,point[1]]
ancestor[k,point[1]]<-ancestor[k,point[2]]
ancestor[k,point[2]]<-temp
}
}
distance<-apply(ancestor, 1, function(x){#计算每个基因的适应度
return(getTotalDistance(cities = cities,seq = x))
})
min<-min(distance)
if (min < best_value){
best_value<- min
best<-ancestor[which(distance == min),]#最优基因
}
best<-c(best,best[1])#路径首尾相连
plot(cities[best,],type = 'o',main = paste(i,' generation',''),xlab = '',ylab = '')#画出路径
ancestor<-ancestor[sample(1:(2*biomass),size = biomass,replace = F,prob = getProb(distance)),]#淘汰差的基因型,维持种群总数
}
}