R 画网络图
目的:用R做生信分析,画基因样本的网络图,从中观察样本的致病性情况。
一、所用到的包
- library(tidyr)
- library(ggplot2)
- library(reshape2)
- library(readr)
- library(network)
- library(dplyr)
- library(plyr)
- library(sna)
- library(GGally)
- library(ggnetwork)
- library(tidygraph)# tidy graph analysis
- #library(tidyverse)# tidy data analysis
- library(ggraph)
- library(stringr)
- library(networkD3)
- library(igraph)
- library(visNetwork)
- library(threejs)
- library(ndtv)
- library(grid)
- library(Rmisc)
- #library(vioplot)
- library(tibble)
13行中:tidyverse包集成了很多画图的包:如下
tidyverse_packages(include_self = TRUE)
[1] "broom" "cli" "crayon" "dplyr" "dbplyr" "forcats" "ggplot2" "haven" "hms" "httr"
[11] "jsonlite" "lubridate" "magrittr" "modelr" "purrr" "readr" "readxl\n(>=" "reprex" "rlang" "rstudioapi"
[21] "rvest" "stringr" "tibble" "tidyr" "xml2" "tidyverse"
但是如果同时使用这些包,会提示tidyverse_conflicts(),使用tidyverse_conflicts()可以查看tidyverse和其他哪些包冲突:
> tidyverse_conflicts()
-- Conflicts ------------------------------------------ tidyverse_conflicts() --
x tidygraph::arrange() masks dplyr::arrange(), plyr::arrange()
x tibble::as_data_frame() masks igraph::as_data_frame(), dplyr::as_data_frame()
x purrr::compact() masks plyr::compact()
x purrr::compose() masks igraph::compose()
x dplyr::count() masks plyr::count()
x tidyr::crossing() masks igraph::crossing()
x dplyr::failwith() masks plyr::failwith()
x tidygraph::filter() masks dplyr::filter(), stats::filter()
x igraph::groups() masks tidygraph::groups(), dplyr::groups()
x dplyr::id() masks plyr::id()
x dplyr::lag() masks stats::lag()
x tidygraph::mutate() masks dplyr::mutate(), plyr::mutate()
x tidygraph::rename() masks dplyr::rename(), plyr::rename()
x purrr::simplify() masks igraph::simplify()
x dplyr::summarise() masks plyr::summarise()
x dplyr::summarize() masks plyr::summarize()
所以我注释掉了tidyverse,需要用哪个包就自己添加。
二、画网络图遇到的问题
1.颜色自定义
我根据list中属性的种类来定义nodes的颜色,de[,5]中的值为“P”、“V”、“B”,三个类别。
常规的写法是:
geom_node_point(size = 3, alpha = 1,aes(color =de[,5])),
这样系统自动分陪颜色,红绿蓝给BPV,
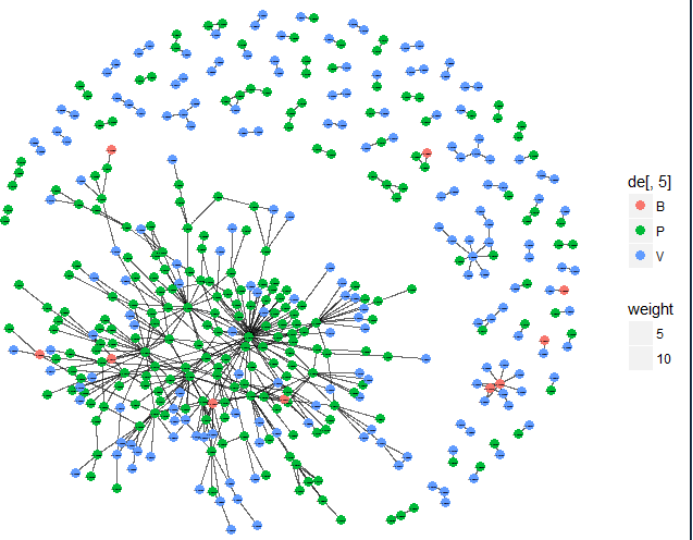
但是会出现以下问题:
A) de[,5]中PVB 出现的顺序会影响到颜色的标记。
B) de[,5]中PVB并不是三种都出现,而是三种的随机组合出现,比如只有PV或者只有VB或者只有PB。
以上原因导致网络图中颜色不对应,第一幅图中红=B 绿=P 蓝=V,如果de[,5]中只有BV两种,会生成红=B,绿=V,导致图例和实际的PVB值不对应。
同时因为看的人肯能出现色盲的情况,一般不用红色和绿色来表示nodes的颜色,所以需要自定义颜色。
比如我需要自定义颜色:效果是这样
'V'='#b27390', 'P'='#f15941','B'='#42a3c6'
我用的ggraph这个画图函数,所以只需要加上scale_color_manual就行:
(如果使用的是ggplot,则用scale_fill_manual)。
这里需要注意scale_color_manua有两种写法:
1)scale_color_manual(name="diagnosis",labs=c(‘V’,’P’,’B’) values=c(''#b27390', '#f15941','#42a3c6'))
2)scale_color_manual(name="diagnosis", values=c('V'='#b27390', 'P'='#f15941','B'='#42a3c6'))
必选参数values:https://blog.csdn.net/songzhilian22/article/details/49388973
values,用于指定这个标度应该生成的值。如果这个向量中的元素是有名称的。则它将自动匹配输入和输出的值,否则它将按照离散型变量中水平的先后次序进行匹配。所以选择第2中方法给定义颜色,以免随机产生颜色。
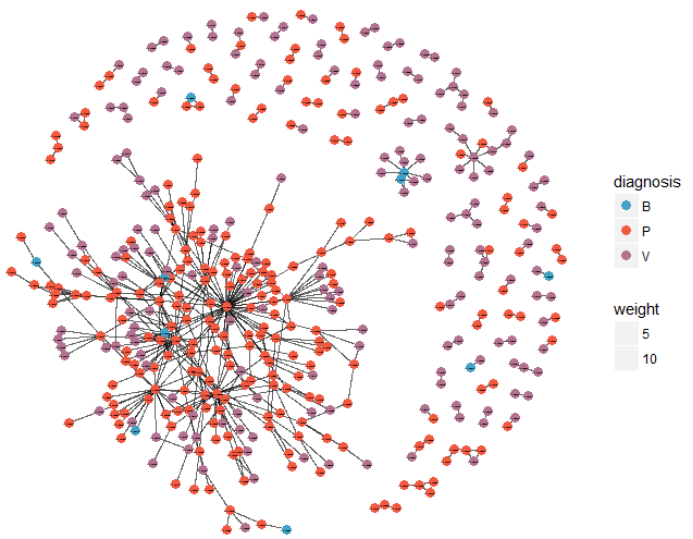
最后的结果如下:
2.多样本for循环的使用问题
本数据中包含多个样本,在画图之前要对样本进行切割,使用split最好不过了。
split(data1, as.factor(data1$gene))#按照基因切割
split(gene, gene$sampleid)#按照样本切割
切割后添加过滤条件,一个样本至少携带两个variant的sample保留下来做网络图。行数≥2的说明只有两个variant,按照gene切割的安装代码如下,用到第一个for循环:
for (gene in data_per_gene) {
data_per_sample_per_gene <- split(gene, gene$sampleid)
########### Then we enumerate each sample to obtain the edges ###############
for (sample in data_per_sample_per_gene) {
######### we demand sample carry >=2 variants in the same gene to be considered #############
if ((nrow(sample)>=2)&&(sum(sample$GT)==2)){
####### create combinations of variants #################################
a <- combn(sample$num_id,2)
b <- data.frame(t(a))
b$sample <- as.character(unique(sample$sampleid))
b$gene <- as.character(unique(sample$gene))
edge <- rbind(edge, b)
}
}
}
然后对edge中的边所对应的gene切割
edge_per_gene <- split(edge, as.factor(edge$gene))
接下来我们为每个基因的每一个variant创建边:用到第二个for循环
for (gene in edge_per_gene)
这个循环里,我们不仅要完成边和点的连接,还要完成画图。
3.连接边和点
对edge中的X1,X2进行分组操作,然后计算边的权重。(不同的样本有相同的边也合并计算权重,值保留一条边,用权重weight表示边连接的次数)
edge2 <- gene %>%
group_by(X1, X2) %>%
summarise(weight = n()) %>%
ungroup()
unique(edge2)
然后定义nodes,选择X1和X2中唯一出现的num_id合并去重,
nodesX1 <- subset(nodes0, (nodes0$num_id %in%edge2$X1))
nodesX2 <- subset(nodes0, (nodes0$num_id %in%edge2$X2))
nodes2<-unique(rbind(nodesX1,nodesX2))
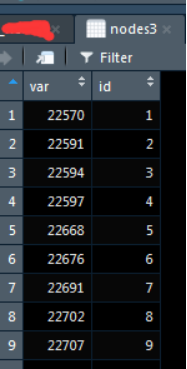
把nodes2的第一列单独拿出来:
nodes3<-as.data.frame(nodes2[,1])
接下来建立点和边的连接关系,我们把nodes3 重新编号命名,
nodes3$ID <- seq(1:nrow(nodes3))
colnames(nodes3) <- c("var","id")
nodes3$var <- as.integer(nodes3$var)
edges2中的边也通过id与nodes3建立连接:
edges <- edge2 %>%
left_join(nodes3, by = c(X1 = "var")) %>%
rename(from = id)
edges <- edges %>%
left_join(nodes3, by = c(X2 = "var")) %>%
rename(to = id)
最后,选择边的集合:
edges <- select(edges, from,to,weight)
4.画网络图
我们创建无向图,directed = FALSE。
我使用的library(network),网上还有library(networkD3),library(sna)等。
routes_network <- network(edges,
vertex.attr = nodes3,
matrix.type = "edgelist",
ignore.eval = FALSE)
routes_tidy <- tbl_graph(nodes = nodes3, edges = edges, directed = FALSE)
5.ggraph画图
画图之前,我们自定义点的名字nodename
de<- routes_tidy %>%
activate(nodes) %>%
as.data.frame()
de<-cbind(de,nodes2$color_net)
colnames(de) <- c("var","id","degree","centrality","diagnosis")
nodename<-paste(de$degree,"-",de$var)
最后使用ggraph画图:
ggraph(routes_tidy ,layout = "fr") +
geom_edge_link(aes(width = weight),alpha = 0.6) +
geom_node_point(size = 3, alpha = 1,aes(color =de[,5])) +
scale_color_manual(name="diagnosis", values=c('V'='#b27390', 'P'='#f15941','B'='#42a3c6'))+#分配颜色
geom_node_text(aes(label = nodename), size = 0.5, repel = FALSE) +
scale_edge_width(range = c(0.2, 2)) +
labs(x = "", y = "", title = filename) +
theme(axis.ticks = element_blank()) +
theme(axis.text.x = element_blank())+
theme(axis.text.y = element_blank())+
theme(panel.grid=element_blank())+
theme(panel.background=element_blank())+
theme(panel.border=element_blank())
5.计算nodes的相关属性
中心度:点度频率(每种点度数的个数/所有点个数)
routes_tidy <-routes_tidy %>%
activate(nodes) %>%
mutate(centrality = degree(routes_tidy)/vcount(routes_tidy))
度:degree
#nodes的度
routes_tidy <-routes_tidy %>%
activate(nodes) %>%
mutate(degree = centrality_degree())