2.3 Segmentation图像分割
第一步:Highlight the “Volumes” field and then select the subject’s registered anatomical image eg.
sM00223_002.img.
第二步:Highlight “Save Bias Corrected” and select “Save Bias Corrected”.
第三步:Highlight “Deformation Fields” the bottom of the list and select “Forward”.
第四步:Save the job file as segment.mat and then press RUN.
SPM将会利用默认设置的组织概率图分割结构像,所利用的原理详看文献
J. Ashburner and K.J. Friston. Computing average shaped tissue probability templates. NeuroImage, 45(2):333–341, 2008
经过图像分割后,将会得到灰质图和白质图像,利用CheckReg按钮可以观察图像与结构像的差距,
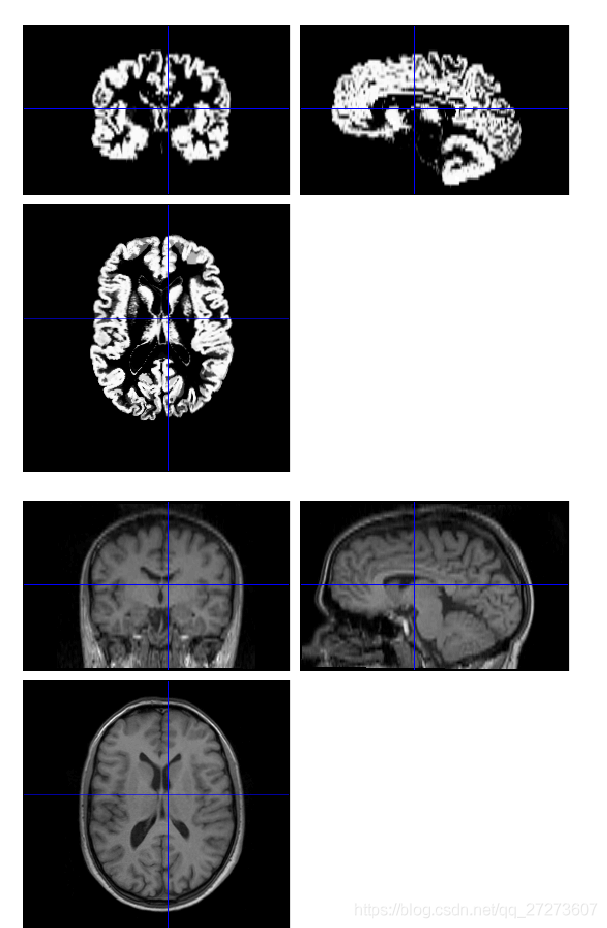
SPM will also write a deformation field,file y_sM00223_002.nii。
2.4 Normalise(配置到某个大脑模板上)
Select Normalise (Write) from the Normalise pulldown menu。然后按照如下步骤操作即可,

SPM将空间规范化后的数据写进文件夹dummy中,这些文件名字有w开头。
If you wish to superimpose a subject’s functional activations on their own anatomy, you will also need to apply the spatial normalisation parameters to their (bias-corrected) anatomical image
按照如下操作即可。

同样可以用利用CheckReg按钮可以观察图像与初始的结构像的差距。

2.5 Smoothing(平滑处理)
Press the Smooth button。然后按如下步骤操作即可。

然后利用CheckReg按钮可以比较平滑之前的数据和平滑之后的数据的区别。

经过以上几个步骤后数据预处理部分已经完成,接下来就要进行统计分析了。