题目以及数据介绍
Github 代码以及数据
Github
初始思想
1.从头开始,先看一下初始数据以及数据的简单分析吧
训练数据,最后一列是血糖:
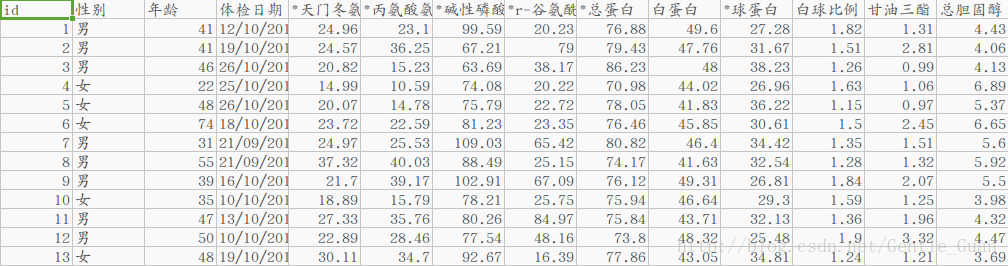
A榜测试数据
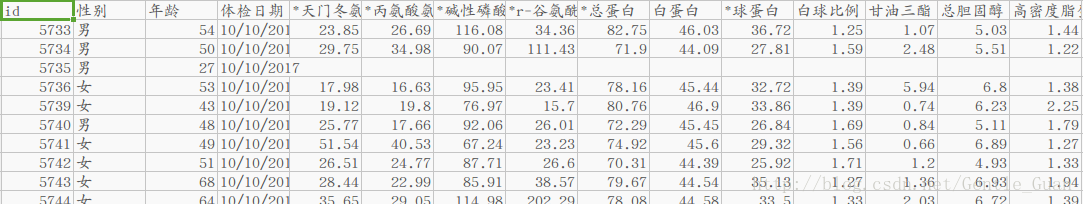
第九个特征与标签的关系分布
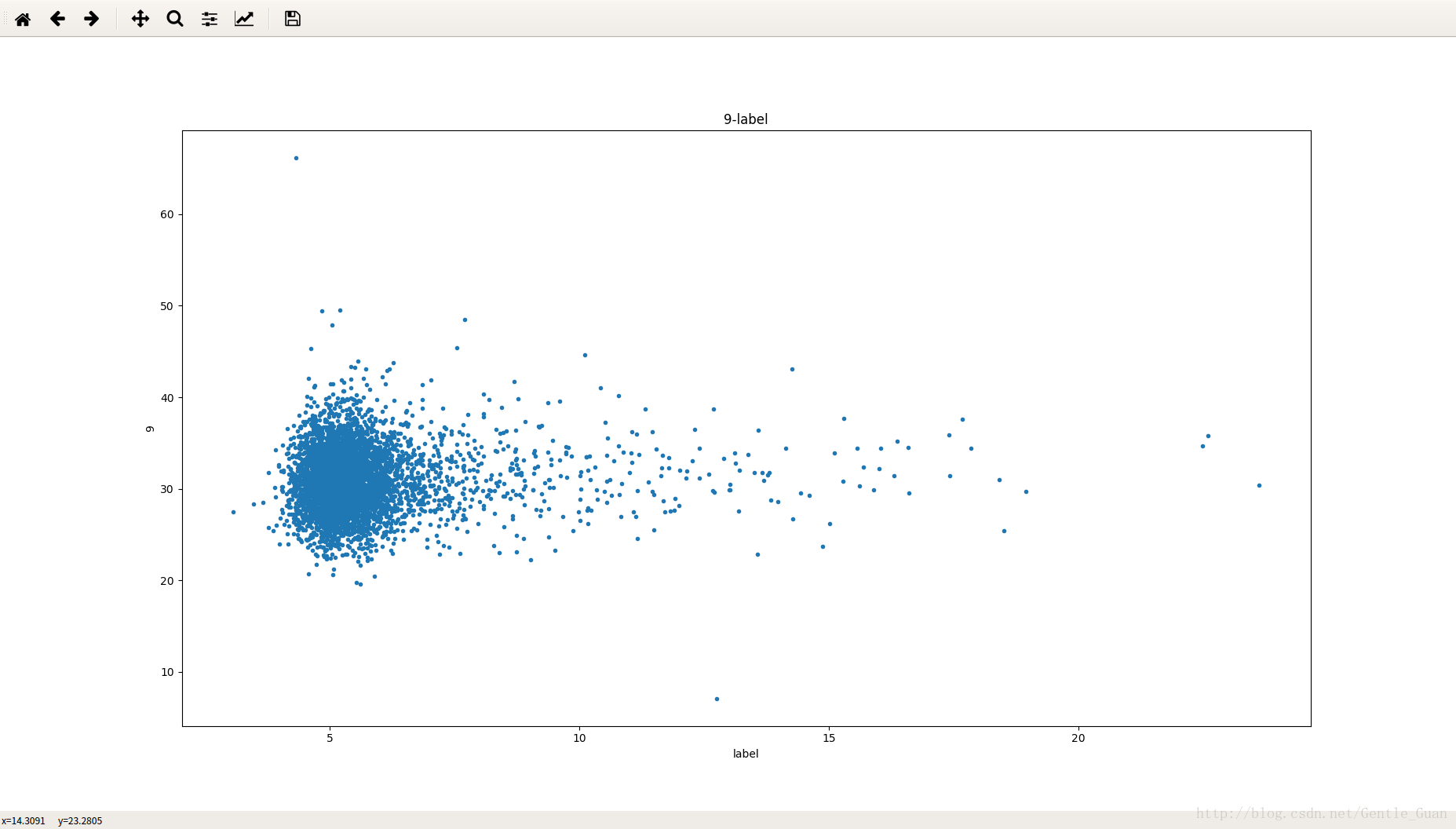
第三十八个
各个特征计数(有点糊)
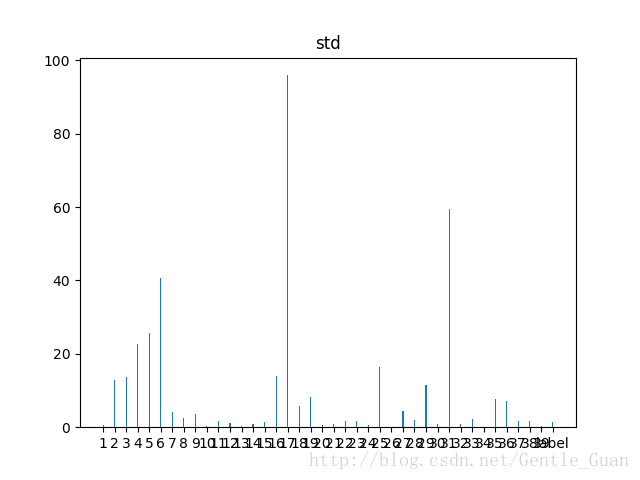
各个特征(标签)的标准差
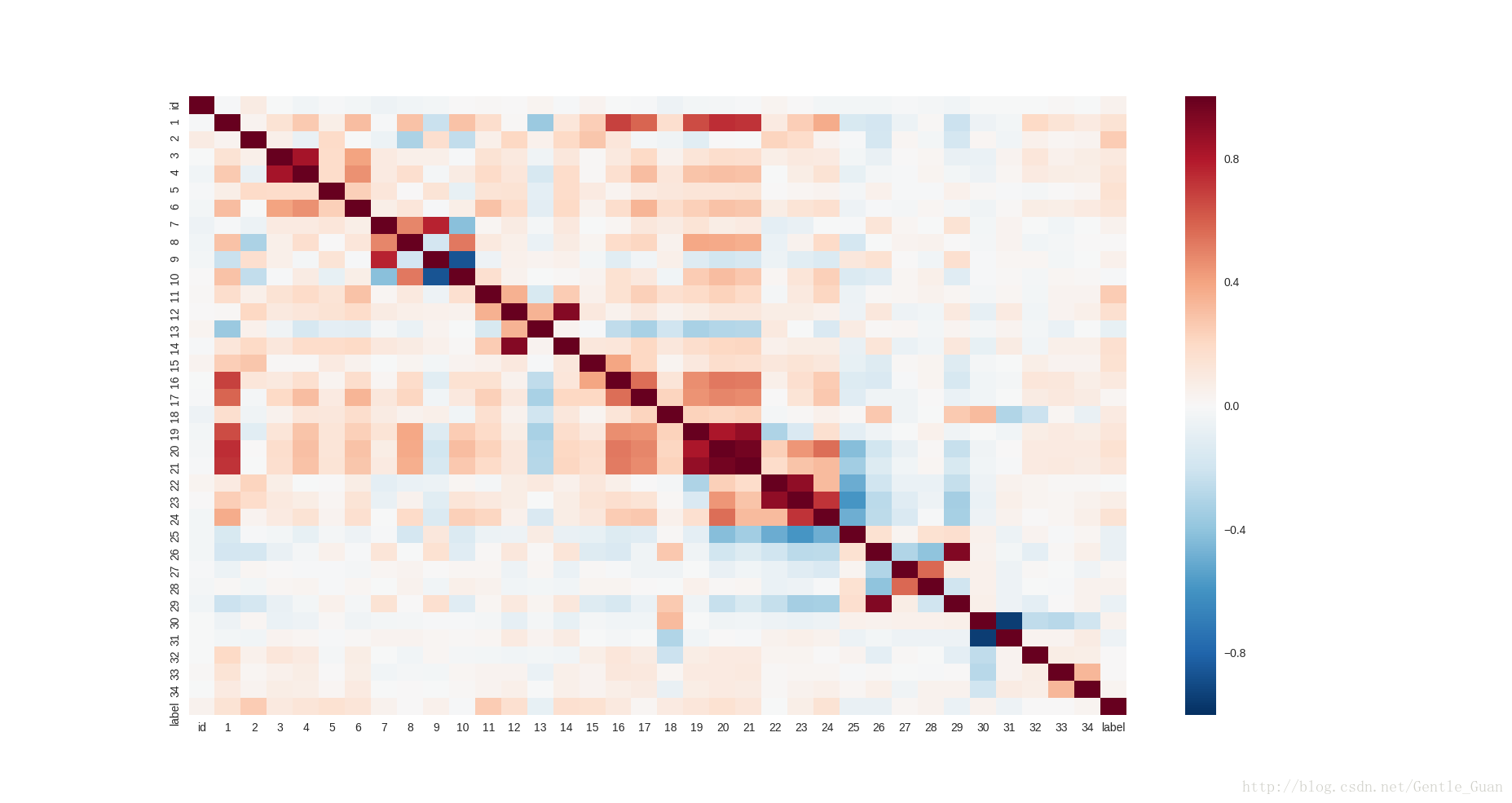
热力图(反应相关性)
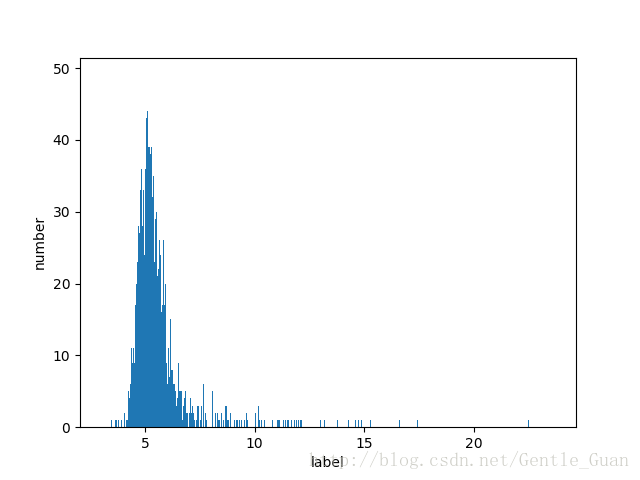
标签数量分布(简单取整)
2. 对以上数据分析一下:
1)特征的数据分布基本上是类似图中的分布规律(符合实际情况,说明该指标大部分人都正常)
2)个别特征的标准差比较大,说明有离群值,先baseline在进行离群值处理
3)个别特征缺失值比较多(尤其中间五个,缺失近乎75%,实际指标为乙肝五项),所以暂不使用
4)由于特征是医学指标,所以需要挖掘内在的医学上的关系
5)数据中时间以及id暂不使用,并且性别映射为数值: 男1女0
3. 根据上述分析进行简单模型跑出baseline,xgboost模型
# 得分score按照题目要求实现
# 利用xgboost的树模型以及泊松分布(label大致符合泊松分布)
# 误差rmse为均方根误差(均方误差开方)
# 参数如下,1000-50次迭代
para = {
'booster': 'gbtree',
'objective': 'count:poisson',
'eval_metric': 'rmse',
'eta': 0.3, # alpha
'silent': 1,
'max_depth': 3,
'subsample': 0.9, # 0.9样本选取率 防止过拟合
"miss": -999,
"lambda":1.5
}
结果:
本地cv-5折测试得分score
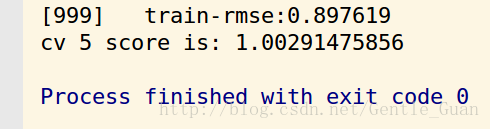
提交后得分score

效果不是很好0.0
中期思想
1.
1) 既然是医学指标,那么就要结合医学上进行分组,就是同一器官和平均值+总量这种东西融合在一起
2) 建立阀值,删除.(经过本地测试 18最优)
# 去掉乙肝五项 id 体检日期
train_file.drop(['乙肝表面抗原', '乙肝表面抗体', '乙肝e抗原', '乙肝e抗体', '乙肝核心抗体', 'id', '体检日期'],axis=1,inplace=True)
test_file.drop(['乙肝表面抗原', '乙肝表面抗体', '乙肝e抗原', '乙肝e抗体', '乙肝核心抗体', 'id', '体检日期'],axis=1,inplace=True)
# 白球比例是指肝功能白球比例(白球比)=白蛋白/球蛋白=白蛋白/(总蛋白-白蛋白)
# 单核细胞 白细胞冲突
train_file.drop(['单核细胞%','白球比例','白蛋白','*总蛋白'],axis=1,inplace=True)
test_file.drop(['单核细胞%','白球比例','白蛋白','*总蛋白'],axis=1,inplace=True)
# 合并相似项目 酶
train_file['酶'] = train_file['*天门冬氨酸氨基转换酶'] + train_file["*丙氨酸氨基转换酶"] \
+ train_file["*碱性磷酸酶"] + train_file["*r-谷氨酰基转换酶"]
test_file['酶'] = test_file["*天门冬氨酸氨基转换酶"] + test_file["*丙氨酸氨基转换酶"] \
+ test_file["*碱性磷酸酶"] + test_file["*r-谷氨酰基转换酶"]
# 血细胞计数 * 平均参数
train_file['红细胞总血红蛋白量'] = train_file['红细胞计数'] * train_file['红细胞平均血红蛋白量']
test_file['红细胞总血红蛋白量'] = test_file['红细胞计数'] * test_file['红细胞平均血红蛋白量']
train_file['红细胞总血红蛋白浓度'] = train_file['红细胞计数'] * train_file['红细胞平均血红蛋白浓度']
test_file['红细胞总血红蛋白浓度'] = test_file['红细胞计数'] * test_file['红细胞平均血红蛋白浓度']
train_file['红细胞总体积'] = train_file['红细胞计数'] * train_file['红细胞平均体积']
test_file['红细胞总体积'] = test_file['红细胞计数'] * test_file['红细胞平均体积']
train_file['血小板总体积'] = train_file['血小板计数'] * train_file['血小板平均体积']
test_file['血小板总体积'] = test_file['血小板计数'] * test_file['血小板平均体积']
# cv 0.7912094213627026
# 肾指标
train_file['肾'] = train_file['尿酸'] + train_file['尿素'] + train_file['肌酐']
test_file['肾'] = test_file['尿酸'] + test_file['尿素'] + test_file['肌酐']
# 嗜酸细胞
train_file["嗜酸细胞"] = train_file['白细胞计数'] * train_file["嗜酸细胞%"]
test_file["嗜酸细胞"] = test_file['白细胞计数'] * test_file["嗜酸细胞%"]
# 删除嗜碱细胞 与嗜酸细胞矛盾
train_file.drop(['嗜碱细胞%'],axis=1,inplace=True)
test_file.drop(['嗜碱细胞%'],axis=1,inplace=True)
# 删除年龄异常值
train_file = train_file[train_file['血糖'] <= 18]
# 利用lgb中goss+泊松模型的lgb参数设置
# 1500-50迭代
lgb_para = {
'learning_rate': 0.03,
'boosting_type': 'goss',
'objective': 'poisson',
'metric': 'rmse',
'max_depth': 4,
'sub_feature': 0.9,
'colsample_bytree': 0.9,
'verbose': -1
}关于嗜酸性粒细胞
lgb结果:
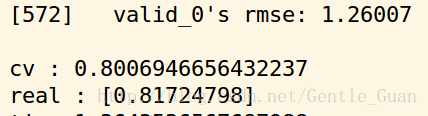
效果还可以
线下cv 5折结果得分稍微比真实高一点
这个real score是A榜数据答案全量测试得分
当然第一次做的时候没有这个答案,第一次得分就是随机选点测试得分
xgboost版本:
xgb_para = {
'booster': 'gbtree',
'objective': 'count:poisson',
'eval_metric': 'rmse',
'eta': 0.03, #
'silent': 1,
'max_depth': 4, # 4 best
'subsample': 0.9, # 0.9 best
"miss": -999,
"colsample_bytree": 0.7,
"lambda":1.5 # 1.5 best
}xgb结果:
其实score差不多
但是
很明显xgboost的时间比lgb长
其他思想
0) catboost应用
1) cnn应用(效果不是很好)
2) 上述模型的融合
心得感悟
觉得模型调参只是短暂的繁琐但是脑力消耗比较小
其次,特征处理尤为重要
总结: 对特征不够敏感, 尤其是比较专业的方面, 其次,数学功底有待加强, 否则分析数据手足无措 = =
后续: 加强模型数学理解比如本次的goss+泊松, 准备知识储备去面试 = =
