1. 参考基因组下载
目前常用的国际通用的人类参考基因组在NCBI,UCSC和ENSEMBL的版本和对应情况如下:
| NCBI | UCSC | ENSEMBL |
|---|---|---|
| GRCH36 | hg18 | ENSEMBL release_52 |
| GRCH37 | hg19 | ENSEMBL release_59/61/64/68/69/75 |
| GRCH38 | hg38 | ENSEMBL release_76/77/78/80/81/82 |
ENSEMBL和NCBI数据库的版本较为复杂,因此我们选择下载UCSC的hg19作为参考基因组
进入UCSC后选择Downloads > Genome Data > Human > hg19 Full Dataset
使用axel(sudo apt install axel)下载chromFa.tar.gz文件
下载完成后需要解压文件并将所有染色体序列文件拼接整合成一个完整的hg38.fa文件
#解压文件
x chromFa.tar.gz
#整合文件
cat *.fa > hg19.fa
#删除多余文件
rm -rf chr*
2.基因注释文件下载
参考基因组的解读需要额外的注释文件帮助,我们要在gencode数据库(http://www.gencodegenes.org/)下载需要的注释文件,进入网站后选择Data > Human > GRCh37-mapped Releases

选择最新版本的注释信息,下载GFF和GTF文件
axel ftp://ftp.ebi.ac.uk/pub/databases/gencode/Gencode_human/release_28/GRCh37_mapping/gencode.v28lift37.annotation.gtf.gz
axel ftp://ftp.ebi.ac.uk/pub/databases/gencode/Gencode_human/release_28/GRCh37_mapping/gencode.v28lift37.annotation.gff3.gz
x *.gz基因注释文件的两种格式GFF和GTF的区别:
数据结构:都是由9列构成,分别是reference sequence name; annotation source; feature type; start coordinate; end coordinate; score; strand; frame; attributes.前8列都是相同的,第9列不同。
GFF第9列:都是以键值对的形式,键值之间用“=”连接,不同属性之间用“;”分隔,都是以ID这个属性开始。下图中有两个ID,说明是不同的序列。
GTF第9列:同样以键值对的形式,键值之间是以空格区分,值用双引号括起来;不同属性之间用“;”分隔;开头必须是gene_id, transcipt_id两个属性
3. IGV下载与使用
Integrative Genomics Viewer(IGV)是一种探索大型综合基因组数据的高性能交互式可视化工具。它支持各种各样的数据类型,包括基于芯片测序、二代测序数据和基因组注释数据等。
#IGV下载解压
axel http://data.broadinstitute.org/igv/projects/downloads/2.4/IGV_2.4.10.zip
x *.zip
mv IGV_2.4.10 ~/biosoft解压好后的IGV直接运行IGV.sh就可以使用了
首先,从文件中导入下载好的参考基因组文件
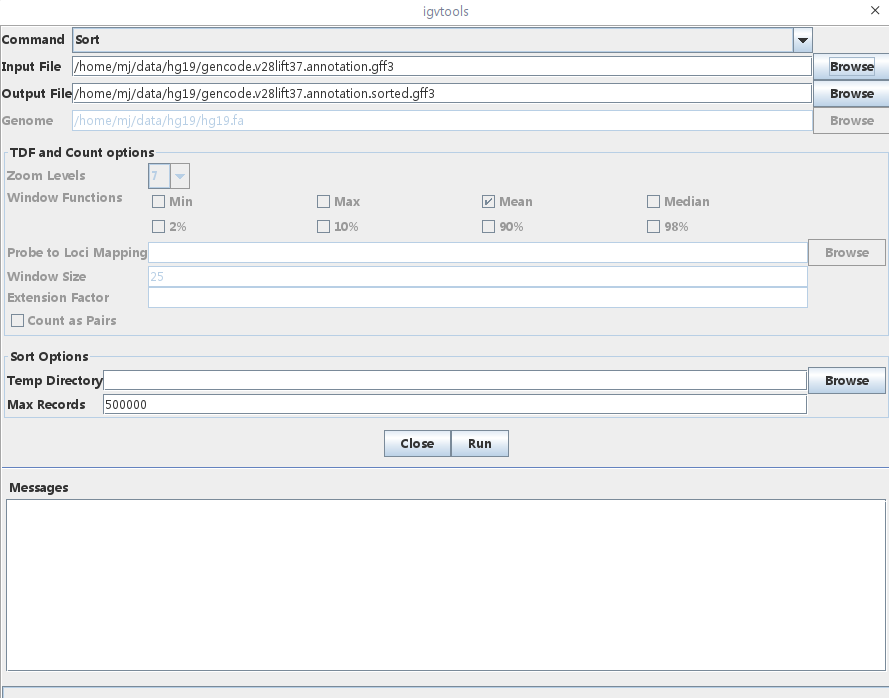
接着导入基因注释文件,打开igvtools对基因注释文件进行sort,之后再导入sort好的注释文件
在NCBI上查找感兴趣的基因(比如LGR5),找到它的染色体位置信息

此处需要注意的是由于基因数据库的版本在不断变化,而hg19属于比较旧的版本,因此我们需要找到之前版本的位置信息即chr12:71832931..71980090
在IGV工具栏的搜索框中输入基因位置chr12:71832931-71980090即可查看lgr5基因的结构

参考阅读:
生物信息学100个基础问题 —— 第24题 GFF,GTF到底是什么?
生物信息学100个基础问题 —— 第25题 GTF/GFF的注释是怎么来的,应该从哪里下载?
