运用深度学习预测肺癌诊断
原文:Forecasting Lung Cancer Diagnoses with Deep Learning
注:本文为The Data Science Bowl (DSB) 2017竞赛的第二名获奖团队中Daniel Hammack的解决方案,其另一成员Julian de Wit的解决方案,可查看用CNN识别CT图像检测肺癌一文。
摘要
在2017年由Kaggle举办的数据科学竞赛中,本团队的解决方案获得了第二名。本次竞赛的目标为构建一个系统,其能根据患者的CT图像,预测患者在一年内患癌的可能性。本文作者的解决方案已在Github上公开。
背景
肺癌是最常见的癌症之一,尤其在北美地区。其是男性最常见的癌症形式,其次为女性。全球每年有160万人死于肺癌,仅在美国每年就有225000肺癌新增病例。此外,肺癌也是低存活率的癌症之一,平均5年的存活率低于20%。然而,早期发现的平均概率至少是肺癌存活率的两倍。
The Data Science Bowl (DSB) 是Kaggle举办的年度机器学习竞赛。2017年的竞赛是该赛事的第三届比赛,该次比赛共有2000名选手,其奖金池也高达100万美元。该比赛的目标为构建一个自动化系统,其能预测患者在下一年的CT扫描诊断中是否会被诊断为肺癌。该比赛的数据集只提供了每位患者的一次CT扫描图像,并删除了患者的相关信息。
工具
解决方案完全使用Python高级程序设计语言,并使用了相关的开源科学计算库:
- keras
- theano
- numpy
- scipy
- scikit-learn
- pandas
数据集
本次竞赛的数据集为1600幅高分辨率的胸部CT扫描图像,其切片厚度均小于3mm。扫描图像的大小为 体素,但在世界坐标系的单位中,其大小约为 。感兴趣区域通常在 左右。在训练集中,每幅CT扫描图像对应一个二值标签。
额外数据集
额外的数据集为LUNA16竞赛的数据集,其数据集来源于LIDC数据集,在该数据集的结节注释中包含如下特征:
- diameter
- lobulation
- spiculation
- malignancy
- calcification
- sphericity
在该数据集中放射科医师标记了1200个结节,重点关注结节的直径、分叶、毛刺和恶性程度。
方法
主要步骤为:
- 归一化CT图像;
- 找寻感兴趣区域;
- 预测结节属性;
- 综合结节属性预测,预测患者诊断。
最终的解决方案结合了17层的3D卷积神经网络模型,并由两个集合(ensemble)组成。每个集合(ensemble)中的模型采用不同的体系结构、训练计划、目标、子数据集和激活函数。
数据集归一化
首先,将每幅CT图像的大小调整为每一像素表示 的体积,从而保证同一模型能够应用于不同切片厚度的CT图像。然后,将切片的HU值的范围 转换为 ,其中HU值为-1000将映射为0,400将映射为1。最后,对CT图像进行粗略肺部分割,从而消除与肺部不相交区域。
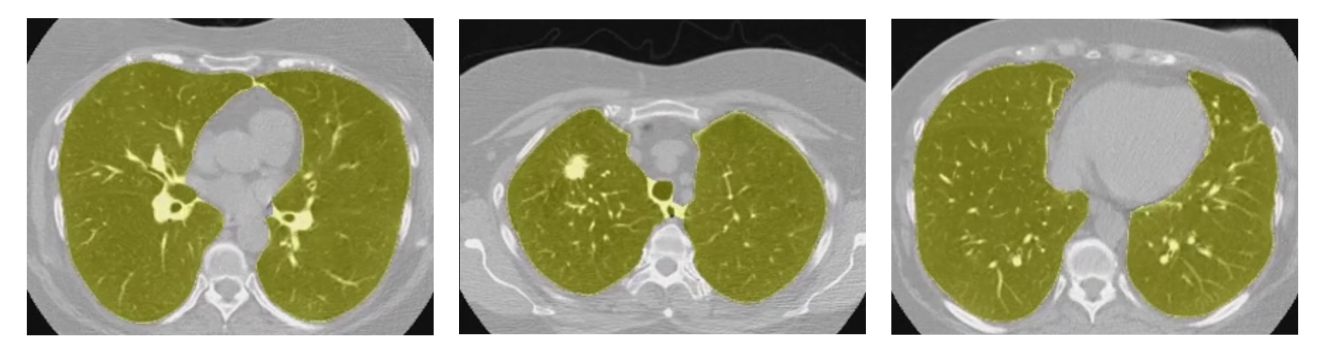
上图为肺部分割例子,其中肺部区域为黄色。
候选结节检测
第二步为检测CT图像中对于诊断有有用信息的区域,即感兴趣区域。以一幅典型的CT图像为例,一幅原始的CT图像每个轴的尺寸为 ,而本方案使用 的子图像用于诊断,因此对于结节模型需要对每一幅CT图像处理上百次。然而,即使在肺癌晚期的情况下,大部分肺部看起来也是正常的。因此,若结节模型处理的区域无异常时,将造成巨大的计算浪费。
为了避免这一问题,从而构建一个识别处理区域是否异常的模型。该模型与结节模型类似,但其只用了90%的正样例和10%的异常样本进行训练。该模型使用一个单一输出的回归目标用于表示结节的加权属性,其中结节属性越重要,权重也越大,即越异常的区域,其权值也越大。(此处可理解为将整个肺部视为异常区域,越是感兴趣区域其权值越大。)
在每次处理中,异常区域的取值范围为 。其中,异常区域定义为CT图像中任一预测值大于某一阈值的 的区域。(本解决方案选择1作为这一阈值。)
结节属性预测
鉴于本部分的重要性,构建了两个集合(ensemble)用于结节属性(直径、分叶、毛刺和恶性程度)的预测。
在训练中发现,结节属性模型与本方案的体系结构相比,多任务训练好于单一任务(以结节的恶性程度为目标)训练。再进一步发现,“分支”模型在训练多个目标时产生了更好的效果。若分支处理越晚,模型输出越相关,则为下一步骤提供较少的信息。
采用每一输入( )的几个随机3D转换,并对结果进行平均。
下图显示了训练期间不可见结节的3D特征图。基于每个位置的像素对恶性肿瘤的贡献来对图像进行着色,其中通过黑化图像的立方体并记录恶性程度的变化来估计其贡献。

从结节属性预测诊断结果
手动构建如下特征:
- max malignancy, spiculation, lobulation, and diameter across nodules (4 features)
- stdev of malignancy, spiculation, lobulation, and diameter predictions across nodules (4 features)
- location of most malignant nodule (3 features, one for each dimension in the scan)
- stdev of nodule locations in each dimension (3 features)
- nodule clustering features (4 features)
其中,结节聚类特征涉及在结节位置上运行聚类算法(用于避免重复计数)。
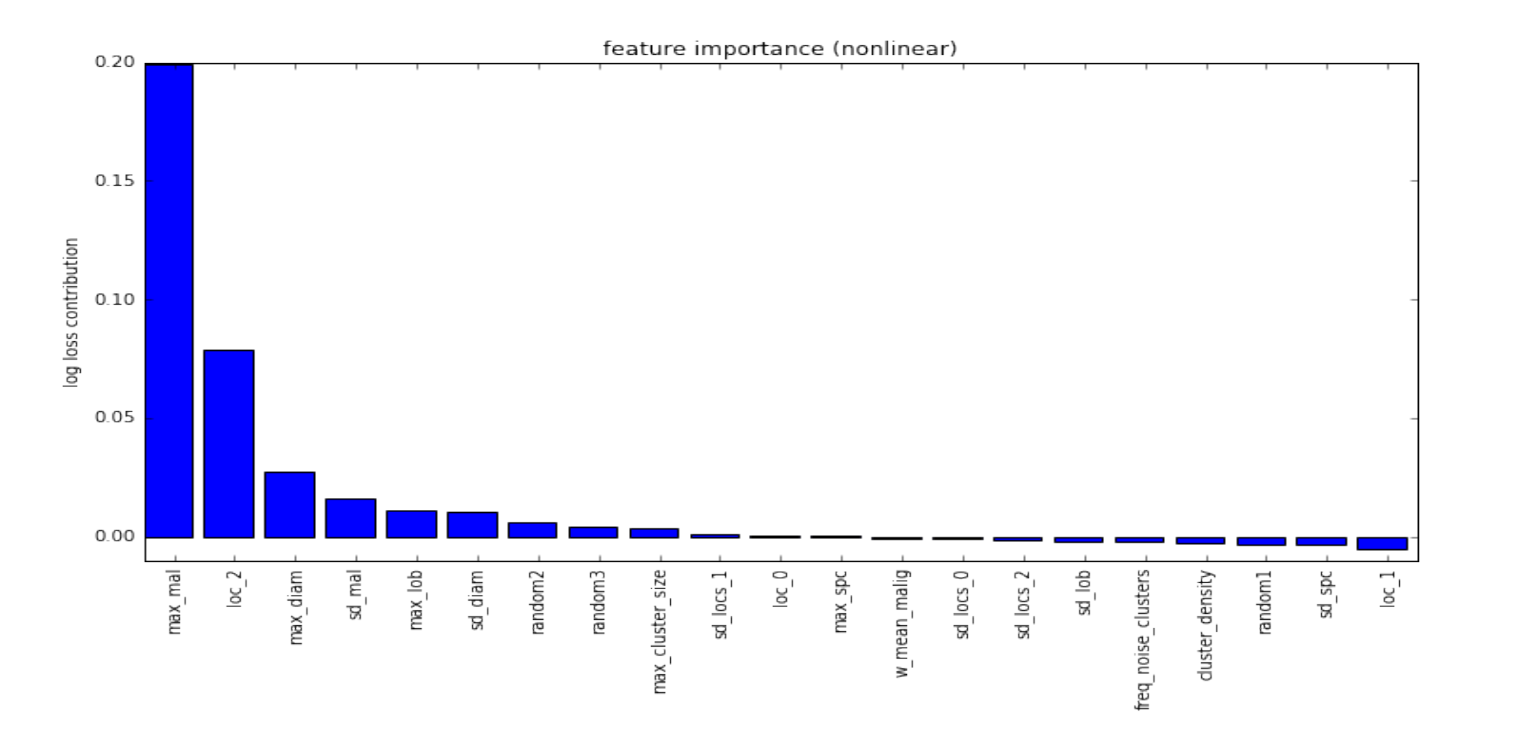
上图为特征重要性图。这18个特征被输入至“微小非线性”分类器中用于预测最终的诊断结果。分类器由L1-penalized逻辑回归模型组成以及一个极限随机树模型用于拟合线性模型的残差。附加特征——Julian的肿块(mass)检测模型的输出,用于预测患者肺部“异常肿块(mass)”的数量。
神经网络模型结构和训练细节
神经网络模型的输入数据为 的CT,训练数据集包含了LUNA16的带注释的数据和LUNA16的假阳性数据。模型由5个卷积块(conv block)组成,以及全局最大池化层和带softplus激活函数的非负回归层。为了模型能在不同尺度上捕获信息,原始输入通过降采样(downsample)的方式输入至模型的每一层。由于模型的目标为非负的,因此使用softplus激活函数。3D卷积大小为 ,池化层采用步长为2的 的kernel。在每次卷积和池化后进行批量归一化,随后进行降采样处理。模型使用75%的数据用于训练,25%的数据用于测试,大多数模型使用Leaky ReLU激活函数。其中,用于检测异常的模型使用90%的非结节数据和10%的结节数据训练,而用于预测结节属性的模型与此相反。
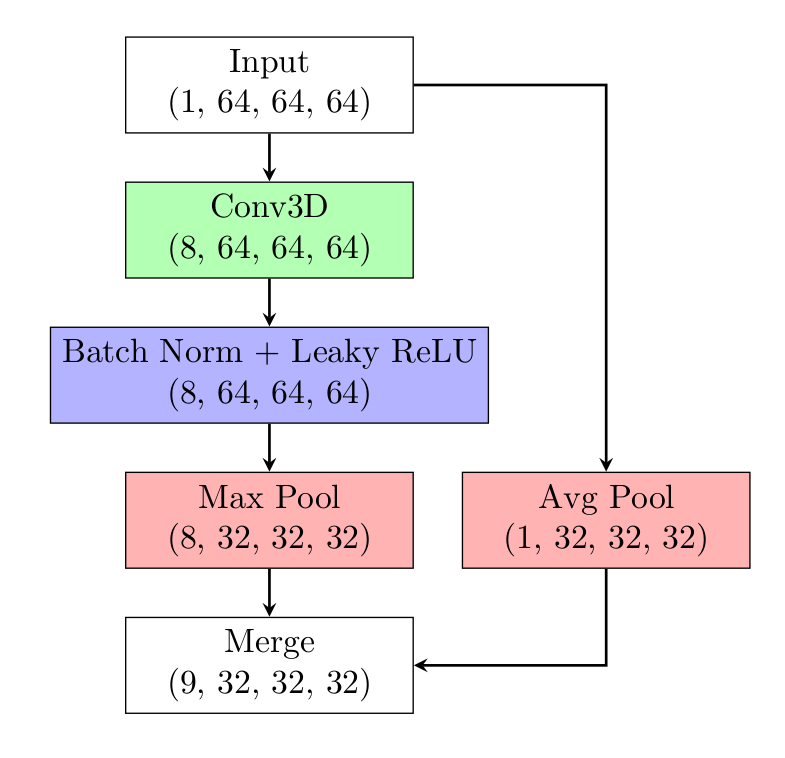
上图为一个卷积块(conv block)的结构。
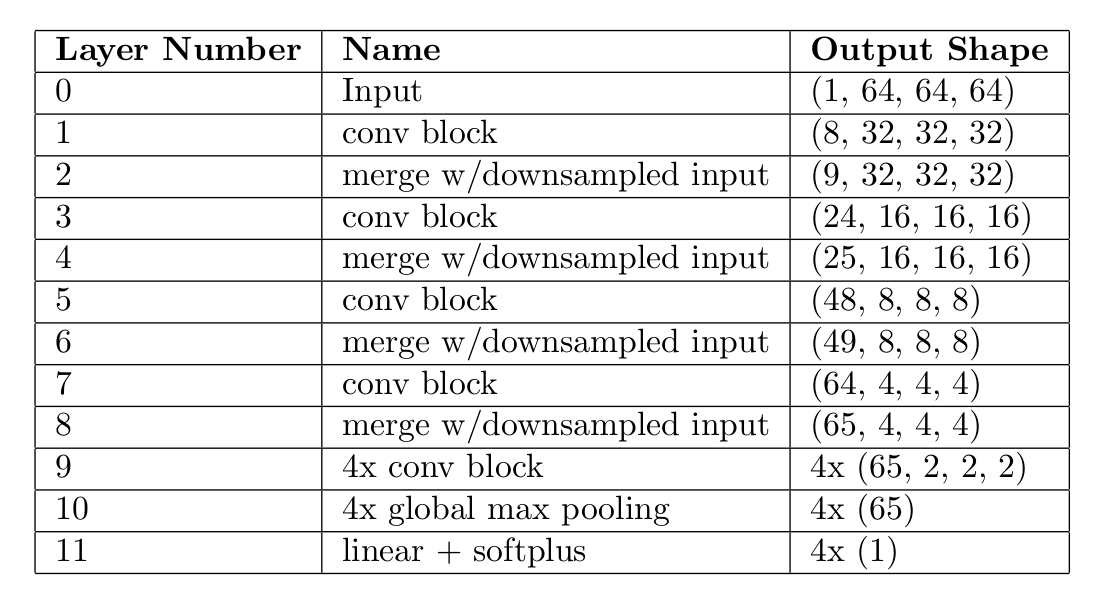
上表为重要结节属性的结构。其中,”4x“表示该层的4个并行副本,对于多输出模型中的每个输出都有一个副本。
数据增强
由于数据集中只含有1200个正例(来自LUNA16数据集中的注释结节),因此使用数据增强操作增加数据集的样本数,利用3D空间的对称性,使用无损和有损的方式进行数据增强。例如: 增量的随机旋转,随机转置,少量随机缩放,轴随机重排和小程度( )的随机旋转。由于实时有损增强计算量过大,因而通过在并行过程中不断重建不同版本的训练集实现。每隔几个时期(epoch),训练集都会加载一个新增加的版本。
模型训练
使用递进学习(curriculum learning)加速模型的训练。由于使用了全局最大池化层,其能处理大小为 以上的输入数据。因此,首先使用大小为 的数据进行训练,其中批次大小为128,对大约2000个参数进行了更新;然后使用大小为 的数据进行训练,其中批次大小为64,对大约6000个参数进行了更新。训练的前25个时期(epoch)随机使用75%的结节数据,后5个使用全部数据集。学习率初始化为0.1,每隔几个时期(epoch)逐步降低,但在最后几个训练时期(epoch)采用 的学习率。模型使用了keras库中的NAdam优化器,但并未过多尝试优化算法。