文章目录
1 概述
本节展示在1D时间序列上的低星滤形 (lower star filtration) 和子阶集滤形,其是描述时间序列上关键点 (局部最小或者最大) 的一种方式。直观上,我们将计算时间序列图上方“水池上升”时得到的连通分量的 H 0 H_0 H0。当一个新的池子在局部最小值形成时,其对应于初始;反之在局部最大处形成时,其对应于消亡。在这个过程中,我们将展示如何使用沿稀疏距离矩阵对角线的非零元素来设置顶点初始时间。
2 一些必须的库
import numpy as np
import matplotlib.pyplot as plt
import time
from scipy import sparse
from ripser import ripser
from persim import plot_diagrams
3 测试用的时间序列
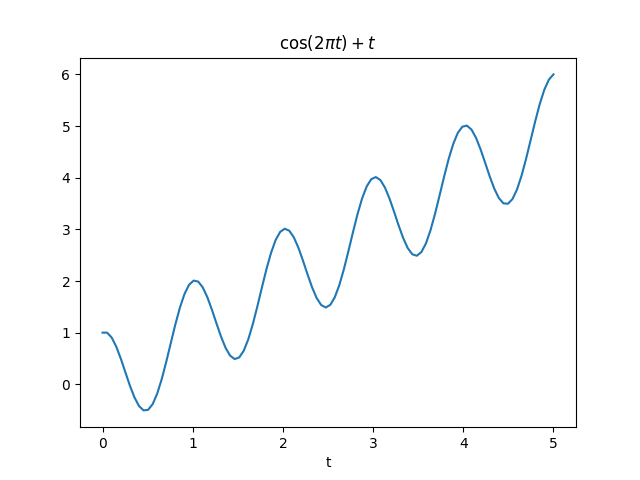
N = 100
t = np.linspace(0, 5, N)
x = np.cos(2*np.pi*t) + t
plt.plot(t, x)
plt.title("$\\cos(2 \pi t) + t$")
plt.xlabel("t")
plt.show()
输出如下:
4 子阶集滤形
稀疏距离矩阵将用于子阶集滤形。时间序列中的每个点都有一个顶点,初始在时间序列中该点的高度。然而,边界连接时间序列中的邻接点,并且它们添加的高度等于它们连接的顶点的两个高度中较大的一个:
# 在邻接点之间添加边界,其中的距离值等于它们连接的顶点中两个高度中较大的那一个
I = np.arange(N-1)
J = np.arange(1, N)
V = np.maximum(x[0:-1], x[1::])
# 研究距离矩阵的对角线添加顶点初始时间
I = np.concatenate((I, np.arange(N)))
J = np.concatenate((J, np.arange(N)))
V = np.concatenate((V, x))
# 创建稀疏距离矩阵
D = sparse.coo_matrix((V, (I, J)), shape=(N, N)).tocsr()
dgm0 = ripser(D, maxdim=0, distance_matrix=True)['dgms'][0]
dgm0 = dgm0[dgm0[:, 1]-dgm0[:, 0] > 1e-3, :]
allgrid = np.unique(dgm0.flatten())
allgrid = allgrid[allgrid < np.inf]
xs = np.unique(dgm0[:, 0])
ys = np.unique(dgm0[:, 1])
ys = ys[ys < np.inf]
plt.figure(figsize=(12, 6))
ylims = [-1, 6.5]
plt.subplot(121)
plt.plot(t, x)
ax = plt.gca()
ax.set_yticks(allgrid)
ax.set_xticks([])
plt.ylim(ylims)
plt.grid(linewidth=1, linestyle='--')
plt.title("$\\cos(2 \pi t) + t$")
plt.xlabel("t")
plt.subplot(122)
ax = plt.gca()
ax.set_yticks(ys)
ax.set_xticks(xs)
plt.ylim(ylims)
plt.grid(linewidth=1, linestyle='--')
plot_diagrams(dgm0, size=50)
plt.title("Persistence Diagram")
plt.show()
输出如下:
