使用enrichKEGG做通路富集分析时,一直报错:显示No gene can be mapped....
k <- enrichKEGG(gene = gene, organism = "hsa", pvalueCutoff =1, qvalueCutoff =1)
但是之前用同样的基因做分析是能够成功地富集到通路,即便是网上的数据会更新,也不可能变化的这么大吧,我换了一组基因,出现相同的问题。去B站视频教程的评论去找答案,发现有小伙伴在前几天刚刚评论说出现和我一样的问题,可能这个问题是刚新出的,网上也一直没找到解决这个问题的方法。
我考虑是不是因为clusterProfiler版本的原因,但我没有立刻更换clusterProfiler版本。(先留个扣,到底是不是版本的原因?)
看到了网上很多在本地进行富集分析的教程,于是,便冒着今天科研进度为零的风险,大胆尝试一下把数据下载到本地,进行通路富集分析,因为这么做也有个优点,就是数据库在本地,不会随着网上的更新而导致富集结果的更新,因为这样避免了前期的实验在后期复现不出来的麻烦。
参考了这个方法:
先去上述官方地址找到自己研究的物种在KEGG数据里的3字符缩写,比如:我研究的是人类和玉米,缩写是'hsa'和'zma'。
#安装Y叔的包,
#安装创建KEGG数据库的包的包
remotes::install_github("YuLab-SMU/createKEGGdb")
#创建自己的物种的包create_kegg_db,会自动创建名称为KEGG.db_1.0.tar,gz的包。物种名称的简写,在
createKEGGdb::create_kegg_db('zma') #人类是'hsa'
#安装这个包(默认的包的路径在当前工作目录,根据实际情况修改路径)
install.packages("~/KEGG.db_1.0.tar.gz",repos=NULL,type="source")
但是在第二步时出现了小插曲,应该是说我的clusterProfiler有问题,我去安装了最新版本的clusterProfiler,clusterProfiler下载链接,在导入时又出现了麻烦,说最新版的clusterProfiler引入了DOSE,我的DOSE版本太低,我用BiocManager安装DOSE,但是BiocManager安装的DOSE依旧不符合要求,我考虑是否是BiocManager版本太低,又更新了BiocManager在安装DOSE,还是不行。迫不得已,我直接更新了R,从4.2.0更新了4.2.2,重新安装BiocManager、DOSE依旧解决不了问题。机缘巧合下(之前一直用某度,完全找不到官方信息,使用Bing浏览器搜到了这个,仿佛打开了新世界的大门),我找到了官方的DOSE,变下载安装包,在RStudio中手动安装,安装成功,继续导包clusterProfiler,又出现了一下包的关联问题,像GOSemSim, HDO, DO等,用同样的方法去官网下载安装包手动安装,因为BiocManager更新迟后,使用BiocManager不能安装最新版。后面甚至是JSON都需要安装,最后,clusterProfiler导包成功了!!!!
执行第二步和第三步,也都成功了!
还需要把“~/KEGG.db_1.0.tar.gz”手动安装,最后成为KEGG.db包,才可以使用library导入。
附上运行成功的代码(注意本地分析的话use_internal_data = TRUE,线上分析是默认的FALSE):
library("clusterProfiler")
library("org.Hs.eg.db")
library("enrichplot")
library("ggplot2")
library("pathview")
library("ggnewscale")
library("DOSE")
library(stringr)
library(AnnotationHub) #library导入需要使用的数据包
library(eoffice)
library('KEGG.db')
# 以上有些包在共享的代码中没用到,是后续画图用的
# 把SYMBOL转成gene_id
EG2SYMBOL=toTable(org.Hs.egSYMBOL)
geneLists=read.table("data/gene_module_5.txt",sep="\t",check.names=F,header=F)
colnames(geneLists) <- c('symbol')
results=merge(geneLists,EG2SYMBOL,by='symbol',all.x=T) # 合并两个数据框
gene <- results$gene_id # 选择一列
# 进行通路分析
k <- enrichKEGG(gene = gene, organism = "hsa", pvalueCutoff =1, qvalueCutoff =1, use_internal_data = TRUE)
# 强制转成数据框,便于查看
KEGG=as.data.frame(k)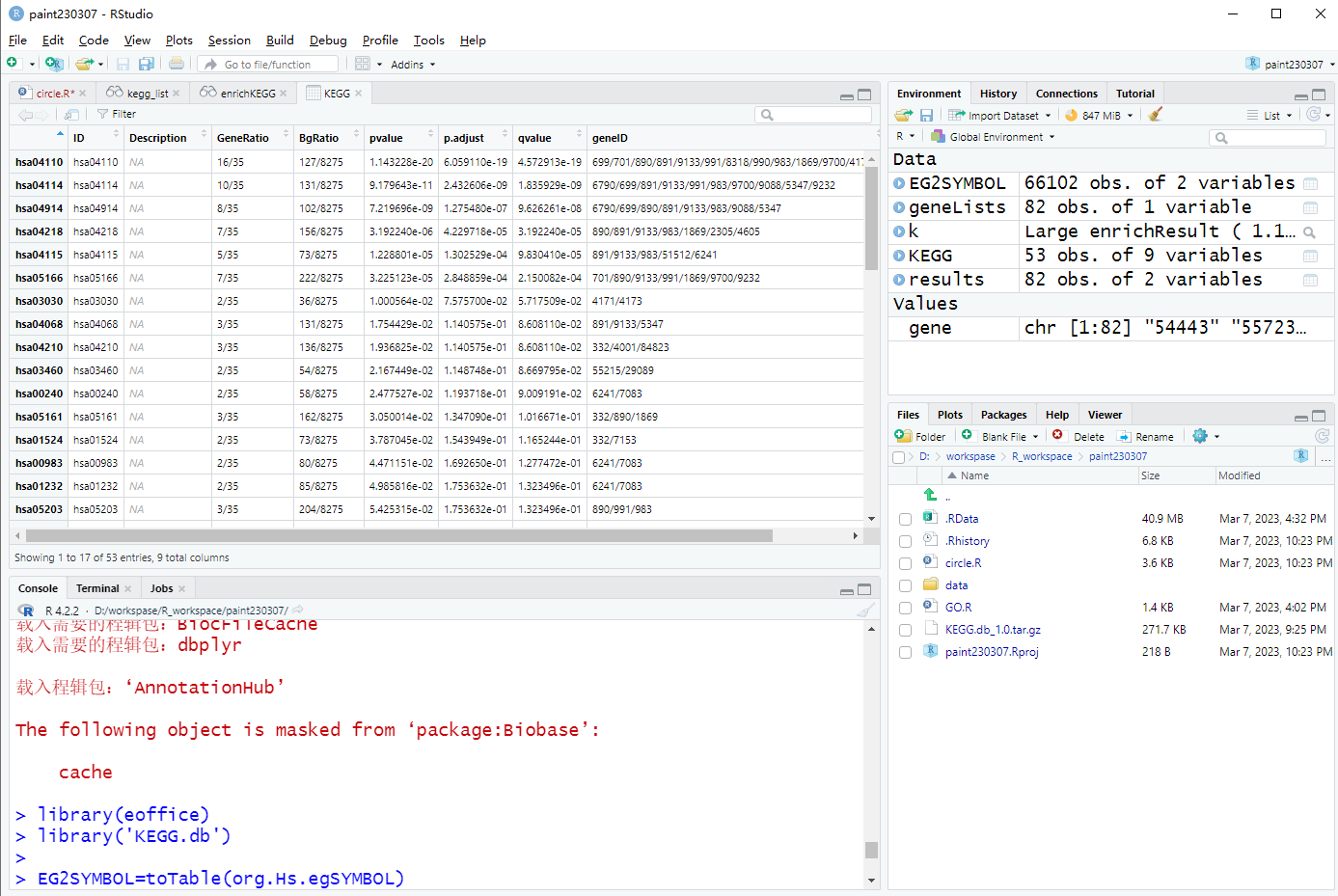
本地通路富集分析完成!!!
下面看一下是不是clusterProfiler版本的原因,我运行
k <- enrichKEGG(gene = gene, organism = "hsa", pvalueCutoff =1, qvalueCutoff =1)仍然报错:

那就不是clusterProfiler版本的原因了,是什么也不重要了,过些日子说不定就好了。
知识储备有限,描述不专业,敬请见谅!!!