1.解决问题
UNet++是通过设计一个具有嵌套和密集跳过连接的架构而开发的一种改进的Unet。然而,它只是融合了同一层级的特征图,并没有从全面探索充分的信息,仍然有很大的改进空间。
2.核心思想
不同尺度的特征地图探索了独特的信息。底层详细特征图捕捉了丰富的空间信息,突出了器官的边界;而高级语义特征图则包含了位置信息,它们位于器官所在的位置。然而,当逐渐向下和向上采样时,这些精细的信号可能会逐渐被稀释。为了充分利用多尺度特性,作者提出了一种新的基于u形的架构,名为UNet 3+,重新设计了编码器和解码器之间的相互连接以及解码器之间的内部连接,以从全尺度捕获细粒度细节和粗粒度语义。为了进一步从全尺度聚合特征图中学习层次表示,每边输出都连接一个混合损失函数,这有助于精确分割,特别是在医学图像体积中不同尺度出现的器官。除了提高精度外,我们还表明,所提出的UNet 3+可以减少网络参数,以提高计算效率。同时扩展了一个分类任务来预测输入图像是否有器官,为分割任务提供了指导。
3.主要贡献
i)设计一种新的UNet 3+,通过引入全尺度跳跃连接,充分利用多尺度跳跃特征,结合全尺度特征图的高级语义,但参数较少;(ii)开发深度监督,从全尺度聚合特征图学习层次表示,优化混合损失函数以增强器官边界;(iii)提出一种分类引导模块,通过与图像级分类联合训练来减少非器官图像的过度分割;(iv)对肝脏和脾脏数据集进行了广泛的实验,其中UNet 3+在一些基线上产生了一致的改进。
4. METHODS
4.1. Full-scale Skip Connections
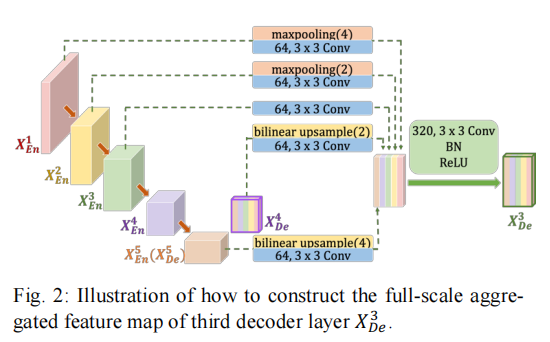
为了弥补UNet和UNet++中的缺陷,UNet 3+中的每个解码器层都包含了来自编码器的小、相同尺度的特征图,以及来自解码器的大尺度特征图,它们在全尺度上捕获细粒度细节和粗粒度语义。
如图所示,由
以及
上采样融合得到,首先,需要将5层特征图的通道数量统一,做法是用64个大小为3*3的滤波器进行卷积。然后,进一步对5个尺度的连接特征图进行了特征聚合机制,该机制由320个大小为3×3的过滤器、一个批归一化和一个ReLU激活函数组成。形式化表达如下:i表示下采样的次数,N表示Ecoder的层数

其中,函数∁(∙)表示卷积操作,ℋ(∙)通过卷积、批量归一化和ReLU激活函数来实现特征聚合机制D(.)和U(∙)分别表示上采样和下采样操作,[∙]表示连接。
值得一提的是,UNet 3+在更少的参数下更有效。在编码器子网络中,UNet、UNet++和UNet 3+共享相同的结构,其中有
个通道。对于解码器,UNet中的特征映射的深度与编码器是对称的,因此
也有3
个通道。U-net 3+第i个decoder参数的数量
![]() :
:

表示卷积核大小,D(∙)表示节点的深度。
U-net ++第i个decoder参数的数量 ![]()

因此,U-net 3+的参数量小于U-net++
2.2. Full-scale Deep Supervision
为了从全尺度聚合特征图中学习层次表示,在UNet 3+中进一步采用了全尺度深度监督。与在UNet++中生成的全分辨率特征图进行深度监督相比,提出的UNet 3+从每个解码器阶段产生一个侧输出,该输出由地面真实监督。为了实现深度监督,每个解码器阶段的最后一层被输入到一个普通的3×3卷积层中,然后是一个双线性上采样和一个s型函数。
为了进一步增强器官的边界,作者提出了一个多尺度结构相似性指数(MS-SSIM)[9]损失函数来为模糊边界分配更高的权值。因此,UNet 3+将密切关注模糊边界,因为区域分布差异越大,MS-SSIM值越高。从分割结果P和地面真实掩码G中裁剪出两个对应的N×N大小的补丁,可以记为![]()
![]() 和
和![]() 各自地p和g的MSSSIM损失函数定义为:
各自地p和g的MSSSIM损失函数定义为:

M表示层级的数量,表示对应的均值和方差 ,
和
定义了每个尺度中两个分量的相对重要性。两个小的常数C1= 0.01和C2 = 0.03(为了避免除以零的不稳定情况。在我们的实验中,我们根据[9]将量表设置为5。
通过结合焦点损失(![]() MS-SSIM损失
MS-SSIM损失![]() 和IoU损失(
和IoU损失(![]() )[11],作者定义的分割损失为:
)[11],作者定义的分割损失为:
![]()
2.3. Classification-guided Module (CGM)
在大多数医学图像分割中,在非器官图像中出现假阳性是一种不可避免的情况。它很可能是由于背景残留在较浅层的噪声信息引起的,导致了过分割的现象。为了实现更准确的分割,作者尝试通过添加一个额外的分类任务来解决这个问题,该任务被设计用于预测输入图像是否有器官。
如图3所示,经过一系列操作,包括辍学、卷积、最大池和s型运算后,从最深处![]() 产生一个二维张量,每个张量表示有/没有器官的概率。得益于最丰富的语义信息,分类结果可以进一步分两步引导每个分割的侧输出。首先,在argmax函数的帮助下,将二维张量转换为{0,1}的单一输出,表示有/没有器官。随后,我们将单个分类输出与侧边分割输出相乘。由于二值分类任务的简单性,该模块在二值交叉熵损失函数[12]的优化下轻松地获得了准确的分类结果,实现了纠正无器官图像过分割缺陷的指导。
产生一个二维张量,每个张量表示有/没有器官的概率。得益于最丰富的语义信息,分类结果可以进一步分两步引导每个分割的侧输出。首先,在argmax函数的帮助下,将二维张量转换为{0,1}的单一输出,表示有/没有器官。随后,我们将单个分类输出与侧边分割输出相乘。由于二值分类任务的简单性,该模块在二值交叉熵损失函数[12]的优化下轻松地获得了准确的分类结果,实现了纠正无器官图像过分割缺陷的指导。
3. EXPERIMENTS AND RESULTS
3.1. Datasets and Implementation
该方法在肝和脾两个器官上进行了验证。肝脏分割的数据集来自ISBI LiTS 2017挑战。它包含131张腹部3DCT扫描,其中103卷和28卷分别用于训练和测试。来自医院的脾脏数据集通过了伦理批准,包含40个和9个用于训练和测试的CT体积。为了加快训练速度,输入图像有三个通道,包括要分割的切片和上、下切片,分别裁剪到320×320。利用随机梯度下降来优化网络,它的超参数被设置为默认值。每个病例均以Dce系数作为评价指标。

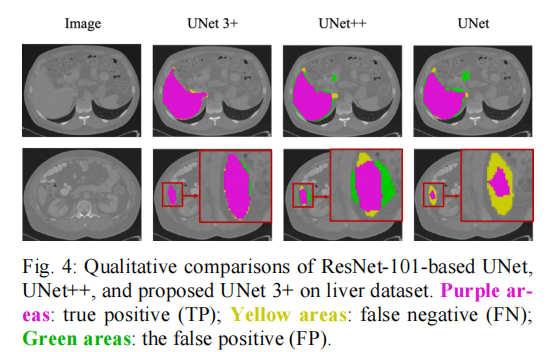
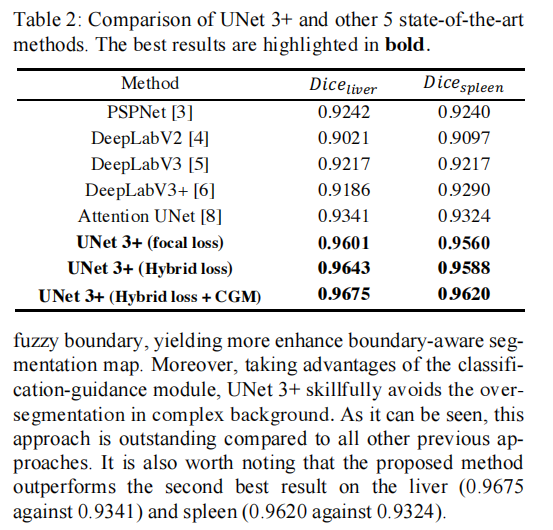
3.3. Comparison with the State of the Art
PSPNet [3],DeepLabV2 [4],DeepLabV3 [5],DeepLabV3+ [6]和Attention UNet [8]。