UNET 3+: A FULL-SCALE CONNECTED UNET FOR MEDICAL IMAGE SEGMENTATION
UNET 3+:一种用于医学图像分割的全尺寸互联UNET
Abstract
近年来,基于深度学习的语义分割受到越来越多的关注。UNET是一种编解码器结构的深度学习网络,广泛应用于医学图像分割。多尺度特征组合是实现精确分割的重要因素之一。**UNET++是通过设计具有嵌套和密集跳跃连接的体系结构而开发的一种改进的UNET。**然而,它没有从完整的尺度上发掘足够的信息,仍有很大的改进空间。**在本文中,我们提出了一种新的UNET3+,它利用了全尺寸跳跃连接和深度监控的优点。全尺度跳跃连接结合了不同尺度特征图的低层细节和高层语义,而深度监督则从全尺度聚合特征图中学习层次表示。建议的方法尤其有利于出现在不同尺度上的器官。除了提高精度外,建议的UNET3+还可以减少网络参数以提高计算效率。在此基础上,提出了一种混合损失函数,并设计了分类引导模块,增强了非器官图像中的器官边界,减少了过度分割,得到了更准确的分割结果。**在两个数据集上验证了该方法的有效性。代码可在github.com/ZJUGiveLab/UNET-Version上找到
Introduction
为了减少UNET中语义不同特征与普通跳过连接的融合,UNET++[7]通过引入嵌套的密集跳过连接进一步加强了这些连接,目的是缩小编码器和解码器之间的语义鸿沟。
正如许多分割研究[1-7]所证明的那样,不同尺度的特征地图探索不同的信息。低层细节特征图捕捉了丰富的空间信息,突出了器官的边界;高层语义特征图体现了位置信息,定位了器官所在的位置。然而,当逐步向下和向上采样时,这些精致信号可能会逐渐被稀释。为了充分利用多尺度特性,我们提出了一种新的基于U-Shape的体系结构UNET3+。在该体系结构中,我们重新设计了编码器和解码器之间的互连以及解码器之间的内部连接,以获取全尺度上的细粒度细节和粗粒度语义。为了进一步从全尺度聚集特征图中学习层次表示,每一侧输出都与混合损失函数相连接,该函数有助于准确分割,特别是对于医学图像体积中以不同尺度出现的器官。除了精度的提高外,我们还证明了所提出的UNET3+可以减少网络参数以提高计算效率。
为了满足医学图像更精确分割的要求,我们进一步研究了如何有效地降低非器官图像中的假阳性。现有的方法通过引入注意机制[8]或在推理时进行预定义的精化方法(如CRF[4])来解决这个问题。与这些方法不同的是,我们扩展了一个分类任务来预测输入图像是否有器官,为分割任务提供了指导。
综上所述,我们的主要贡献有四个方面:(I)设计了一种新的UNET3+,它通过引入全尺度跳跃连接来充分利用多尺度特征,该连接结合了全尺度特征图中的低层细节和高层语义,但参数更少;(Ii)开发了一种深度监督来从全尺度聚集特征图中学习分层表示,它优化了混合损失函数来增强器官边界;(Iii)提出了分类制导模块,通过联合训练来减少非器官图像的过度分割。(Iv)对肝脏和脾脏数据集进行广泛的实验,在这些实验中,UNET 3+在一些基线上产生了一致的改善。
Methods
图1给出了UNET、UNET++和建议的UNET 3+的简化概述。与UNET和UNET++相比,UNET 3+通过重新设计跳跃连接以及利用全面的深度监控结合了多尺度特征,提供的参数更少,但生成的位置感知和边界增强型分割图更准确。
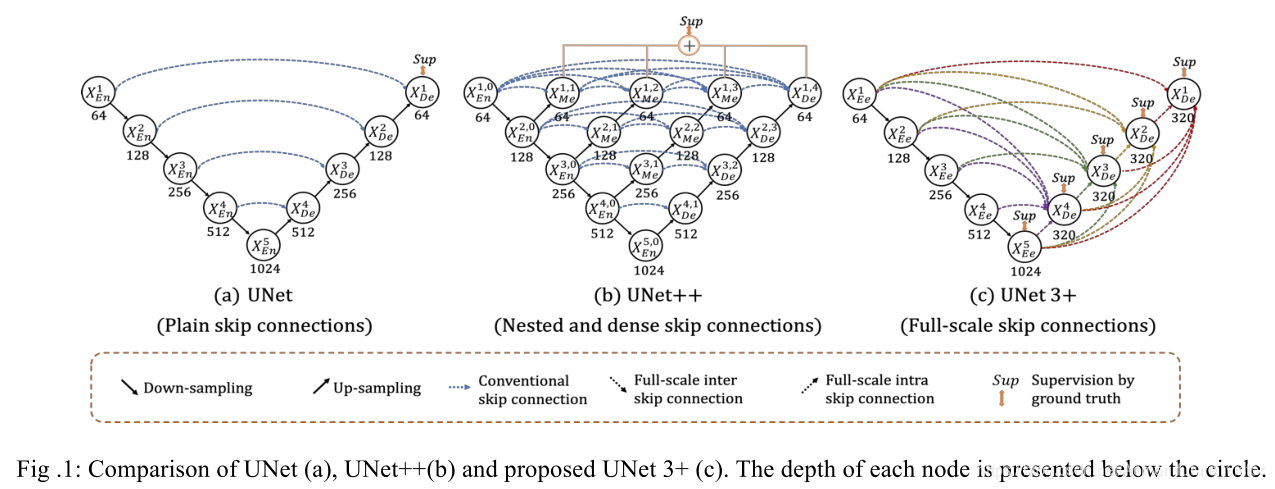
Full-scale Skip Connection(全尺寸跳跃连接)
所提出的全尺度跳跃连接转换了编码器和解码器之间的互连以及解码器子网络之间的内联。无论是普通连接的UNET,还是嵌套密集连接的UNET++,都缺乏从全尺度探索足够的信息,无法明确了解器官的位置和边界。为了弥补UNET和UNET++的不足,UNET 3+中的每个解码层都融合了编码器的小尺度特征图和等尺度特征图,以及解码器的大尺度特征图,能够全面捕捉细粒度细节和粗粒度语义。
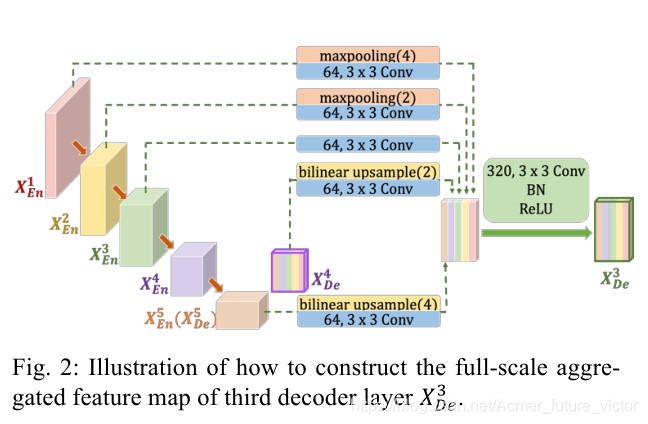
作为一个例子,图2说明了如何构建XDe3的特征映射。与UNET相似,解码器直接接收来自相同尺度编码层XEn3的特征地图。与UNET相比,一组编码-解码器间跳跃连接通过应用非重叠的最大池化操作来传递来自较小规模编码层XEn1和XEn2的低级别详细信息;而一串解码器内部跳跃连接通过利用双线性插值来传输来自较大规模解码器层XDe4和XDe5的高级语义信息。有了五张相同分辨率的特征图,我们需要进一步统一通道的数量,同时减少多余的信息。我们认为,用64个大小为3×3的滤波器进行卷积是一个令人满意的选择。为了实现浅层精细信息与深层语义信息的无缝融合,我们进一步从5个尺度对拼接后的特征图进行了特征聚合,该机制由320个大小为3×3的过滤器、批量归一化和RELU激活函数组成。形式上,我们将跳跃连接表示为:设i沿编码器索引下采样层,N表示编码器的总数。由XDei表示的特征地图堆栈计算为
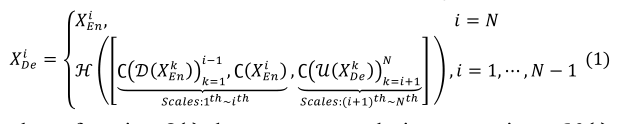
其中,函数∁(∙)表示卷积运算,ℋ(∙)利用卷积之后的批归一化和RELU激活函数来实现特征聚集机制。D(∙)和U(∙)分别表示上采样和下采样操作,[∙]表示级联。
值得一提的是,我们建议的UNET 3+参数更少,效率更高。在编码子网络中,UNET、UNET++和UNET 3+具有相同的结构,其中Xeni有32×2^i
个通道。在解码器方面,UNET的特征映射深度与编码器是对称的,因此XDei也有32×2^i 个通道。UNET的第i级解码器的参数数(P^iU-de)可以计算为
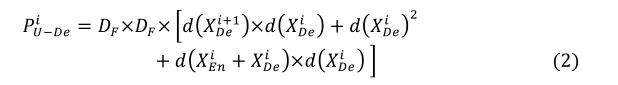
其中,DF是卷积核大小,d(∙)表示节点的深度。当涉及UNET++时,它利用沿每个跳跃路径的密集卷积块,其中P^I U^++ -De可以计算为

可以看出,P^i U^++ -De,大于P^i U-de。而在UNET 3+中,每个解码器特征映射是从N个尺度导出的,产生64×N个通道。P^i U^3+ -De,可以计算为
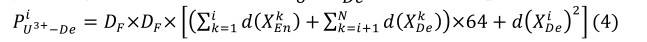
为了减少通道,UNET 3+的参数比UNET和UNET++少。
Full-scale Deep Supervision(全面深度监管)
为了从全尺度聚合特征图中学习层次表示,UNET3+进一步采用了全尺度深度监督。与UNET++中对生成的全分辨率特征图进行深度监督相比,所提出的UNET+3+算法在每个解码器阶段产生一个侧面输出,该侧面输出由ground truth监督。为了实现深度监督,每一解码器级的最后一层被馈入平坦的3×3卷积层,然后是双线性上采样和Sigmoid函数。
为了进一步增强器官边界,我们提出了一种多尺度结构相似性指数(MS-SSIM)[9]损失函数来赋予模糊边界更高的权重。受益于此,UNET3+将密切关注模糊边界,因为区域分布差异越大,MS-SSIM值越高。从分割结果P和地面真实掩码G中裁剪出两个对应的N×N大小的块,其可以表示为p={pj:j=1,……,N2}和g={gj:j=1,……N2}。p和g的MSSSIM损失函数定义为
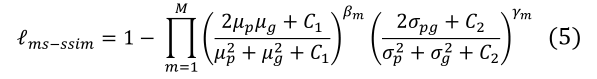

通过结合focal loss(ℓFL:)[10]、MS-SSIM损失(ℓms-ssim)和IOU损失(ℓiou)[11],我们提出了一种混合损失方法,用于pixel、patch和map三个层次的分割,能够捕捉到边界清晰的大尺度和细微结构。混合分段损失(ℓSEG)定义为
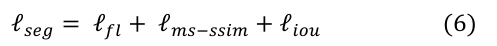
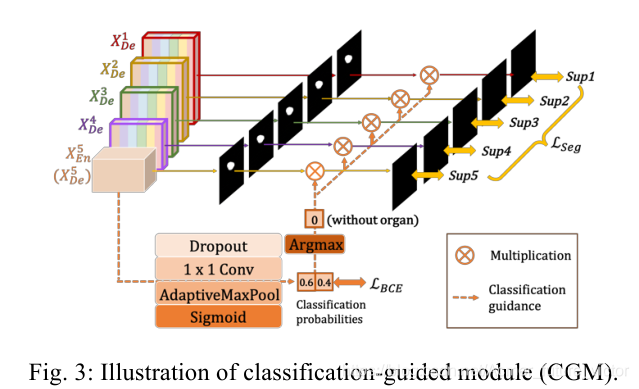
Classification-guided Module(CGM)
在大多数医学图像分割中,非器官图像出现假阳性是不可避免的。这很可能是由于背景噪声信息残留在较浅层,导致过分割现象所致。为了实现更准确的分割,我们试图通过增加一个额外的分类任务来解决这个问题,该任务是为了预测输入图像是否有器官而设计的。
如图3所示,在经过dropout、卷积、maxpool和sigmoid等一系列操作后,从最深层XEn5产生一个二维张量,每个张量代表有/没有器官的概率。得益于最丰富的语义信息,分类结果可以进一步分两步指导每个切分副输出。首先,在argmax函数的帮助下,将二维张量转化为{0,1}的单一输出,{0,1}表示有/没有器官。随后,我们将单分类输出与边分割输出相乘。由于二值分类任务简单,该模块在二值交叉熵损失函数[12]的优化下,毫不费力地获得了准确的分类结果,为弥补非器官图像过度分割的缺陷提供了指导。
Experiments and Results
Datasets and Implementation
该方法在肝脏和脾脏两个器官上进行了验证。肝脏分割的数据集来自ISBI LITS 2017挑战赛。它包含131张对比度增强的腹部三维CT扫描,其中103张和28张分别用于培训和测试。来自医院的脾脏数据集通过了伦理批准,包含40和9个CT卷用于培训和测试。为了加快训练速度,输入图像有三个通道,包括待分割的切片和上下切片,并将其裁剪为320×320。我们利用随机梯度下降法对网络进行优化,并将其超参数设置为默认值。以骰子系数作为每个病例的评价指标。
Comparison with UNet and UNet++
在本节中,我们首先将建议的UNET 3+与UNET和UNET++进行了比较。每种方法使用的损失函数都是focal loss。
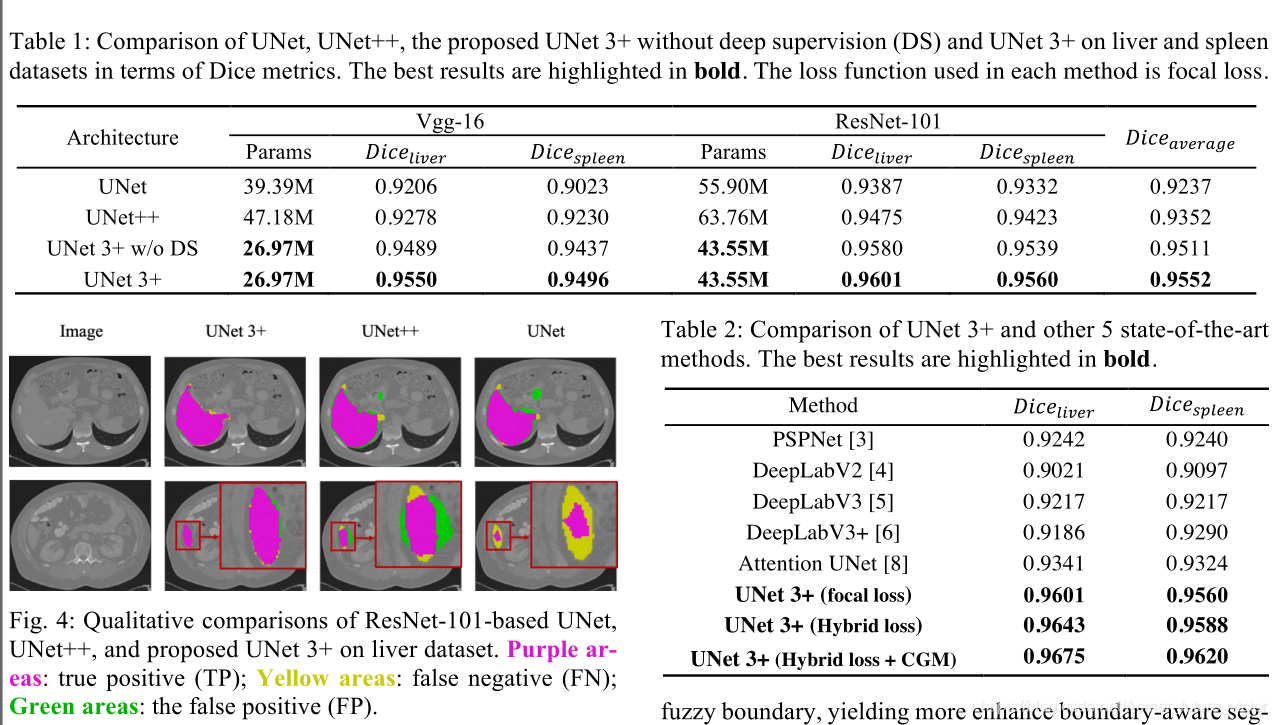
定量比较:基于VGG-16和ResNet-101的主干,表1比较了UNET、UNET++和建议的UNET 3+架构在肝脏和脾脏数据集的参数数量和分割精度方面的差异。可以看出,在没有深度监督的情况下,UNET 3+的性能超过了UNET和UNET++,在两个数据集上执行的两个主干之间获得了2.7和1.6个点的平均改进。考虑到肝脏和脾脏在CT切片上都有不同的尺度出现,UNET3+结合全面深度监测进一步提高了0.4分。
定性比较:图2显示了基于ResNet-101的UNET、UNET++和UNET 3+在肝脏数据集上的全面深度监控的分割结果。可以观察到,我们所提出的方法不仅可以精确地定位器官,而且可以产生连贯的边界,即使在小物体的情况下也是如此。
Comparison with the State of the Art
我们将我们基于ResNet-101的UNET 3+与几种最新的最先进方法进行了定量比较:PSPNet[3]、DeepLabV2[4]、DeepLabV3[5]、DeepLabV3+[6]和注意UNET[8]。值得一提的是,所有结果都直接来自单一模型测试,不依赖任何后处理工具。利用文中提出的损耗函数对所有网络进行了优化。
表2总结了定量比较结果。可以看出,提出的混合损失函数通过考虑pixel、patch、map级别的优化,极大地提高了性能。特别是,patch级别的MS-SSIM损失函数有助于将更高的权重分配给表2:UNET 3+和其他5种最先进方法的比较。最佳结果以粗体突出显示。模糊边界,生成更多增强的边界感知分割图。此外,利用分类引导模块的优势,UNET3+巧妙地避免了复杂背景下的过分割。由此可见,与以往的所有方法相比,这种方法是非常出色的。同样值得注意的是,所提出的方法在肝脏(0.9675对0.9341)和脾脏(0.9620对0.9324)上的表现优于第二好的结果。
Conclusion
为了最大限度地利用全尺度的特征地图,以较少的参数实现准确的分割和高效的网络结构,本文提出了一种具有深度监督的全尺度互联的UNET(UNET 3+)。进一步引入分类引导模块和混合损失函数,得到更精确的位置感知和边界感知的分割图。在肝脏和脾脏数据集上的实验结果表明,UNET3+超过了所有以前最先进的方法,突出了器官并产生了连贯的边界。