多元回归分析
多元回归分析预测法,是指通过对两个或两个以上的自变量与一个因变量的相关分析,建立预测模型进行预测的方法。当自变量与因变量之间存在线性关系时,称为多元线性回归分析。
多元线性回归的适用条件:
(1)自变量对应变量的变化具有显著影响。
(2)自变量与应变量间的线性相关必须是真实的,而非形式上的。
(3)自变量之间需有一定的互斥性。
(4)应具有完整的统计数据。
下面,我们就选取一个例子,用R实现多元线性回归模型。
训练数据:txt格式(行为样本,列为食物)。此例子中,我们选取10类食物作为特征,即自变量为10类食物。因变量为产生的蛋白质含量(Protein)。
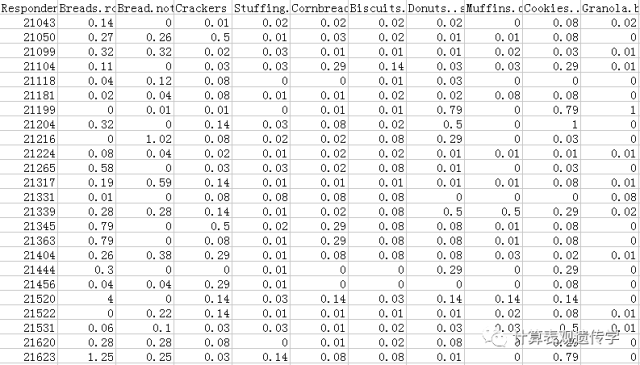
1 检查两两因变量之间的关系
x=read.table("test.txt",header=T,sep="\t",row.names=1)
x=as.matrix(x)
z=x[,1:10]
cor <- cor(z,use="pairwise", method="pearson")
cor
corrplot(cor)
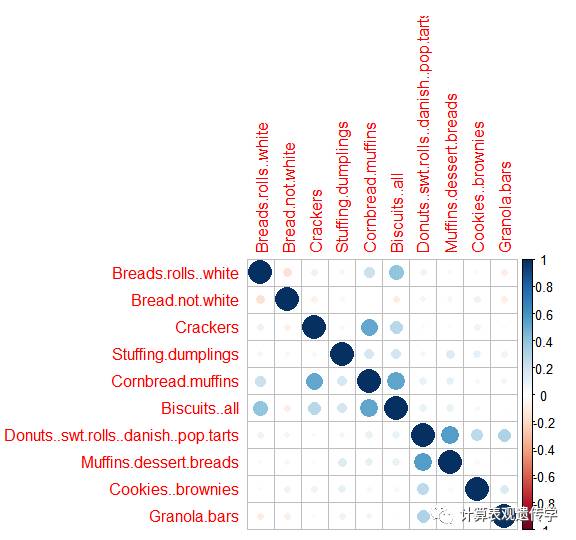
相关性图的展示使用的是corrplot包。具体步骤参见R语言绘图-相关矩阵的可视化。
从图中可以看出,各个自变量之间的线性程度不高,可以直接作为回归参数输入。因为如果自变量之间的线性关系超过自变量与因变量之间的线性关系,则回归模型稳定性受到破坏。
1 多元回归模型建立
接下来,我们用10个特征作为自变量,蛋白质含量作为因变量。建立多元线性回归模型。
y=x[,11]
lm.result <-lm(y~z)
summary(lm.result)

***表示极为显著,**表示高度显著,*表示显著。以上是模型检验的结果。因为我们的例子数据不全,所以大家可以用自己数据试一试,看看结果好不好。
1 残差分析
par(mfrow=c(2,2))
plot(lm.results)
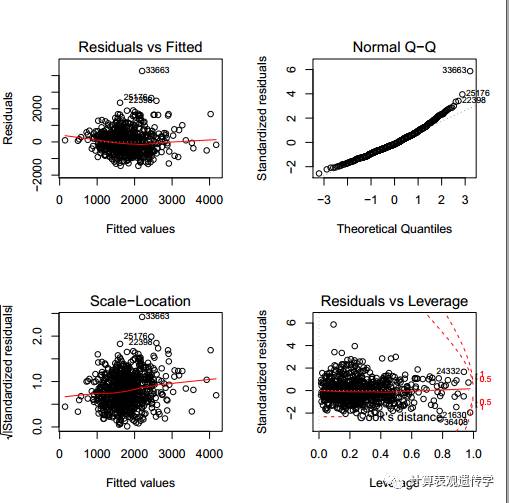
以上就是使用R实现多元线性回归的过程。
相关矩阵
在生物信息学分析中,经常会计算相关性矩阵。因此,相关矩阵的可视化很重要。许多文献中生动的相关性图形十分吸引眼球。
下面,我们介绍一种R语言中可视化相关性矩阵的方法(corrplot包)。
1 安装corrplot包
install.packages("corrplot")
library(corrplot)
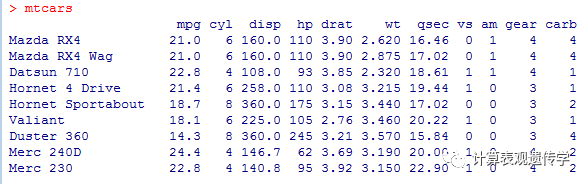
然后,我们使用R中的例子数据mtcars计算相关性矩阵。
data(mtcars)
mtcars
1 计算相关性矩阵
cor=cor(mtcars)
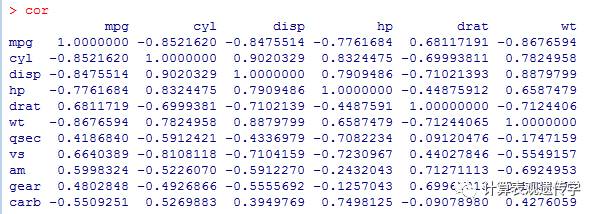
1 计算相关性矩阵
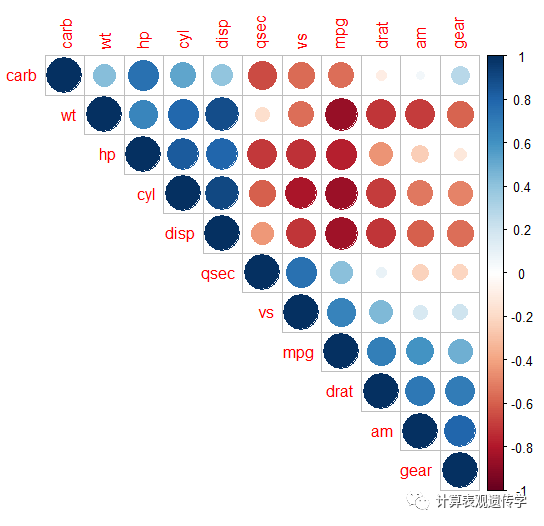
corrplot(cor, method="circle",type="full",order="hclust")
cor 相关性矩阵
method共有7种。"circle"(default), "square","ellipse", "number", "pie", "shade" and "color"
type 共有3种。"full" (default), "upper" or "lower"
order 分为以下几种。"original" for original order(default).
"AOE" for the angular orderof the eigenvectors.
"FPC" for the first principalcomponent order.
"hclust" for the hierarchicalclustering order.
"alphabet" for alphabeticalorder.
我们使用另外的参数:
corrplot(cor,method="color",type="upper", order="hclust",addCoef.col = "black")
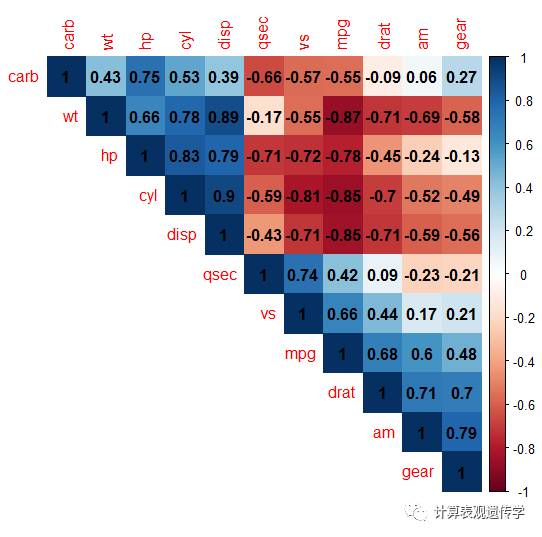
具体颜色,形状的改变,来获得更漂亮的图片就只需调节下参数了。