!!!!声明:不是原创,我只是方便自己学习,原文指路
NCBI-SRA数据库与EBI-ENA数据库
所有已发表文献中的高通量测序数据大多会上传到某个数据库中方便其他人的下载学习与再研究,这其中受众最广的自然是出身NCBI的SRA数据库。同时出身EBI的ENA数据库对于下载数据有很多便利之处,所以在具体下载文件之前先了解一下这两个数据库的情况。
NCBI与EBI同属于INSDC:International Nucleotide Sequence Database Collaboration,提交给所属三个数据库的数据是可以互通的。该架构内容具体如下:
- NCBI: National Center for Biotechnology Information
- EBI: European Bioinformatics Institute
- DDBJ:DNA Data Bank of Japan
SRA数据库: Sequence Read Archive,
- 是一个保存高通量测序数据以及比对信息和元数据(meta data)的数据库,所有已经发表的文献中的高通量测序数据基本上都会上传到该数据库中,这个数据库隶属于NCBI。
- SRA数据库的各种编号
元数据(meta data):是指与测序实验及其实验样品相关的数据, 如实验目的、 实验设计、 测序平台、 样本数据(物种, 菌株,个体表型等),在SRA数据库中,meta数据分如下层次来存储:
【1】研究课题(study):在 SRA 数据库中,研究课题的检索号(accession number)以前缀 DRP,ERP 或 SRP开头。
【2】样本信息(sample):样本的检索号以前缀 DRS,ERS 或 SRS 开头。 样本信息可以包括物种信息、 菌株(品系)信息、家系信息、表型数据、临床数据, 组织类型等。
【3】实验信息(experiment):实验的检索号以前缀 DRX,ERX或 SRX 开头。 实验是 SRA 数据库的最基本单元, 就像 PubMed 数据库的每一篇文献是 PubMed数据库的基本单元一样。 一个实验隶属于某个研究课题,对一个或多个样本进行测序,产生的测序数据以 runs 的形式存储于SRA数据库。
【4】序列数据:包括序列及其质量信息等,在 SRA 数据库中以 run 为单元存储。run 的检索号以前缀DRR,ERR 或 SRR 开头。
ENA数据库: European Nucleotide Archive
- 隶属于EBI,功能上应该是与SRA类似的,但是其搜索界面更加亲民,并且对于下载fastq文件以及检查下载数据完整性更加友好,所以强烈推荐优先使用。

-
ENA数据库的优势
【1】可以直接获取得到 fastq 文件
【2】使用ENA数据库还有一个优势是可以确认下载数据的完整性。生信数据的大体量性带来的下载时间长(期间网络万一不正常就会波动)可能会造成下载数据的缺失等问题,这些问题一般很难在获得数据的初期被发现。ENA数据库提供了md5码这种途径来检查数据的完整性。 -
ENA数据库使用
首先,在数据库页面右上角搜索栏输入目标SRA检索号,确认后稍等片刻可得结果页面

其次,点击选取 Experiment 可以获得该实验下所有的测序序列数据的信息
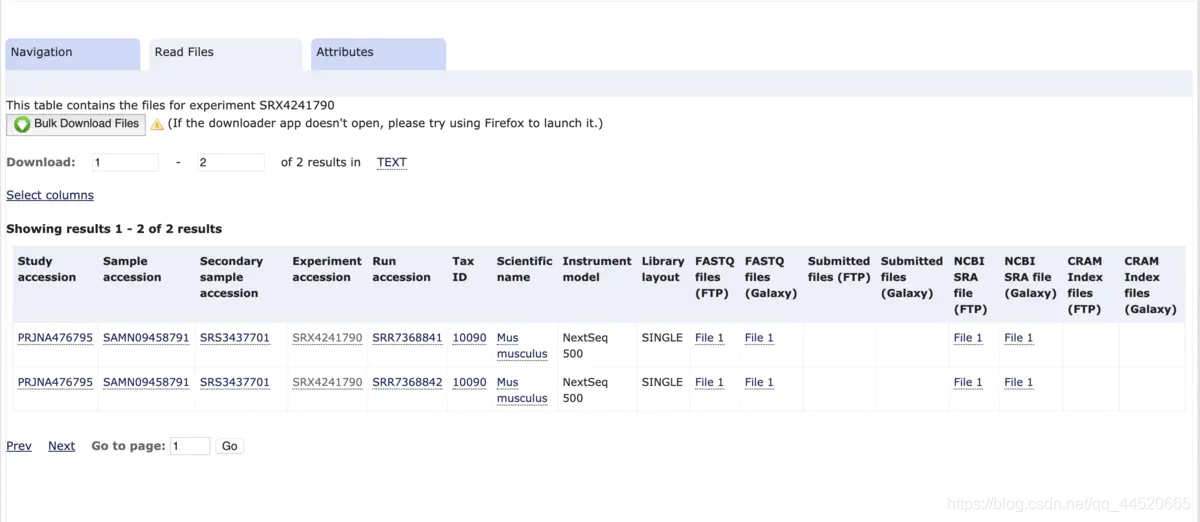
我们可以看到隶属于该实验的两个序列数据信息,并且可以在 FASRTQ files(FTP) 栏中获得直接下载 fastq 文件的FTP 地址。
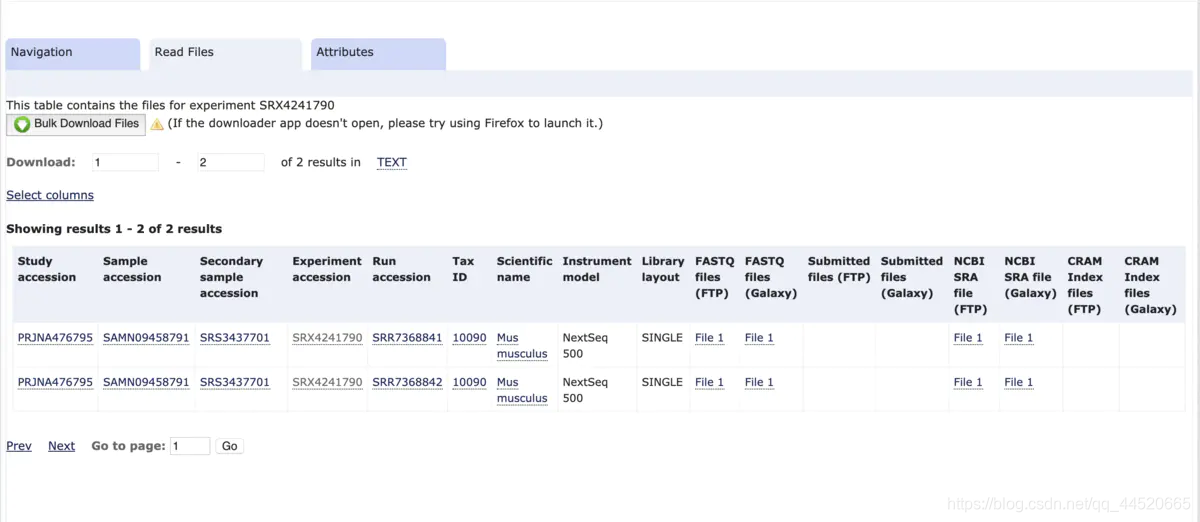
获取直接下载 fastq 文件的FTP地址