本文为《机器学习实战:基于Scikit-Learn和TensorFlow》的读书笔记。
中文翻译参考
- 特征维度太大,降维加速训练
- 能筛掉一些噪声和不必要的细节
更高维度的实例之间彼此距离可能越远,空间分布很大概率是稀疏的
1. 降维方法
1.1 投影
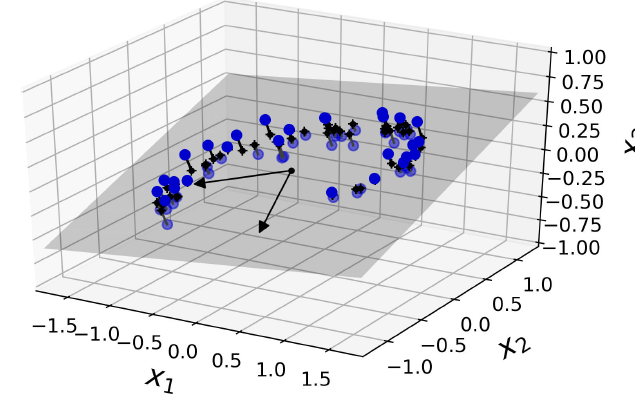
上图,三维空间中的点,都近似在灰色平面附近,可以投影到其上
- 投影并不总是最佳的方法
1.2 流行学习
Manifold Learning
假设:在流形的较低维空间中表示,它们会变得更简单(并不总是成立)
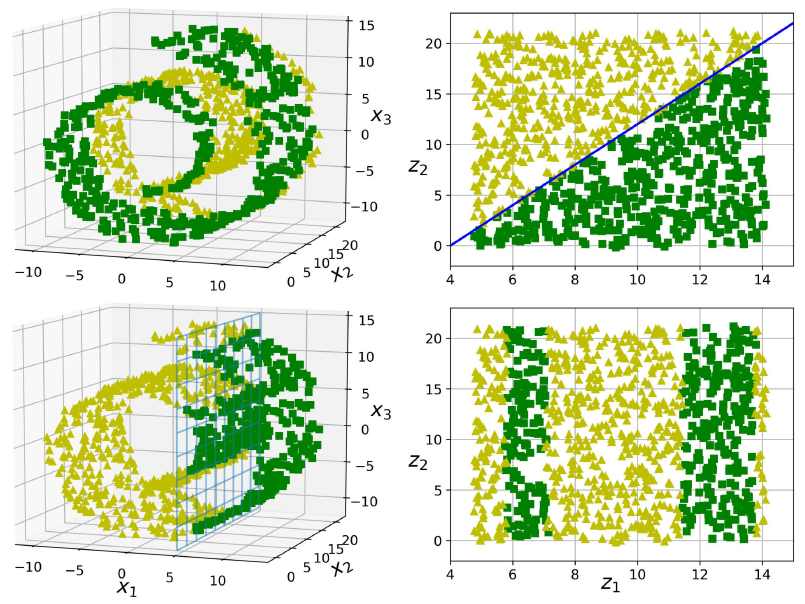
- 是否流行学习会更好,取决于数据集
- 第一行的情况,展开后更好分类,第二行的则,直接一个面分类更简单
2. 降维技术
2.1 PCA
《统计学习方法》主成分分析(Principal Component Analysis,PCA)笔记
目前为止最流行的降维算法
- 首先它找到接近数据集分布的超平面
- 然后将所有的数据都投影到这个超平面上
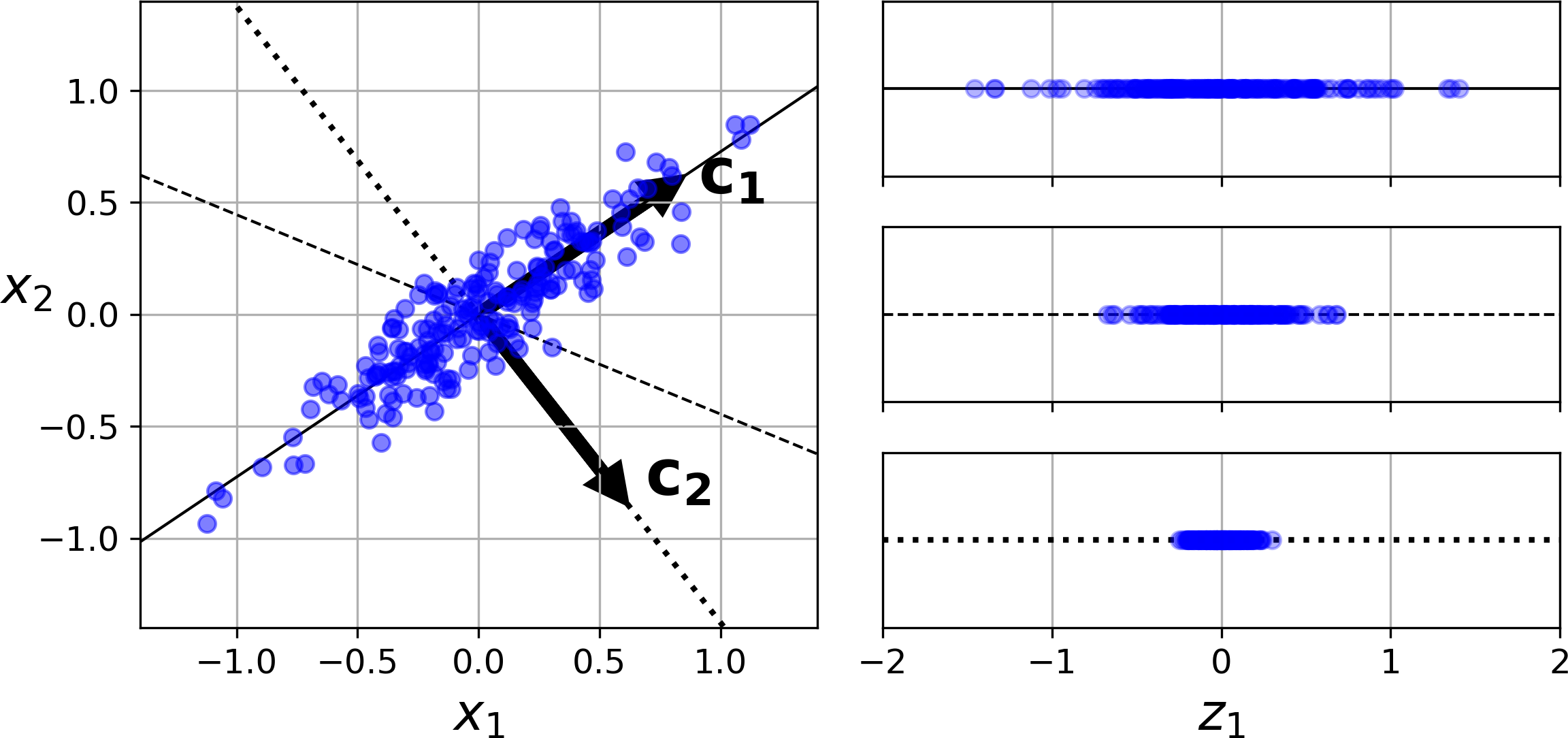
- 将数据投影到方差最大的轴(损失更少的信息)
- 或者理解为,点到该轴的均方距离最小
矩阵的 SVD分解 可以帮助找到主成分
X_centered=X-X.mean(axis=0)
U,s,V=np.linalg.svd(X_centered)
c1=V.T[:,0]
c2=V.T[:,1]
- sklearn 的 PCA 类使用 SVD 分解实现
from sklearn.decomposition import PCA
pca=PCA(n_components=2)
X2D=pca.fit_transform(X)
-
用
components_访问每一个主成分(它返回水平向量的矩阵,如果我们想要获得第一个主成分则可以写成pca.components_.T[:,0]) -
方差解释率(Explained Variance Ratio),它表示位于每个主成分轴上的数据集方差的比例
print(pca.explained_variance_ratio_)
array([0.84248607, 0.14631839])
看出第二个轴上的比例为14.6%
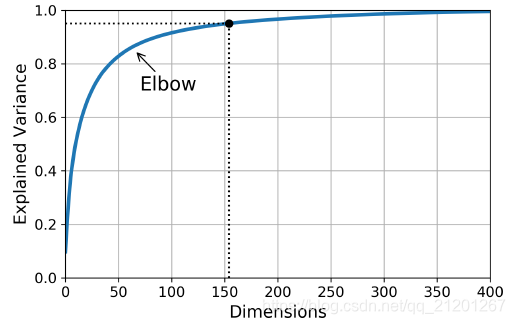
- 选择方差解释率占比达到足够(例如95%)的维度即可
pca=PCA()
pac.fit(X)
cumsum=np.cumsum(pca.explained_variance_ratio_)
d=np.argmax(cumsum>=0.95)+1
d为选取的主成分个数
pca=PCA(n_components=0.95)
设置为小数,表明保留的方差解释率为0.95
X_reduced=pca.fit_transform(X)
2.2 增量PCA
对大型数据集友好,可在线使用
from sklearn.decomposition import IncrementalPCA
n_batches=100
inc_pca=IncrementalPCA(n_components=154)
for X_batch in np.array_split(X_mnist,n_batches):
inc_pca.partial_fit(X_batch)
X_mnist_reduced=inc_pca.transform(X_mnist)
注意:array_split()将数据分开,partial_fit(),部分 fit
X_mm=np.memmap(filename,dtype='float32',mode='readonly',shape=(m,n))
batch_size=m//n_batches
inc_pca=IncrementalPCA(n_components=154,batch_size=batch_size)
inc_pca.fit(X_mm)
使用np.memmap方法,就好像文件完全在内存中一样,后面可跟fit
2.3 随机PCA
可以快速找到前 d 个主成分的近似值
- 它的计算复杂度是 ,而不是 ,所以当 d 远小于 n 时,它比之前的算法快得多
rnd_pca=PCA(n_components=154,svd_solver='randomized')
X_reduced=rnd_pca.fit_transform(X_mnist)
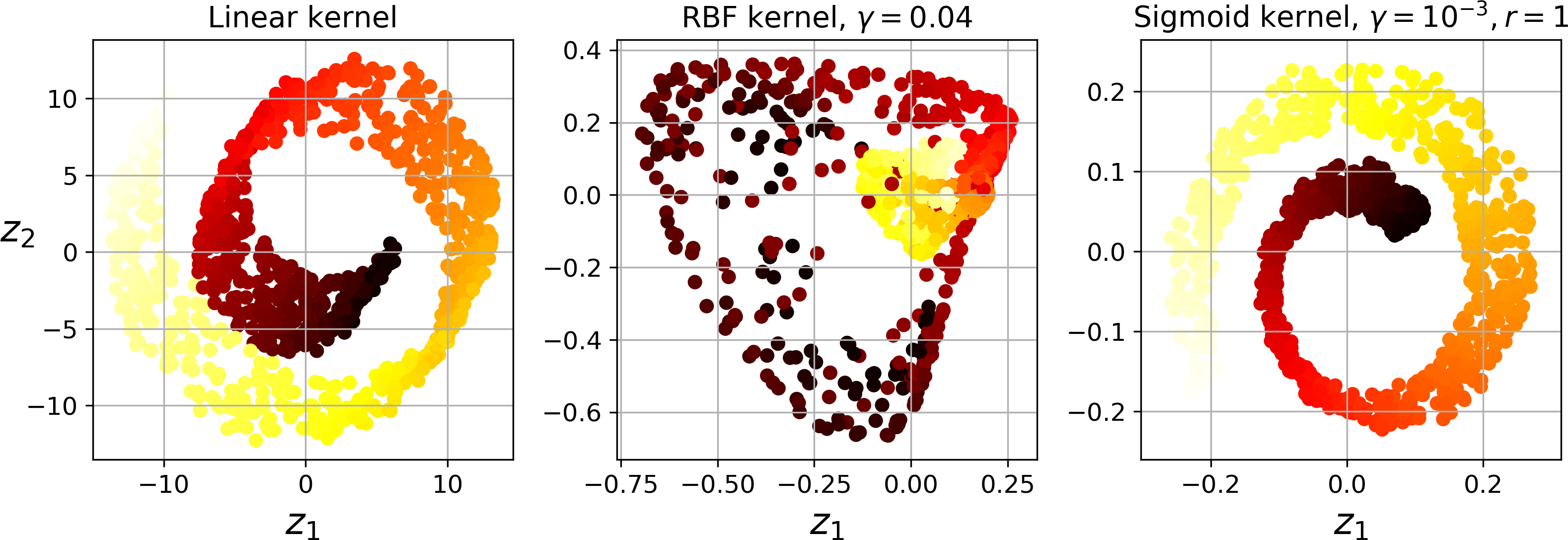
2.4 核PCA
from sklearn.decomposition import KernelPCA
rbf_pca=KernelPCA(n_components=2,kernel='rbf',gamma=0.04)
X_reduced=rbf_pca.fit_transform(X)
2.5. 调参
由于 kPCA 是无监督学习算法,没有明显的性能指标帮助选择参数
- 使用网格搜索来选择最佳表现的核方法和超参数
from sklearn.model_selection import GridSearchCV
from sklearn.linear_model import LogisticRegression
from sklearn.pipeline import Pipeline
clf = Pipeline([
("kpca", KernelPCA(n_components=2)),
("log_reg", LogisticRegression())
])
param_grid = [{
"kpca__gamma": np.linspace(0.03, 0.05, 10),
"kpca__kernel": ["rbf", "sigmoid"]
}]
grid_search = GridSearchCV(clf, param_grid, cv=3)
grid_search.fit(X, y)
获得最佳参数
print(grid_search.best_params_)
{'kpca__gamma': 0.043333333333333335, 'kpca__kernel': 'rbf'}
还可以比较重构后的数据跟原始数据的误差来找最佳参数
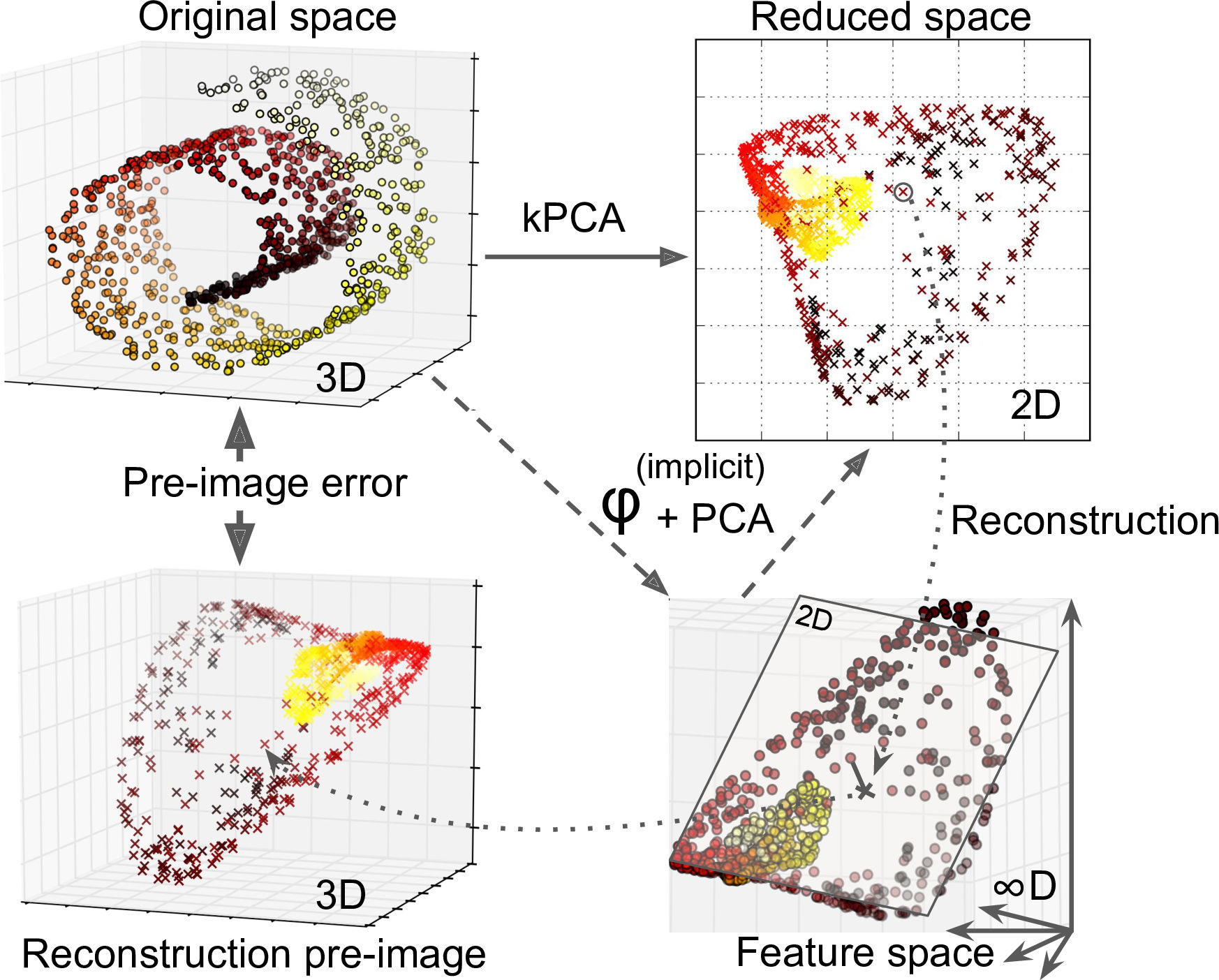
rbf_pca = KernelPCA(n_components = 2, kernel="rbf", gamma=0.0433,fit_inverse_transform=True)
X_reduced = rbf_pca.fit_transform(X)
X_preimage = rbf_pca.inverse_transform(X_reduced)
from sklearn.metrics import mean_squared_error
mean_squared_error(X, X_preimage)
32.786308795766132
然后网格搜索最小误差的 核方法 和 超参数
2.6 LLE
局部线性嵌入(Locally Linear Embedding)是另一种非常有效的非线性降维(NLDR)方法,是一种流形学习技术
- 它特别擅长展开扭曲的流形
from sklearn.manifold import LocallyLinearEmbedding
lle=LocallyLinearEmbedding(n_components=2,n_neighbors=10)
X_reduced=lle.fit_transform(X)
这个算法在处理 大数据集 的时候 表现 较差
2.7 其他方法
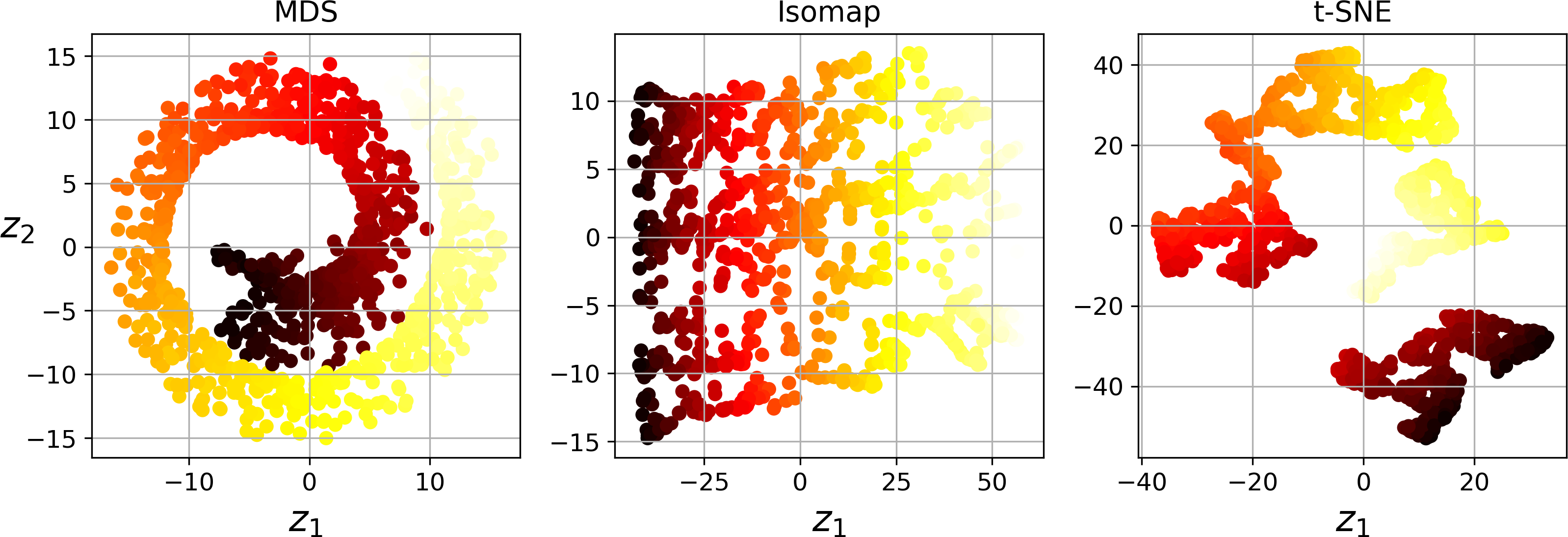
- 多维缩放(MDS)在尝试保持实例之间距离的同时降低了维度
- Isomap 通过将每个实例连接到最近的邻居来创建图形,然后在尝试保持实例之间的测地距离时降低维度
- t-分布随机邻域嵌入(t-Distributed Stochastic Neighbor Embedding,t-SNE)可以用于降低维度,同时试图保持相似的实例临近并将不相似的实例分开。
它主要用于可视化,尤其是用于可视化高维空间中的实例(例如,可以将MNIST图像降维到 2D 可视化) - 线性判别分析(Linear Discriminant Analysis,LDA)实际上是一种分类算法,但在训练过程中,它会学习类之间最有区别的轴,然后使用这些轴来定义用于投影数据的超平面
LDA 的好处是投影会尽可能地保持各个类之间距离,所以在运行另一种分类算法(如 SVM 分类器)之前,LDA 是很好的降维技术