进化算法,或称“演化算法” (evolutionary algorithms, EAS) 是一个“算法簇”,尽管它有很多的变化,有不同的遗传基因表达方式,不同的交叉和变异算子,特殊算子的引用,以及不同的再生和选择方法,但它们产生的灵感都来自于大自然的生物进化。与传统的基于微积分的方法和穷举法等优化算法相比,进化计算是一种成熟的具有高鲁棒性和广泛适用性的全局优化方法,具有自组织、自适应、自学习的特性,能够不受问题性质的限制,有效地处理传统优化算法难以解决的复杂问题。
最近看了一篇进化计算的论文:
ISDE+ - An Indicator for Multi and Many-objective Optimization:地址
文章目录
1. 源码下载与介绍
代码可以在上述链接的最后下载,这里讲解一下代码的具体含义。
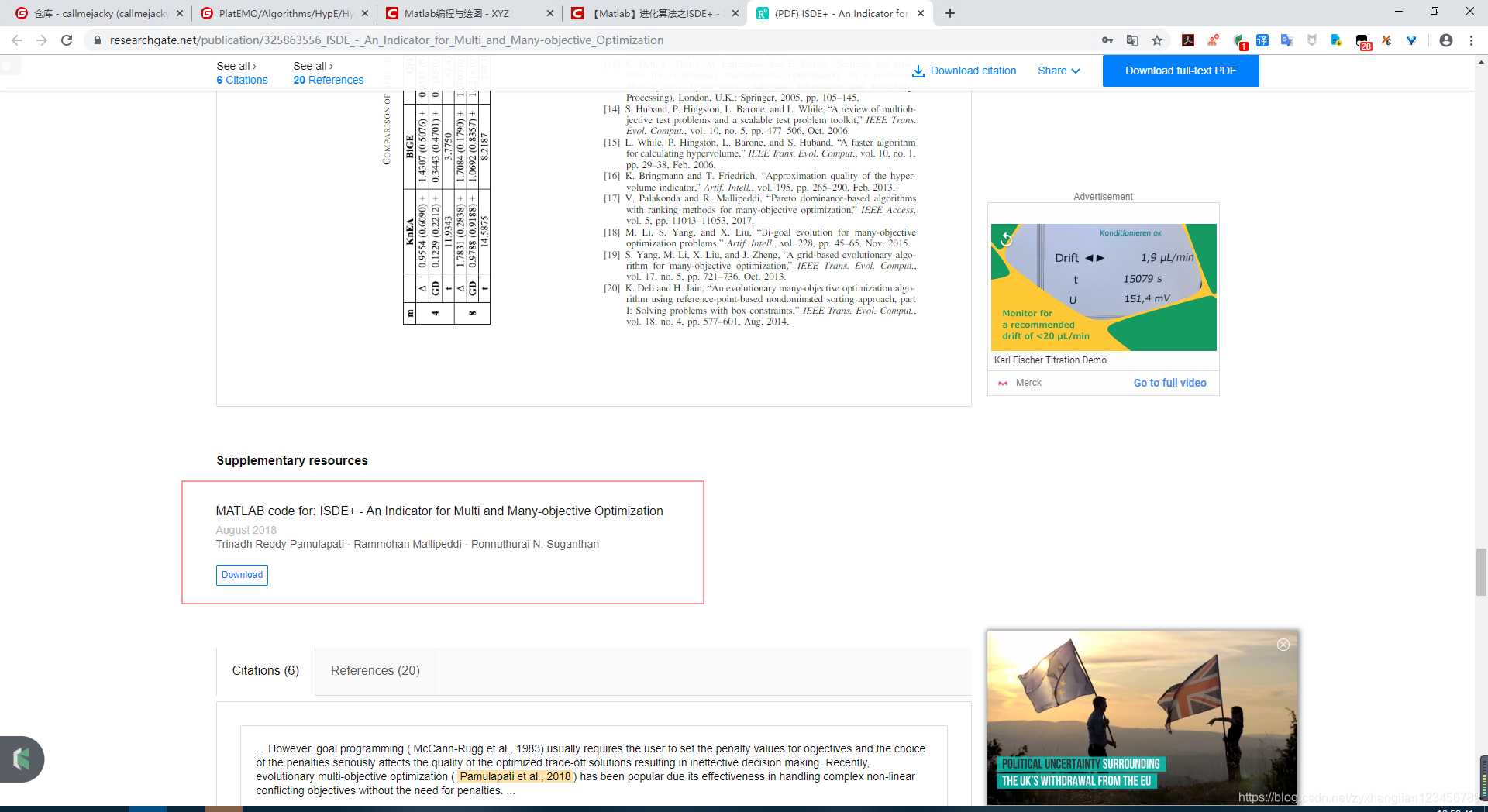
下载并解压后可以看到两个子文件夹,相信熟悉进化计算的同学应该知道DTLZ和WFG的含义。
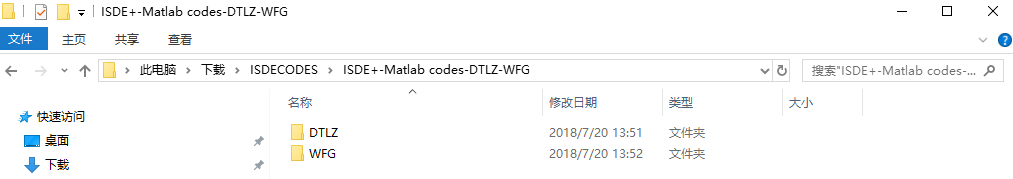
没错,它们是进化算法中常用的两组测试函数。拿DTLZ为例,可以看到,其中的内容如下:
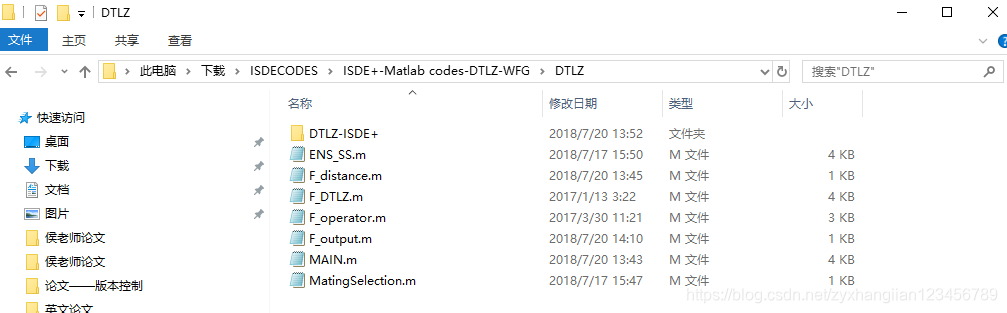
共有7个matlab文件,和一个空的文件夹。
2.详细细节
2.1 主要流程
算法的主要流程见下面的伪代码:

DTLZ7主要包含7个测试问题,即DTLZ1-DTLZ7,Main.m文件中首先对该问题进行一些参数设置,然后对算法进行一些参数配置,如设置种群的规模、评价次数等,具体的逻辑部分的讲解放在代码的注释中,大家可以详细看一下代码:
for Problem = 1:7
%-----------------------------------------------------------------------------------------
% DTLZ问题参数设置
% M代表目标函数的个数
for M = 4:2:10
if Problem == 1 % DTLZ1
K = 5; % the parameter in DTLZ1
elseif Problem == 2 || 3 || 4
K = 10; % the parameter in DTLZ2, DTLZ3, DTLZ4,
elseif Problem == 5 || 6
K = 10; % the parameter in DTLZ5, DTLZ6
elseif Problem == 7 % DTLZ7
K = 20; % the parameter in DTLZ7
fmax = repmat(max(PopObj,[],1),N,1);
fmin = repmat(min(PopObj,[],1),N,1);
end
D = M + K - 1;
MinValue = zeros(1,D);
MaxValue = ones(1,D);
%-----------------------------------------------------------------------------------------
% 算法参数设置
% 评价次数
if Problem == 1
Generations = 700; % number of iterations
elseif Problem == 3
Generations = 1000;
else
Generations = 250;
end
% 种群规模
if M == 2
N = 100;
elseif M == 4
N = 120;
elseif M == 6
N = 132;
elseif M == 8
N = 156;
elseif M == 10
N = 276;
end
% 独立运行次数
Runs = 30;
% 变量取值范围
Boundary = [MaxValue;MinValue];
%-----------------------------------------------------------------------------------------
% 独立运行若干次实验
for run = 1: Runs
% 种群初始化
Population = repmat(MinValue,N,1) + repmat(MaxValue - MinValue,N,1).*rand(N,D); % 随机初始化种群
FunctionValue = F_DTLZ(Population,Problem,M,K); % 计算目标函数值
DistanceValue = F_distance(FunctionValue,M); %计算距离值
%-----------------------------------------------------------------------------------------
% 开始迭代
for Gene = 1 : Generations
% 按照规模为2的锦标赛算法进行: 交配选择操作
% 距离值小的更容易获得产生下一代的机会,类似于自然界中的优秀的个体,
% 更容易产生后代,使得优秀的基因得以延续和发展,即优胜劣汰
MatingPool = MatingSelection(FunctionValue,DistanceValue,M);
NewPopulation = F_operator(Population(MatingPool',:),Boundary); %生成后代种群
Population = [Population;NewPopulation]; % 合并两个种群
FunctionValue = F_DTLZ(Population,Problem,M,K); % 再次计算目标函数值,若研究的问题不同,这里的目标函数计算方法也不同
DistanceValue = F_distance(FunctionValue,M); %计算距离值
[~,rank] = sort(DistanceValue,'ascend'); %按照升序排序
% 产生下一代,即从合并后的2N个个体中淘汰掉N个个体,使得种群总体保持为N
Population = Population(rank(1:N),:);
FunctionValue = FunctionValue(rank(1:N),:);
DistanceValue = DistanceValue(rank(1:N));
end
% 输出结果
F_output(Population,toc,'DTLZ-ISDE+',Problem,M,K,run);
end
end
end
2.2 计算ISED+距离
这里的距离值可以直观地理解为个体的适应度,距离值越大,个体适应能力越强。能力强的个体有更大的概率能够延续下一代,并且以较大的概率使得自身能够不被环境所淘汰。
ISED作为一种密度估计指标,通常是作为基于Pareto的进化算法的备用指标,即当进行非支配排序后,若不能够区分不同个体的优劣后,再利用该指标进行判断。ISED虽然能够同时评估种群的收敛性和多样性,但是由于其选择的压力不够大,因此,不能单独作为一种评价指标来指导种群收敛到Pareto前沿。
改进的ISED通过将目标值首先进行最大最小值标准化,再将个体的每个目标值进行求和,使得选择的压力得以有效地改进,这里计算方法如下:
function DistanceValue = F_distance(FunctionValue,M)
% FunctionValue为目标函数值 ,N为种群规模,M为目标函数的数量
[N,M] = size(FunctionValue);
PopObj = FunctionValue;
%% 目标值求和:这里并没有直接求和,而是通过求均值的方式,这里主要是想实现排序
fmax = repmat(max(PopObj,[],1),N,1);
fmin = repmat(min(PopObj,[],1),N,1);
PopObj = (PopObj-fmin)./(fmax-fmin); % 常见的归一化方法:最大最小归一化
fpr = mean(PopObj,2); % 求归一化后每一列元素的均值,即求归一化后个体的标准目标函数的均值
[~,rank] = sort(fpr); % 按照个体目标值的均值的升序排序
%%%%%%%%%%%%%% SDE with Sum of Objectives(核心代码) %%%%%%%%%%%%%%%%%%%%
DistanceValue = zeros(1,N);
for j = 2 : N
% 当个体j的目标函数值大于种群中其他个体q时,取j的目标值,后面在计算欧式距离时,将不会计算当前目标值的欧式距离。
SFunctionValue = max(PopObj(rank(1:j-1),:),repmat(PopObj(rank(j),:),(j-1),1));
Distance = inf(1,j-1); % 先定义欧式距离为无穷大
for i = 1 : (j-1)
Distance(i) = norm(SFunctionValue(i,:)-PopObj(rank(j),:))/M; %norm(A,2) %求A的欧几里德范数 ,和norm(A)相同。
end
Distance = min(Distance); % 求个体j与种群中其他个体的欧式距离的最小值
DistanceValue(rank(j)) = exp(-Distance); % 这里,通过e的-x的方式,将个体j的距离归一到0~1之间。
% 由于DistanceValue和Distance之间呈负相关关系,因此距离Distance越大越好的问题被转换为DistanceValue越小越好。
end
2.3 交配选择
交配选择操作主要是通过规模为2的锦标赛选择方式,这是在进化算法中常见的选择方式,模拟自然界中一个普遍存在的规律,即优秀个体有更大的概率能够繁衍后代。具体过程如下所示:

function MatingPool = MatingSelection(PopObj,DistanceValue,M)
N = size(PopObj,1);
%% 规模为2的锦标赛选择
Parent1 = randi(N,1,N); % 随机产生一个1×N的1~N之间的随机整数
Parent2 = randi(N,1,N); % 也可以理解为从N个个体中有放回地随机选择N个个体
MatingPool = zeros(1,N);
for i = 1:N
% 根据距离值比较两个个体,距离值小的个体会被保留下来,以进行交叉和变异操作
if DistanceValue(Parent1(i)) < DistanceValue(Parent2(i))
MatingPool(i) = Parent1(i);
elseif DistanceValue(Parent1(i)) > DistanceValue(Parent2(i))
MatingPool(i) = Parent2(i);
else
% 距离值相同,则随机选择一个即可
if rand< 0.5
MatingPool(i) = Parent1(i);
else
MatingPool(i) = Parent2(i);
end
end
end
2.4 环境选择
环境选择是在模拟自然界中优胜劣汰的机制,也比较简单,直接计算出个体的ISDE+,并按照升序排列,截取前N个个体即可。

2.5 操作算子
这部分主要讲解一下产生下一代的具体过程,包括模拟二进制交叉和多项式变异两个操作,注意,这是一种目前常见的实数编码下的交叉和变异操作,其他的内容请大家参考代码的注释加以理解:
function Offspring = F_operator(MatingPool, Boundary)
[N,D] = size(MatingPool);
%-----------------------------------------------------------------------------------------
% 参数设置
ProC = 1; % 交叉概率
ProM = 1/D; % 变异概率
DisC = 20; % 交叉操作的分布指数
DisM = 20; % 变异操作的分布指数
%-----------------------------------------------------------------------------------------
% 模拟二进制交叉
Parent1 = MatingPool(1:N/2,:);
Parent2 = MatingPool(N/2+1:end,:);
beta = zeros(N/2,D);
miu = rand(N/2,D);
beta(miu<=0.5) = (2*miu(miu<=0.5)).^(1/(DisC+1));
beta(miu>0.5) = (2-2*miu(miu>0.5)).^(-1/(DisC+1));
beta = beta.*(-1).^randi([0,1],N/2,D);
beta(rand(N/2,D)<0.5) = 1;
beta(repmat(rand(N/2,1)>ProC,1,D)) = 1;
Offspring = [(Parent1+Parent2)/2+beta.*(Parent1-Parent2)/2
(Parent1+Parent2)/2-beta.*(Parent1-Parent2)/2];
%-----------------------------------------------------------------------------------------
% 多项式变异
MaxValue = repmat(Boundary(1,:),N,1);
MinValue = repmat(Boundary(2,:),N,1);
k = rand(N,D);
miu = rand(N,D);
Temp = k<=ProM & miu<0.5;
Offspring(Temp) = Offspring(Temp)+(MaxValue(Temp)-MinValue(Temp)).*((2.*miu(Temp)+(1-2.*miu(Temp)).*(1-(Offspring(Temp)-MinValue(Temp))./(MaxValue(Temp)-MinValue(Temp))).^(DisM+1)).^(1/(DisM+1))-1);
Temp = k<=ProM & miu>=0.5;
Offspring(Temp) = Offspring(Temp)+(MaxValue(Temp)-MinValue(Temp)).*(1-(2.*(1-miu(Temp))+2.*(miu(Temp)-0.5).*(1-(MaxValue(Temp)-Offspring(Temp))./(MaxValue(Temp)-MinValue(Temp))).^(DisM+1)).^(1/(DisM+1)));
%-----------------------------------------------------------------------------------------
% 处理一下下一代个体中越界的变量值
Offspring(Offspring>MaxValue) = MaxValue(Offspring>MaxValue);
Offspring(Offspring<MinValue) = MinValue(Offspring<MinValue);
end
2.6 快速非支配排序
ENS_SS.m函数主要是实现快速非支配排序。先介绍一下非支配排序的概念出现在NSGAII中,下边是一个比较经典的图,表示在父代种群经过交叉变异之后,如何进行选择的过程。
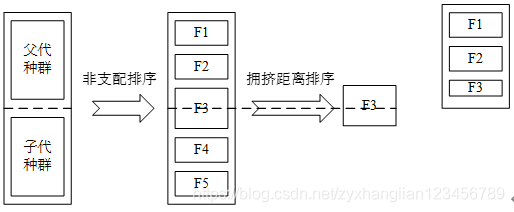
如上图所示,父代种群和子代种群合并在一起,经过非支配排序后,分为5个不同的等级。其中,等级F1中包含合并后种群中的所有非支配解集。等级F2中,包含仅仅被一个F1中的个体支配的个体所组成的集合,其他的以此类推。
然而,非支配排序是很昂贵的,特别是当种群中的个体数量变得很大的时候。这主要是因为在大多数现有的非支配排序算法中,一个解需要与所有其他解进行比较,然后才能分配给一个等级。因此,就有了ENS,即高效的非支配排序算法,感兴趣的同学可以参考一下下面的论文:
An Efficient Approach to Non-dominated Sorting for Evolutionary Multi-objective Optimization
2.7 输出结果
这部分介绍一下,算法如何输出最终结果的,主要涉及到获取非支配解集和导出结果到文件操作。
function F_output(Population,time,Algorithm,Problem,M,K,run)
% 获取非支配解集并导出结果
% 计算目标函数值
FunctionValue = F_DTLZ(Population,Problem,M,K);
% 找到非支配解集
NonDominated = ENS_SS(FunctionValue,'first')==1;
Population = Population(NonDominated,:);
FunctionValue = FunctionValue(NonDominated,:);
% 将结果保存到.m文件中
eval(['save DTLZ-ISDE+\', Algorithm,'_', num2str(Problem),'_', num2str(M),'_', num2str(run), ' Population FunctionValue time'])
% 将结果保存到txt中
%savedata(Population ,[Algorithm,'_', num2str(Problem),'_', num2str(M),'_', num2str(run),'_PS.txt']);
%savedata(FunctionValue,[Algorithm,'_', num2str(Problem),'_', num2str(M),'_', num2str(run),'_PF.txt']);
end
% 将结果保存到txt文件中
function savedata(mat,filename)
f=fopen(filename,'w');
for i=1:size(mat,1)
for j=1:size(mat,2)
fprintf(f,'%15.6e',mat(i,j));
end
fprintf(f,'\r\n');
end
fclose(f);
end
2.7 DTLZ测试问题的定义
有关DTLZ测试问题的详细介绍这里不做单独说明,函数的定义如下:
function FunctionValue = F_DTLZ(Population,Problem,M, K)
if Problem == 1 % DTLZ1
FunctionValue = zeros(size(Population,1),M);
g = 100*(K+sum((Population(:,M:end)-0.5).^2 - cos(20.*pi.*(Population(:,M:end)-0.5)),2));
FunctionValue(:,1) = 0.5.*prod(Population(:,1:M-1),2).*(1+g);
for i = 2 : (M-1)
FunctionValue(:,i) = 0.5*prod(Population(:,1:M-i),2).*(1 - Population(:,M-i+1)).*(1 + g);
end
FunctionValue(:,M) = 0.5*(1 - Population(:,1)).*(1 + g);
end
if Problem == 2 % DTLZ2
FunctionValue = zeros(size(Population,1),M);
g = sum((Population(:,M:end)-0.5).^2,2);
FunctionValue(:,1) = prod(cos(pi/2*Population(:,1:M-1)),2).*(1 + g);
for i = 2 : (M-1)
FunctionValue(:,i) = prod(cos(pi/2*Population(:,1:M-i)),2).* sin(pi/2*Population(:,M-i+1)).*(1 + g);
end
FunctionValue(:,M) =sin(pi/2*Population(:,1)).*(1 + g);
end
if Problem == 3 % DTLZ3
FunctionValue = zeros(size(Population,1),M);
g = 100*(K+sum((Population(:,M:end)-0.5).^2 - cos(20.*pi.*(Population(:,M:end)-0.5)),2));
FunctionValue(:,1) = prod(cos(pi/2*Population(:,1:M-1)),2).*(1 + g);
for i = 2 : (M-1)
FunctionValue(:,i) = prod(cos(pi/2*Population(:,1:M-i)),2).* sin(pi/2*Population(:,M-i+1)).*(1 + g);
end
FunctionValue(:,M) =sin(pi/2*Population(:,1)).*(1 + g);
end
if Problem == 4 % DTLZ4
alpha = 100;
FunctionValue = zeros(size(Population,1),M);
g = sum((Population(:,M:end)-0.5).^2,2);
FunctionValue(:,1) = prod(cos(pi/2*Population(:,1:M-1).^alpha),2).*(1 + g);
for i = 2 : (M-1)
FunctionValue(:,i) = prod(cos(pi/2*Population(:,1:M-i).^alpha),2).* sin(pi/2*Population(:,M-i+1).^alpha).*(1 + g);
end
FunctionValue(:,M) =sin(pi/2*Population(:,1).^alpha).*(1 + g);
end
if Problem == 5 % DTLZ5
FunctionValue = zeros(size(Population,1),M);
g = sum((Population(:,M:end)-0.5).^2,2);
theta(:,1) = pi/2*Population(:,1);
gr = g(:,ones(1,M-2)); %replicates gr for the multiplication below
theta(:,2:M-1) = pi./(4*(1+gr)) .* (1 + 2*gr.*Population(:,2:M-1));
FunctionValue(:,1) = prod(cos(theta(:,1:M-1)),2).*(1 + g);
for i = 2 : (M-1)
FunctionValue(:,i) = prod(cos(theta(:,1:M-i)),2).*sin(theta(:,M-i+1)).*(1 + g);
end
FunctionValue(:,M) = sin(theta(:,1)).*(1 + g);
end
if Problem == 6 % DTLZ6
FunctionValue = zeros(size(Population,1),M);
g = sum(Population(:,M:end).^(0.1),2);
theta(:,1) = pi/2*Population(:,1);
gr = g(:,ones(1,M-2)); %replicates gr for the multiplication below
theta(:,2:M-1) = pi./(4*(1+gr)) .* (1 + 2*gr.*Population(:,2:M-1));
FunctionValue(:,1) = prod(cos(theta(:,1:M-1)),2).*(1 + g);
for i = 2 : (M-1)
FunctionValue(:,i) = prod(cos(theta(:,1:M-i)),2).*sin(theta(:,M-i+1)).*(1 + g);
end
FunctionValue(:,M) = sin(theta(:,1)).*(1 + g);
end
if Problem == 7 % DTLZ7
FunctionValue = zeros(size(Population,1),M);
g = 1 + 9/K *sum(Population(:,M:end),2);
FunctionValue(:,1:M-1) = Population(:,1:M-1);
gaux = g(:,ones(1,M-1)); %replicates the g function
h = M - sum(FunctionValue(:,1:M-1)./(1+gaux).*(1 + sin(3*pi*FunctionValue(:,1:M-1))),2);
FunctionValue(:,M) = h.*(1 + g);
end
