聚类方法以及实现
- 1 聚类方法概览
- 2 保持原始变量分布进行聚类
- 2.1 读入数据
- 2.2 相关性矩阵
- 2.3 看变量的分布
- 2.4 中心标准化进行预处理
- 2.5 评估模型效果
- 2.5.1 评估方法1-轮廓系数
- 2.5.2 评估方法2-RMSSTD平方根标准误差
- 2.5.3 评估方法3-R方¶
- 2.5.4 评估方法4-ARI¶
- 2.5.5 评估方法5 - Calinski-Harabasz Index¶
- 2.6 使用主成分分析降维
- 2.7 确定离群点所在的簇
- 2.8 总结
- 3 对变量进行分布形态转换后聚类
- 4 如何确定聚类个数是否合理?
- 5 后续工作
- 6 数据
- 7 时间复杂度和空间复杂度是多少?
- 8 KMeans初始点如何选择?
- 9 KMeans、层次聚类、DBSCAN三者比较
- 参考
1 聚类方法概览
聚类其实有三大类方法,一种是基于层次的方法(如层次聚类法),一种是基于划分的方法(如KMeans),一种是基于密度的方法(DBSCAN)。
1.1 层次聚类(系统聚类)
1.1.1 思想
分为两种:聚合(一开始各自为一类)和分裂(一开始为一大类)。
-
聚合:一开始将每个样本各自分为一类,之后将距离最近的两个两类合并,建立一个新类别,重复此操作直至为一大类!画出谱系聚类图,根据实际问题的需要确定类别的划分!
-
分裂:一开始将所有样本合为一类,之后将距离最远的样本分到两个新的类,重复此操作直至满足条件为止!画出谱系聚类图,根据实际问题的需要确定类别的划分!
涉及到两个距离的计算:一个是样本间距离,一个是类与类之间距离。
样本间距离:欧氏距离;闵可夫斯基距离;马哈拉诺比斯距离;相关系数;夹角余弦;
类与类之间的距离:最短距离法;最长距离法;重心法;类平均法(两个类中任意两个样本之间距离的均值作为两类之间的距离)。
1.1.2 代码实现
#导入相应的包
import scipy
import scipy.cluster.hierarchy as sch
from scipy.cluster.vq import vq,kmeans,whiten
import numpy as np
import matplotlib.pylab as plt
import pandas as pd
from sklearn import preprocessing
from sklearn.decomposition import PCA
#导入数据
orgData = pd.read_csv('cities_10.csv', index_col="AREA", encoding='gbk')
orgData.head()
#orgData.describe()
| X1 | X2 | X3 | X4 | X5 | X6 | X7 | X8 | X9 | |
|---|---|---|---|---|---|---|---|---|---|
| AREA | |||||||||
| 辽宁 | 5458.2 | 13000 | 1376.2 | 2258.4 | 1315.9 | 529.0 | 2258.4 | 123.7 | 399.7 |
| 山东 | 10550.0 | 11643 | 3502.5 | 3851.0 | 2288.7 | 1070.7 | 3181.9 | 211.1 | 610.2 |
| 河北 | 6076.6 | 9047 | 1406.7 | 2092.6 | 1161.6 | 597.1 | 1968.3 | 45.9 | 302.3 |
| 天津 | 2022.6 | 22068 | 822.8 | 960.0 | 703.7 | 361.9 | 941.4 | 115.7 | 171.8 |
| 江苏 | 10636.3 | 14397 | 3536.3 | 3967.2 | 2320.0 | 1141.3 | 3215.8 | 384.7 | 643.7 |
#标准化
x_scaled = preprocessing.scale(orgData+0.0)#归一化,但是只能用于浮点类型变量
# 如果既有浮点型又有整型 则最好 +0.0 防止出现warnings
pd.DataFrame(x_scaled).head()
| 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | -0.375748 | -0.335897 | -0.570700 | -0.331801 | -0.435115 | -0.686087 | -0.138827 | -0.449589 | -0.360085 |
| 1 | 1.199240 | -0.483152 | 1.144732 | 0.984558 | 0.799910 | 0.865054 | 0.664019 | -0.278096 | 0.349766 |
| 2 | -0.184465 | -0.764857 | -0.546094 | -0.468842 | -0.631008 | -0.491085 | -0.391025 | -0.602244 | -0.688539 |
| 3 | -1.438442 | 0.648116 | -1.017166 | -1.404990 | -1.212338 | -1.164573 | -1.283762 | -0.465286 | -1.128613 |
| 4 | 1.225934 | -0.184302 | 1.172001 | 1.080603 | 0.839647 | 1.067215 | 0.693490 | 0.062534 | 0.462736 |
#变量压缩
pca=PCA(n_components=2)
newData=pca.fit_transform(x_scaled)
pca.explained_variance_ratio_ # 主成分分析的方差解释率
array([0.80112955, 0.12214932])
newData
array([[-1.18945132, -0.31092235],
[ 2.06415695, -0.74854414],
[-1.43769023, -0.80669682],
[-3.23039706, 0.84519783],
[ 2.36892693, -0.44480961],
[ 0.28997221, 2.79266758],
[ 1.2099519 , -0.00638496],
[-2.09689459, -0.22796377],
[ 5.50091159, -0.14275827],
[-3.47948639, -0.94978548]])
#1. 层次聚类
#生成点与点之间的距离矩阵,这里用的欧氏距离:
disMat = sch.distance.pdist(newData,'euclidean')
#进行层次聚类:
Z=sch.linkage(disMat,method='average')
#将层级聚类结果以树状图表示出来并保存为plot_dendrogram.png
P=sch.dendrogram(Z)
plt.savefig('plot_dendrogram2.png')
plt.show()

# 查看距离矩阵
disMat
array([3.28290719, 0.55445008, 2.34564989, 3.56089617, 3.43816294,
2.41865227, 0.91122742, 6.692476 , 2.37747907, 3.50233 ,
5.52922379, 0.43027829, 3.96079688, 1.13157701, 4.19348946,
3.48973623, 5.5472948 , 2.43773537, 3.8237804 , 3.99252321,
2.76595516, 0.87720142, 6.9702948 , 2.04680383, 5.74600282,
4.02313786, 4.52127108, 1.56093039, 8.78702498, 1.81218392,
3.84750715, 1.23912844, 4.47108308, 3.14651599, 5.87017368,
2.94636348, 3.84985024, 5.98085396, 5.311758 , 3.31426174,
4.29312622, 4.78339169, 7.59828393, 1.55967518, 9.01658698])
1.1.3 优缺点
优点:
- 画出谱系聚类图来划分比较直观!
- 无需事先确定聚类个数有多少个,根据图形判断!
- 既可以对变量聚类,又可以对样本进行聚类
缺点:
- 大样本下计算效率低
1.2 K-means聚类
1.2.1 思想
- 需要事先确定聚类的个数K;
- 随机选择k个初始类中心
- 计算每个样本点到聚类中心的距离,离哪个聚类中心近就归为一类!
- 将所有样本过一遍之后,对k个类重新计算类中心,然后重复第3步,直至k个类中心不再发生变化为止!
1.2.2 代码实现
import numpy as np
from sklearn import cluster
import pandas as pd
from sklearn.decomposition import PCA
from sklearn import preprocessing
import matplotlib.pyplot as plt
%matplotlib inline
iris = pd.read_csv('iris.csv')
x=iris.ix[:,"Sepal.Length":"Petal.Width"]
y=iris["Species"]
C:\ProgramData\Anaconda3\lib\site-packages\ipykernel_launcher.py:2: DeprecationWarning:
.ix is deprecated. Please use
.loc for label based indexing or
.iloc for positional indexing
See the documentation here:
http://pandas.pydata.org/pandas-docs/stable/indexing.html#deprecate_ix
归一化的使用说明 http://www.cnblogs.com/chaosimple/p/4153167.html
x_scaled = preprocessing.scale(x+0.0)#归一化,但是只能用于浮点类型变量
pd.DataFrame(x_scaled).head()
| 0 | 1 | 2 | 3 | |
|---|---|---|---|---|
| 0 | -0.900681 | 1.019004 | -1.340227 | -1.315444 |
| 1 | -1.143017 | -0.131979 | -1.340227 | -1.315444 |
| 2 | -1.385353 | 0.328414 | -1.397064 | -1.315444 |
| 3 | -1.506521 | 0.098217 | -1.283389 | -1.315444 |
| 4 | -1.021849 | 1.249201 | -1.340227 | -1.315444 |
pca=PCA(n_components=3)
newData=pca.fit_transform(x_scaled)
pca.explained_variance_ratio_
array([ 0.72962445, 0.22850762, 0.03668922])
score=pd.DataFrame(newData)
score.head()
| 0 | 1 | 2 | |
|---|---|---|---|
| 0 | -2.264703 | 0.480027 | -0.127706 |
| 1 | -2.080961 | -0.674134 | -0.234609 |
| 2 | -2.364229 | -0.341908 | 0.044201 |
| 3 | -2.299384 | -0.597395 | 0.091290 |
| 4 | -2.389842 | 0.646835 | 0.015738 |
- 参考官方文档:http://scikit-learn.org/stable/modules/generated/sklearn.cluster.KMeans.html#sklearn.cluster.KMeans
from sklearn.cluster import KMeans
kmeans = cluster.KMeans(n_clusters=3) #MiniBatchKMeans()分批处理
#kmeans = cluster.KMeans(n_clusters=3, init='random', n_init=1)
result=kmeans.fit(x_scaled)
print(result)
KMeans(algorithm='auto', copy_x=True, init='k-means++', max_iter=300,
n_clusters=3, n_init=10, n_jobs=1, precompute_distances='auto',
random_state=None, tol=0.0001, verbose=0)
result.labels_
array([1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 0, 0, 0, 2, 2, 2, 0, 2, 2, 2, 2, 2, 2, 2, 2, 0, 2, 2, 2,
2, 0, 2, 2, 2, 2, 0, 0, 0, 2, 2, 2, 2, 2, 2, 2, 0, 0, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 0, 2, 0, 0, 0, 0, 2, 0, 0, 0, 0, 0, 0, 2, 2,
0, 0, 0, 0, 2, 0, 2, 0, 2, 0, 0, 2, 0, 0, 0, 0, 0, 0, 2, 2, 0, 0, 0,
2, 0, 0, 0, 2, 0, 0, 0, 2, 0, 0, 2])
lo = plt.scatter(score[0][result.labels_==0],score[1][result.labels_==0], marker='x')
lo = plt.scatter(score[0][result.labels_==1],score[1][result.labels_==1], marker='o')
lo = plt.scatter(score[0][result.labels_==2],score[1][result.labels_==2], marker='+')

1.2.3 优缺点
优点:
- 处理大规模的数据速度较快 实现快速聚类
- 算法比较简单
缺点:
-
k的选取比较敏感
-
初始化的质心的位置选择对后面聚类结果有影响,局部最优的,只适用于凸数据集。即

-
只能对样本进行聚类
-
如果每个类不是簇状的,比如是条状的,k-means效果不好,这时候应该采用DBSCAN。
1.3 基于密度的DBSCAN聚类
1.3.1 思想
DBSCAN(Density-Based Spatial Clustering of Applications with Noise)是一种基于密度的空间聚类算法,它不需要定义簇的个数,而是将具有足够高密度的区域划分为簇,并在有噪声的数据中发现任意形状的簇,在此算法中将簇定义为密度相连的点的最大集合。
完整流程:
1.遍历人群,找到所有的核心对象
2.随机选中一个核心对象作为初始点,层层连接所有可以连接的核心对象,形成第一个簇
3.然后在剩下的核心对象中随机选择一个,重复第二步,直到所有核心对象都被消耗
4.获得生成的聚类簇
在过程中主要需要设置两个参数邻域半径 和最小样本数min_samples。
问题来了:
1、什么叫核心对象?
假设在你的邻域里有超过一定数量的人(比方说大于等于五个),那么你可以被称为一个核心对象
2、什么叫可以连接的核心对象?
假设一个邻域A的核心对象 同时也处在另一个核心对象 的邻域B,那么我们就可以将A和B连在一起(学名:密度直达),将所有能连在一起的邻域都连在一起,就形成一个聚类。
注:一个类中,所有邻域都可以彼此连接,或者透过几层连接关系相连(学名叫密度可达)。而且所有邻域都不能和其余类的邻域相连。
1.3.2 代码实现
详情见 博客:https://www.cnblogs.com/pinard/p/6217852.html
涉及到的调参主要是邻域半径 和最小样本数min_samples。
1.4 聚类效果评估
1.4.1 Silhouette Coefficient
http://scikit-learn.org/stable/modules/clustering.html#clustering
from sklearn import metrics
metrics.silhouette_score(x_scaled, result.labels_, metric='euclidean')
0.45994823920518646
1.4.2 Adjusted Rand index
http://scikit-learn.org/stable/modules/clustering.html#clustering
from sklearn import metrics
metrics.adjusted_rand_score(y, result.labels_ )
0.6201351808870379
1.5 练习
- 使用“profile_telecom”数据集进行聚类
- 下面就是用这个数据集来进行聚类的!使用的方法是KMeans
2 保持原始变量分布进行聚类
2.1 读入数据
## import pandas as pd
profile_telecom = pd.read_csv('profile_telecom.csv')
print(profile_telecom.shape)
profile_telecom.head()
(600, 5)
| ID | cnt_call | cnt_msg | cnt_wei | cnt_web | |
|---|---|---|---|---|---|
| 0 | 1964627 | 46 | 90 | 36 | 31 |
| 1 | 3107769 | 53 | 2 | 0 | 2 |
| 2 | 3686296 | 28 | 24 | 5 | 8 |
| 3 | 3961002 | 9 | 2 | 0 | 4 |
| 4 | 4174839 | 145 | 2 | 0 | 1 |
2.2 相关性矩阵
profile_telecom.loc[:,'cnt_call':].corr()
| cnt_call | cnt_msg | cnt_wei | cnt_web | |
|---|---|---|---|---|
| cnt_call | 1.000000 | 0.052096 | 0.117832 | 0.114190 |
| cnt_msg | 0.052096 | 1.000000 | 0.510686 | 0.739506 |
| cnt_wei | 0.117832 | 0.510686 | 1.000000 | 0.950492 |
| cnt_web | 0.114190 | 0.739506 | 0.950492 | 1.000000 |
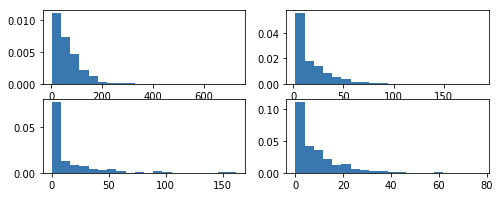
2.3 看变量的分布
import matplotlib.pyplot as plt
plt.figure(figsize=(8,3))
for i in range(4):
plt.subplot(220+i+1)
plt.hist(profile_telecom.iloc[:,i+1], bins=20, normed=True)
plt.show()
/Users/apple/anaconda3/lib/python3.6/site-packages/matplotlib/axes/_axes.py:6521: MatplotlibDeprecationWarning:
The 'normed' kwarg was deprecated in Matplotlib 2.1 and will be removed in 3.1. Use 'density' instead.
alternative="'density'", removal="3.1")
2.4 中心标准化进行预处理
from sklearn.preprocessing import scale
from sklearn.cluster import KMeans
k = 4
tele_scaled = scale(profile_telecom.iloc[:,1:])
tele_kmeans = KMeans(n_clusters=k, n_init=15) # 选取中心点15次 即迭代15次
tele_kmeans = tele_kmeans.fit(tele_scaled) # 拟合
tele_kmeans.cluster_centers_ # 查看聚类中心
/Users/apple/anaconda3/lib/python3.6/site-packages/ipykernel/__main__.py:5: DataConversionWarning: Data with input dtype int64 were all converted to float64 by the scale function.
array([[-0.2145554 , -0.49172093, -0.46821962, -0.5285122 ],
[ 0.39035452, 1.71921055, 3.32673647, 3.18414517],
[ 0.04525479, 0.89415401, 0.56707977, 0.73526001],
[ 4.07714914, -0.2997685 , -0.29238829, -0.31809185]])
2.5 评估模型效果
2.5.1 评估方法1-轮廓系数
思想:
- 单个观测点轮廓系数s取值范围为[-1,1] 越接近于1表示聚类效果越好,越接近于-1表示效果越不好
- s是怎么求的呢?有下面三种情况:
- 首先定义:单个观测点到同类所有点的距离的均值记为a,单个观测点到不同类所有点的距离的均值记为b
- s = ( b - a ) / max( a , b )
- 如果a < b, s = 1 - a/b s越大,即越接近于1,表明该观测点在聚类的类簇中是合理的,聚类效果越好
- 如果a > b, s = b/a - 1 即认为s越小越不好,表明该观测点还不如在别的类簇中,聚类效果不好
- 如果a = b, s = 0 这时候不好判断观测点在哪个类簇中效果好!
- 最后计算所有观测点s的均值,即为轮廓系数,取值为-1到1之间,越接近于1聚类效果越好,反之!
核心就是算每一个点到预测的类别以及其余的类别中的点的距离!如果到自身预测的类别的点的距离小,那就是合理的!聚类效果好!
from sklearn.metrics import silhouette_score
silhouette_score(tele_scaled, tele_kmeans.labels_)
0.4649262558707401
- 0.46表示聚类效果还不错
2.5.2 评估方法2-RMSSTD平方根标准误差
思想:计算某一个群体内所有点的标准差之和再除以样本点个数,记为RMSSTD,比较不同群体的RMSSTD值,值越小,表明群体内(簇内)个体对象之间的相似程度越高,聚类效果越好!
2.5.3 评估方法3-R方¶
思想:R方=组间方差/总方差=1-组内方差/总方差,表明群体间差异的大小,也就是聚类结果可以在多大比例上解释原始数据的方差,R方越大表明组间差异越大,聚类效果越好!
2.5.4 评估方法4-ARI¶
- 适用场景:已知样本点标签的时候使用!
- RI是rand 指数,可以计算!类似于precision【见纸】
- ARI是RI的变形!待补充
2.5.5 评估方法5 - Calinski-Harabasz Index¶
可以从簇内的稠密程度和簇间的离散程度来评估聚类的效果,得到的Calinski-Harabasz分数值s越大则聚类效果越好。
公式:

结论:
类别内部数据的方差越小越好,类别之间的方差越大越好,这样的Calinski-Harabasz分数会高。
参考链接:
- https://www.pangan.win/2018/12/07/MachineLearning/聚类评价指标/
- https://blog.csdn.net/luoleicn/article/details/5350378
- 刘建平博客:https://www.cnblogs.com/pinard/p/6169370.html
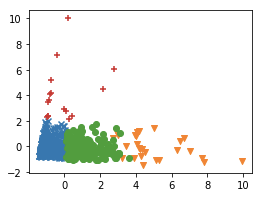
2.6 使用主成分分析降维
from sklearn.decomposition import PCA
plt.figure(figsize=[4,3])
tele_pca = PCA(n_components=2)
tele_pca_score = tele_pca.fit_transform(tele_scaled)
markers = 'xvo+*^dhs|_<,.>'
for cluster, marker in zip(range(k), markers[:k]):
x_axis = tele_pca_score[:,0][tele_kmeans.labels_ == cluster] # 选择预测的不同类别的点!然后涂上不一样的颜色
y_axis = tele_pca_score[:,1][tele_kmeans.labels_ == cluster]
plt.scatter(x_axis, y_axis, marker=marker)
plt.show()
2.7 确定离群点所在的簇
2.7.1 方法1
统计每个群组的样本点数量:
from collections import Counter
Counter(tele_kmeans.labels_)
Counter({0: 390, 1: 28, 2: 166, 3: 16})
结论:
- 第1和3类明显是离群点,样本点的数量只有28和16个!
2.7.2 方法2
pd.DataFrame(tele_pca_score).groupby(tele_kmeans.labels_).count()
| 0 | 1 | |
|---|---|---|
| 0 | 390 | 390 |
| 1 | 28 | 28 |
| 2 | 166 | 166 |
| 3 | 16 | 16 |
2.8 总结
- 上述两个簇所在的离群点可以进行进一步分析,如果目的是进行离群点的检测!
- 目前是没有对原始变量进行分布形态的转换,所以各个类别中样本点十分的不均衡,但是业务人员是希望客户数量在每个类中是大致均匀分布的!所以接下来进行分布形态的转换!
3 对变量进行分布形态转换后聚类
3.1 为什么要转换?
- 聚类的应用之一是进行市场细分,要求我们划分的簇具有相近的规模,便于用户管理和服务。
- 所以需要对严重偏态的数据进行转换,使其更接近对称分布!
3.2 变换方法有哪些
- 取对数
- 百分位秩
- 取rank
- Tukey打分
知识点:
- log1p = log(1 + x)
- 参考:https://docs.scipy.org/doc/numpy-1.14.5/reference/generated/numpy.log1p.html
3.3 进行数据变换
import numpy as np
log_telecom = np.log1p(profile_telecom.iloc[:,1:])
plt.figure(figsize=(8,3))
for i in range(4):
plt.subplot(220+i+1)
plt.hist(log_telecom.iloc[:,i], bins=20, normed=True)
plt.show()
/Users/apple/anaconda3/lib/python3.6/site-packages/matplotlib/axes/_axes.py:6521: MatplotlibDeprecationWarning:
The 'normed' kwarg was deprecated in Matplotlib 2.1 and will be removed in 3.1. Use 'density' instead.
alternative="'density'", removal="3.1")
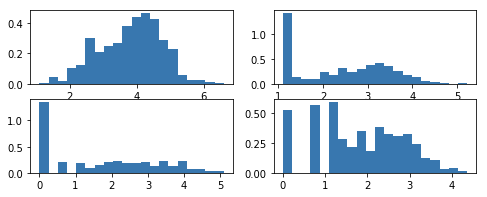
- 可以看到数据变换之后偏态得到了改善!
3.4 维度分析
from sklearn.decomposition import PCA
plt.figure(figsize=[4,3])
log_pca = PCA(n_components=2, whiten=True)
log_pca_score = log_pca.fit_transform(log_telecom)
print(log_pca.explained_variance_ratio_)
[0.79384647 0.14465683]
<Figure size 288x216 with 0 Axes>
3.5 使用主成分打分进行聚类
from sklearn.preprocessing import scale
from sklearn.cluster import KMeans
k = 3
log_pca_kmeans = KMeans(n_clusters=k, n_init=15) # 选取中心点15次 即迭代15次
log_pca_kmeans = log_pca_kmeans.fit(log_pca_score) # 拟合
log_pca_kmeans.cluster_centers_ # 查看聚类中心
array([[-0.74265865, 0.99318607],
[-0.78664736, -1.11507117],
[ 0.9331096 , 0.00421009]])
from sklearn.metrics import silhouette_score
silhouette_score(log_pca_score, log_pca_kmeans.labels_)
0.45245303605250087
3.6 绘图
plt.figure(figsize=[4,3])
markers = 'xvo+*^dhs|_<,.>'
for cluster, marker in zip(range(k), markers[:k]):
x_axis = log_pca_score[:,0][log_pca_kmeans.labels_ == cluster] # 选择预测的不同类别的点!然后涂上不一样的颜色
y_axis = log_pca_score[:,1][log_pca_kmeans.labels_ == cluster]
plt.scatter(x_axis, y_axis, marker=marker)
plt.show()
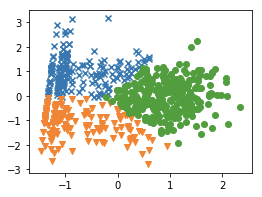
输出三类样本量大小
Counter(log_pca_kmeans.labels_)
Counter({0: 174, 1: 156, 2: 270})
3.7 计算每个类别下原始变量的均值
co = profile_telecom.iloc[:, 1:].groupby(log_pca_kmeans.labels_).mean()
co
| cnt_call | cnt_msg | cnt_wei | cnt_web | |
|---|---|---|---|---|
| 0 | 104.879310 | 5.764368 | 1.206897 | 2.178161 |
| 1 | 14.012821 | 7.782051 | 1.730769 | 2.666667 |
| 2 | 70.077778 | 31.251852 | 31.129630 | 16.788889 |
下面就根据上面的结果进行业务上的解读即可!
4 如何确定聚类个数是否合理?
其实问题就是如何选择合适的聚类个数k!以下三类指标(主要是后两个)分别作为纵轴,横轴是聚类个数k,绘制折线图,选择最优指标对应的k即可!
- 使用碎石图寻找指标较为合理时候的聚类数量!
- 一个指标为:离差平方和:样本到各自簇中心的距离和。越小越好!
- 另一个指标轮廓系数也可以用!通过枚举,令k从2到一个固定值如10,绘制每个k对应的效果评分!越接近1越好!。即最后选取轮廓系数最大的值对应的k作为最终的集群数目。
- 第三个指标可以考虑的就是:Calinski-Harabasz。即类间方差/类内方差。越大越好!也是横坐标选择k 纵轴为指标值!
plt.figure(figsize=[8,2])
Ks = range(2,10)
rssds = []; silhs = []
# 计算
for k in Ks:
model = KMeans(n_clusters=k, n_init=15)
model.fit(log_pca_score)
rssds.append(model.inertia_)
silhs.append(silhouette_score(log_pca_score, model.labels_))
# 绘图
plt.subplot(121);plt.plot(Ks, rssds)
plt.subplot(122);plt.plot(Ks, silhs)
plt.show()
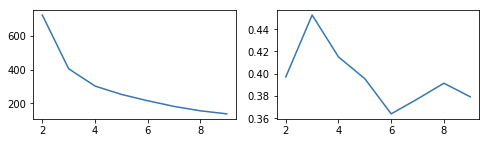
结论:选择k=3还是比较合理的!一方面离差平方和下降的最快,另一方面轮廓系数最大!
5 后续工作
- 案例实现+原理解析 层次聚类以及KMeans以及DBSCAN 刚好结合最近的项目
- rand和ARI的补充,主要是ARI!
6 数据
7 时间复杂度和空间复杂度是多少?
- 空间复杂度O(N)
- 时间复杂度O(I×K×N) 线性复杂度!
- 其中N是样本个数,l是迭代次数,K是聚类中心个数。
8 KMeans初始点如何选择?
8.1 kmeans++
思想:初始的聚类中心之间相互距离尽可能远。
步骤:
- 首先从输入的数据点集合中随机选择一个点作为第一个聚类中心
- 计算每个点到最近的聚类中心的距离D(x),D(x)较大的点,被选取作为聚类中心的概率较大。
- 重复第二步,直至选出了k个初始聚类中心。
至于第二步是怎么选的,可以考虑下面这种算法:
1、先从我们的数据库随机挑个随机点当“种子点”
2、对于每个点,我们都计算其和最近的一个“种子点”的距离D(x)并保存在一个数组里,然后把这些距离加起来得到Sum(D(x))。
3、然后,再取一个随机值,用权重的方式来取计算下一个“种子点”。这个算法的实现是,先取一个能落在Sum(D(x))中的随机值Random,然后用Random -= D(x),直到其<=0,此时的点就是下一个“种子点”。
4、重复2和3直到k个聚类中心被选出来
5、利用这k个初始的聚类中心来运行标准的k-means算法
但个人觉得取距离最大的点也无妨!
8.2 先用层次聚类
选用层次聚类进行初始聚类,然后从k个类别中分别随机选取k个点,来作为kmeans的初始聚类中心点!
不得不说,骚操作!
9 KMeans、层次聚类、DBSCAN三者比较
相同点:
- 都是聚类算法
不同点:
- KMeans需要事先指定聚类个数,而层次聚类,DBSCAN不需要。然而,DBSCAN必须指定另外两个参数:Eps(邻域半径)和MinPts(最少点数)。最后层次聚类也需要结合谱系聚类图看需要聚成几类!
- K均值和层次聚类一般聚类所有对象,而DBSCAN丢弃被它识别为噪声的对象,在sklearn中最后结果会有一个“-1”类,被识别为异常值!
- 思想不同。层次聚类是基于样本点两两之间的距离,K均值使用簇的基于原型的概念,而DBSCAN使用基于密度的概念
- K均值算法的时间复杂度是O(m),而DBSCAN的时间复杂度是O(m^2)。这是不加优化的时候。
【不进行任何优化时,算法的时间复杂度是O( ),通常可利用R-tree,k-d tree, ball tree索引来加速计算,将算法的时间复杂度降为O(Nlog(N)) 】因为考虑这个点的邻域的时候,需要计算这个点到每个点的距离!如果不优化则为平方复杂度!如果有条件限制搜索,则是Nlog(N)。
- 层次聚类,DBSCAN多次运行产生相同的结果,而K均值通常使用随机初始化质心,不会产生相同的结果。
参考
- 很多细节汇总:https://www.cnblogs.com/dudumiaomiao/p/5839905.html
- 刘建平 kmeans-sklearn https://www.cnblogs.com/pinard/p/6169370.html
- 刘建平 kmeans https://www.cnblogs.com/pinard/p/6164214.html
- 凸集:http://sofasofa.io/forum_main_post.php?postid=1000329
- 有趣的DBSCAN:https://jonuknownothingsnow.github.io/2018/10/14/谐门算法:DBSCAN原理/